Strumenti per la valutazione in tempo reale di un modello di infezione da Pseudomonas aeruginosa
Summary
Il mezzo espettorato della fibrosi cistica sintetica (SCFM2) può essere utilizzato in combinazione sia con la microscopia a scansione laser confocale che con lo smistamento cellulare attivato dalla fluorescenza per osservare aggregati batterici ad alta risoluzione. Questo documento descrive in dettaglio i metodi per valutare le popolazioni aggregate durante il trattamento antimicrobico come piattaforma per studi futuri.
Abstract
Pseudomonas aeruginosa (Pa) è uno dei più comuni patogeni opportunistici associati alla fibrosi cistica (FC). Una volta stabilita la colonizzazione della Pa, gran parte dei batteri infettivi forma biofilm all’interno dell’espettorato delle vie aeree. I biofilm di Pa isolati dall’espettorato CF hanno dimostrato di crescere in piccoli aggregati densi di ~ 10-1.000 cellule che sono organizzate spazialmente e mostrano fenotipi clinicamente rilevanti come la tolleranza antimicrobica. Una delle maggiori sfide per studiare come gli aggregati di Pa rispondono al mutevole ambiente dell’espettorato è la mancanza di sistemi nutrizionalmente rilevanti e robusti che promuovano la formazione di aggregati. Utilizzando un mezzo sintetico per l’espettorato CF (SCFM2), la storia di vita degli aggregati di Pa può essere osservata utilizzando la microscopia a scansione laser confocale (CLSM) e l’analisi delle immagini alla risoluzione di una singola cellula. Questo sistema in vitro permette l’osservazione di migliaia di aggregati di varie dimensioni in tempo reale, tridimensionali, e su scala micron. A livello individuale e di popolazione, avere la capacità di raggruppare gli aggregati per fenotipo e posizione facilita l’osservazione degli aggregati in diversi stadi di sviluppo e la loro risposta ai cambiamenti nel microambiente, come il trattamento antibiotico, da differenziare con precisione.
Introduction
Pseudomonas aeruginosa (Pa) è un patogeno opportunistico che stabilisce infezioni croniche in individui immuno-compromessi. Per quelli con la malattia genetica fibrosi cistica (FC), queste infezioni possono durare nel corso della vita. La FC provoca l’accumulo di un espettorato viscoso e ricco di sostanze nutritive nelle vie aeree, che viene colonizzato da una varietà di agenti patogeni microbici nel tempo. Pa è uno dei patogeni CF più diffusi, colonizzando le vie aeree nella prima infanzia e stabilendo infezioni difficili da trattare1. La Pa rimane un problema clinico significativo ed è considerata una delle principali cause di mortalità in quelli con FC, nonostante i regimi terapeutici migliorati negli ultimi anni2,3. Questo fenotipo di persistenza e la crescente tolleranza agli antibiotici hanno fatto guadagnare a Pa un posto in un gruppo di patogeni identificati sia dai Centers for Disease Control (CDC) che dall’Organizzazione Mondiale della Sanità (OMS) come priorità di ricerca per lo sviluppo di nuove strategie terapeutiche: i patogeni ESKAPE4.
Come altri patogeni ESKAPE, la resistenza agli antibiotici acquisita è comune in Pa, ma ci sono anche molte proprietà intrinseche che contribuiscono alla tolleranza antimicrobica Pa. Tra questi c’è la capacità di Pa di formare aggregati-cluster altamente densi di ~ 10-1.000 cellule, che possono essere osservati in infezioni multiple, tra cui l’espettorato del paziente CF5,6. Simile alla Pa studiata in altri sistemi di biofilm, gli aggregati di Pa mostrano fenotipi clinicamente rilevanti come l’aumento della resistenza agli antibiotici e l’attivazione della comunicazione cellula-cellula (quorum sensing (QS)). Ad esempio, gli aggregati di Pa hanno dimostrato di utilizzare comportamenti regolati da QS per combattere altri microbi e tollerare trattamenti antimicrobici come la produzione di piocianina7. La capacità di studiare tali comportamenti offre una visione emozionante degli ecosistemi batterici in un ambiente simile a quello in cui esistono nel corpo umano.
Una delle maggiori sfide per studiare come gli aggregati di Pa rispondono al mutevole ambiente dell’espettorato è la mancanza di sistemi nutrizionalmente rilevanti e robusti che promuovano la formazione di aggregati. Gran parte di ciò che si sa sulla Pa è stato scoperto utilizzando sistemi in vitro in cui le cellule crescono planctonicamente o in una caratteristica architettura “a fungo” attaccata alla superficie che non è stata osservata in vivo8. Mentre i modelli classici di crescita del biofilm, come le cellule di flusso o l’agar solido, hanno prodotto una conoscenza ampia e preziosa sui comportamenti batterici e sui meccanismi di tolleranza agli antibiotici, questi risultati non sempre si traducono in vivo. Molti modelli in vitro hanno una capacità limitata di imitare l’ambiente di crescita del sito di infezione umana, rendendo necessari costosi studi in vivo. A loro volta, molti modelli in vivo mancano della flessibilità e della risoluzione offerte dalle tecniche in vitro.
L’espettorato sintetico della fibrosi cistica (SCFM2) è progettato per fornire un ambiente per la crescita di Pa simile a quello sperimentato durante l’infezione cronica nel polmone CF. SCFM2 include fonti nutrizionali identificate nella CF sputa espettorata oltre a mucina, lipidi e DNA. La crescita di Pa in SCFM2 richiede un set genetico quasi identico a quello richiesto per la crescita nell’espettorato reale e supporta la formazione naturale di aggregati di Pa 9,10. Dopo l’inoculazione, le cellule planctoniche formano aggregati che aumentano di dimensioni attraverso l’espansione. Le singole cellule (denominate migranti) vengono rilasciate dagli aggregati, migrano verso aree non colonizzate e formano nuovi aggregati10. Questa storia di vita può essere osservata utilizzando CLSM e l’analisi delle immagini alla risoluzione di una singola cellula. Gli aggregati di Pa formati in SCFM2 sono di dimensioni simili a quelli osservati nel polmone CF10. Questo modello consente l’osservazione di aggregati multipli di varie dimensioni in tempo reale e in tre dimensioni su scala micron. La microscopia time-lapse consente il tracciamento di migliaia (~ 50.000) di aggregati in un unico esperimento. L’uso di software di analisi delle immagini consente la quantificazione di fenotipi aggregati da micrografie, tra cui volume aggregato, superficie e posizione in tre dimensioni fino all’aeroporto di 0,1 μm, sia a livello di aggregato individuale che di popolazione. Avere la capacità di raggruppare gli aggregati per fenotipo e posizione consente la differenziazione degli aggregati in diversi stadi di sviluppo con precisione, nonché la loro risposta a un microambiente mutevole6,11.
L’applicazione di SCFM2 per studiare aggregati di Pa in saggi a basso volume e ad alto rendimento lo rendono un modello flessibile ed economico. Come mezzo definito, SCFM2 offre uniformità e riproducibilità su più piattaforme, fornendo un metodo nutrizionalmente e fisicamente rilevante per studiare gli aggregati di Pa in vitro9. Le applicazioni includono il suo uso in combinazione con CLSM per osservare l’organizzazione spaziale e la tolleranza agli antibiotici ad alta risoluzione (come descritto in questo documento sui metodi). La capacità di eseguire esperimenti che forniscono dati in tempo reale su scala micron consente lo studio delle interazioni intra-specie e inter-specie come possono verificarsi in vivo. Ad esempio, SCFM2 è stato precedentemente utilizzato per studiare le dinamiche spaziali della comunicazione cellula-cellula in popolazioni aggregate attraverso una rete di sistemi utilizzati da Pa per regolare più geni che contribuiscono alla virulenza e alla patogenesi6.
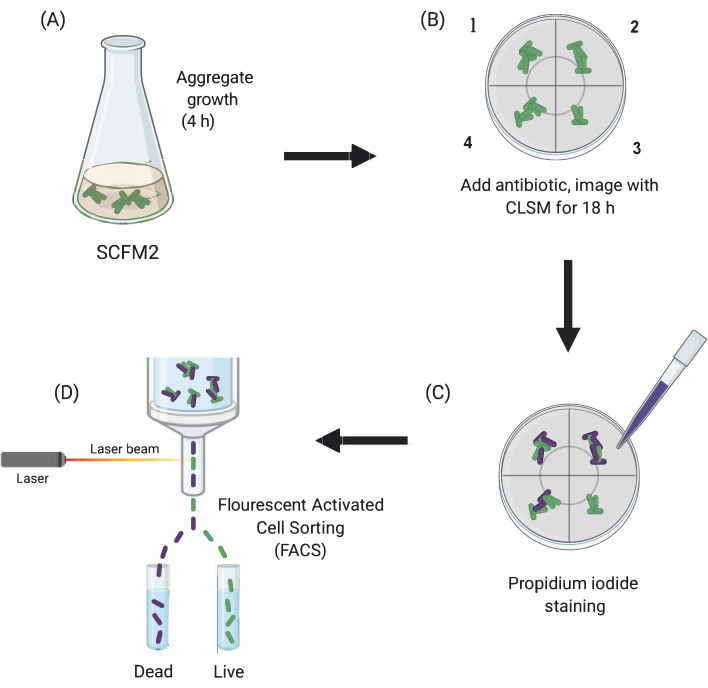
Figura 1: Rappresentazione grafica delle principali fasi sperimentali. (A) SCFM2 viene inoculato con cellule Pa e lasciato formare aggregati in un piatto di coltura con fondo di vetro. (B) Gli aggregati vengono trasferiti al microscopio confocale e viene aggiunto l’antibiotico. Sono raffigurate tre repliche tecniche (camere 1-3) e un pozzo di controllo (4) di SCFM2 inoculato senza trattamento antibiotico. Gli aggregati vengono ripresi utilizzando CLSM nel corso di 18 ore. (C) Dopo l’imaging iniziale di 18 ore, gli aggregati vengono trattati con ioduro di propidio per visualizzare le cellule morte e ripresi utilizzando CLSM (D) Gli aggregati con fenotipo desiderato vengono separati da SCFM2 utilizzando FACS. Abbreviazioni: SCFM2 = mezzo espettorato sintetico della fibrosi cistica; Pa = Pseudomonas aeruginosa; CLSM = microscopia a scansione laser confocale; FACS = selezione cellulare attivata dalla fluorescenza. Fare clic qui per visualizzare una versione più grande di questa figura.
Qui, viene dimostrata l’utilità di SCFM2 per studiare l’impatto del trattamento antibiotico sugli aggregati di Pa in tempo reale, seguita dall’uso di un approccio di ordinamento cellulare per isolare popolazioni di aggregati con fenotipi distinti per l’analisi a valle (Figura 1).
Protocol
Representative Results
Discussion
Questo lavoro ha introdotto metodologie che possono essere combinate per studiare le popolazioni di aggregati batterici in presenza e assenza di trattamento antibiotico. ClSM ad alta risoluzione consente la visualizzazione dei cambiamenti nella biomassa aggregata e l’orientamento strutturale degli aggregati in tempo reale quando esposti agli antibiotici. Inoltre, le caratteristiche fisiche e strutturali della biomassa che rimangono dopo il trattamento con antibiotici possono essere quantificate, con l’obiettivo di correl…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
S.E.D è supportato da fondi di start-up forniti dal Dipartimento di Medicina Molecolare, The University of South Florida, nonché da una borsa di ricerca CFF (DARCH19G0), dal N.I.H (5R21AI147654 – 02 (PI, Chen)) e dall’USF Institute on Microbiomes. Ringraziamo il laboratorio whiteley per la collaborazione in corso che coinvolge set di dati relativi a questo manoscritto. Ringraziamo il Dr. Charles Szekeres per aver facilitato lo smistamento FACS. Le figure sono state create da A.D.G e S.E.D utilizzando Biorender.com.
Materials
| Amino acids | |||
| Alanine | Acr s Organics s Organics |
56-41-7 | |
| Arginine HCl | MP | 1119-34-2 | |
| Asparagine | Acr s Organics s Organics |
56-84-8 | Prepared in 0.5 M NaOH |
| Cystine HCl | Alfa Aesar | L06328 | |
| Glutamic acid HCl | Acr s Organics s Organics |
138-15-8 | |
| Glycine | Acr s Organics s Organics |
56-40-6 | |
| Histidine HCl H2O | Alfa Aesar | A17627 | |
| Isoleucine | Acr s Organics s Organics |
73-32-5 | |
| Leucine | Alfa Aesar | A12311 | |
| Lysine HCl | Alfa Aesar | J62099 | |
| Methionine | Acr s Organics s Organics |
63-68-3 | |
| Ornithine HCl | Alfa Aesar | A12111 | |
| Phenylalanine | Acr s Organics s Organics |
63-91-2 | |
| Proline | Alfa Aesar | A10199 | |
| Serine | Alfa Aesar | A11179 | |
| Threonine | Acr s Organics s Organics |
72-19-5 | |
| Tryptophan | Acr s Organics s Organics |
73-22-3 | Prepared in 0.2 M NaOH |
| Tyrosine | Alfa Aesar | A11141 | Prepared in 1.0 M NaOH |
| Valine | Acr s Organics s Organics |
72-18-4 | |
| Antibiotic | |||
| Carbenicillin | Alfa Aesar | J6194903 | |
| Day-of Stocks | |||
| CaCl2 * 2H2O | Fisher Chemical | C79-500 | |
| Dextrose (D-glucose) | Fisher Chemical | 50-99-7 | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Fisher (Avanti Polar Lipids) | 4235-95-4 | shake 15-20 min at 37 °C to evaporate chloroform |
| FeSO4 * 7H2O | Acr s Organics s Organics |
7782-63-0 | this stock equals 1 mg/mL, MUST make fresh |
| L-lactic acid | Alfa Aesar | L13242 | pH stock to 7 with NaOH |
| MgCl2 * 6H2O | Acr s Organics s Organics |
7791-18-6 | |
| N-acetylglucosamine | TCI | A0092 | |
| Prepared solids | |||
| Porcine mucin | Sigma | M1778-100G | UV-sterilize |
| Salmon sperm DNA | Invitrogen | 15632-011 | |
| Stain | |||
| Propidium iodide | Alfa Aesar | J66764MC | |
| Salts | |||
| K2SO4 | Alfa Aesar | A13975 | |
| KCl | Alfa Aesar | J64189 | add solid directly to buffered base |
| KNO3 | Acr s Organics s Organics |
7757-79-1 | |
| MOPS | Alfa Aesar | A12914 | add solid directly to buffered base |
| NaCl | Fisher Chemical | S271-500 | add solid directly to buffered base |
| Na2HPO4 | RPI | S23100-500.0 | |
| NaH2PO4 | RPI | S23120-500.0 | |
| NH4Cl | Acr s Organics s Organics |
12125-02-9 | add solid directly to buffered base |
| Consumables | |||
| Conical tubes (15 mL) | Olympus plastics | 28-101 | |
| Conical tubes (50 mL) | Olympus plastics | 28-106 | |
| Culture tubes w/air flow cap | Olympus plastics | 21-129 | |
| 35 mm four chamber glass-bottom dish | CellVis | NC0600518 | |
| Luria Bertani (LB) broth | Genessee Scientific | 11-118 | |
| Phosphate-buffered saline (PBS) | Fisher Bioreagents | BP2944100 | |
| Pipet tips (p200) | Olympus plastics | 23-150RL | |
| Pipet tips (p1000) | Olympus plastics | 23-165RL | |
| Serological pipets (5 mL) | Olympus plastics | 12-102 | |
| Serological pipets (25 mL) | Olympus plastics | 12-106 | |
| Serological pipets (50 mL) | Olympus plastics | 12-107 | |
| Ultrapure water (RNAse/DNAse free); nanopure water | Genessee Scientific | 18-194 | Nanopure water used for preparation of solutions in Table 1 |
| Syringes (10 mL) | BD | 794412 | |
| Syringes (50 mL) | BD | 309653 | |
| 0.22 mm PES syringe filter | Olympus plastics | 25-244 | |
| PS cuvette semi-mico | Olympus plastics | 91-408 | |
| Software | |||
| Biorender | To prepare the figures | ||
| FacsDiva6.1.3 | Becton Dickinson, San Jose, CA | ||
| Imaris | Bitplane | version 9.6 | |
| Zen Black | |||
| Equipment | |||
| FacsAriallu | Becton Dickinson, San Jose, CA | ||
| LSM 880 confocal laser scanning microscope | Zeiss |
Referenzen
- Ramsay, K. A., et al. The changing prevalence of pulmonary infection in with fibrosis: A longitudinal analysis. Journal of Cystic Fibrosis. 16 (1), 70-77 (2017).
- Bessonova, L., et al. Data from the US and UK cystic fibrosis registries support disease modification by CFTR modulation with ivacaftor. Thorax. 73 (8), 731-740 (2018).
- Breuer, O., et al. Changing prevalence of lower airway infections in young children with cystic fibrosis. American Journal of Respiratory and Critical Care Medicine. 200 (5), 590-599 (2019).
- O’Donnell, J. N., Bidell, M. R., Lodise, T. P. Approach to the treatment of patients with serious multidrug-resistant Pseudomonas aeruginosa infections. Pharmacotherapy. 40 (9), 952-969 (2020).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Darch, S. E., et al. Spatial determinants of quorum signaling in a Pseudomonas aeruginosa infection model. Proceedings of the National Academy of Sciences of the United States of America. 115 (18), 4779-4784 (2018).
- Zhu, K., Chen, S., Sysoeva, T. A., You, L. Universal antibiotic tolerance arising from antibiotic-triggered accumulation of pyocyanin in Pseudomonas aeruginosa. PLoS Biology. 17 (12), 3000573 (2019).
- Ciofu, O., Tolker-Nielsen, T. Tolerance and resistance of Pseudomonas aeruginosa biofilms to antimicrobial agents-how P. aeruginosa can escape antibiotics. Frontiers in Microbiology. 10, 913 (2019).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proceedings of the National Academy of Sciences of the United States of America. 112 (13), 4110-4115 (2015).
- Darch, S. E., et al. Phage inhibit pathogen dissemination by targeting bacterial migrants in a chronic infection model. MBio. 8 (2), 00240 (2017).
- Jorth, P., et al. Regional isolation drives bacterial diversification within cystic fibrosis lungs. Cell Host & Microbe. 18 (3), 307-319 (2015).
- Palmer, K. L., Aye, L. M., Whiteley, M. Nutritional cues control Pseudomonas aeruginosa multicellular behavior in cystic fibrosis sputum. Journal of Bacteriology. 189 (22), 8079-8087 (2007).
- Davies, D. G., et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm. Science. 280 (5361), 295-298 (1998).
- Hartmann, R., et al. Quantitative image analysis of microbial communities with BiofilmQ. Nature Microbiology. 6 (2), 151-156 (2021).
- Stacy, A., et al. Bacterial fight-and-flight responses enhance virulence in a polymicrobial infection. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7819-7824 (2014).

