La evaluación simultánea de los cardiomiocitos de síntesis de ADN y la ploidía: Un Método de Ayuda a la cuantificación de los cardiomiocitos Regeneración y Facturación
Summary
Quantification of cardiomyocyte turnover is challenging. The protocol described here makes an important contribution to this challenge by enabling accurate and sensitive quantification of neo-cardiomyocyte nuclei generation and nuclei ploidy.
Abstract
Aunque se acepta que el corazón tiene un potencial limitado para regenerar los cardiomiocitos después de la lesión y que los bajos niveles de rotación de los cardiomiocitos se producen durante el envejecimiento normal, la cuantificación de estos eventos sigue siendo un reto. Esto es en parte debido a la rareza del proceso y el hecho de que múltiples fuentes celulares contribuyen al mantenimiento del miocardio. Además, la duplicación del ADN dentro de los cardiomiocitos a menudo conduce a un cardiomiocito poliploides y sólo rara vez conduce a nuevos cardiomiocitos por división celular. Con el fin de cuantificar con precisión el volumen de negocios de los cardiomiocitos discriminación entre estos procesos es esencial. El protocolo descrito aquí emplea etiquetado nucleósido a largo plazo con el fin de etiquetar todos los núcleos que han surgido como resultado de la replicación del ADN y los núcleos de cardiomiocitos identificados mediante la utilización de aislamiento de núcleos y posterior immunolabeling PCM1. En conjunto, esto permite la identificación precisa y sensible del etiquetado nucleósido de la cardiomyocyte población núcleos. Además, 4 ', el etiquetado y el análisis de la ploidía núcleos 6-diamidino-2-fenilindol, permite la discriminación de los núcleos neo-cardiomiocitos a partir de núcleos que han incorporado nucleósido durante polyploidization. Aunque este método no puede controlar para binucleación cardiomiocitos, permite una cuantificación rápida y robusta de núcleos neo-cardiomiocitos mientras que representa el polyploidization. Este método tiene una serie de aplicaciones posteriores, incluyendo la evaluación de los agentes terapéuticos potenciales para mejorar la regeneración de los cardiomiocitos o la investigación de los efectos de la enfermedad cardiaca en el recambio de los cardiomiocitos y la ploidía. Esta técnica también es compatible con las técnicas inmunohistoquímico aguas abajo adicionales, lo que permite la cuantificación de la incorporación de nucleósido en todos los tipos de células cardíacas.
Introduction
En los últimos años ha habido una acumulación de pruebas que cuestionan la suposición de que el corazón es un post-mitótico 1,2 de órganos terminales diferenciadas. Sin embargo, la cuantificación del volumen de negocios de los cardiomiocitos y la regeneración sigue siendo un reto.
Las dificultades para identificar con precisión la generación de cardiomiocitos raras mediante técnicas de inmunohistoquímica estándar están bien reportaron 3. Además, la fuente celular de la generación de cardiomiocitos sigue siendo incierto con la evidencia para las contribuciones de la proliferación de los cardiomiocitos, así como por la diferenciación de células madre 4-6. Por lo tanto, el uso de modelos de linaje rastreo que requieren el conocimiento del fenotipo de cardiomiocitos progenitor es imposible y la cuantificación de la proliferación en una única población, incluyendo los cardiomiocitos, es inapropiado. Además un cardiomiocito tiene el potencial para endoreplication sin cariocinesis (que resulta en un coche poliploidediomyocyte) o cariocinesis en ausencia de la citocinesis (que resulta en un cardiomiocito binucleadas) 7,8. La cuantificación precisa de la rotación de los cardiomiocitos depende de la capacidad de distinguir entre estos eventos y la verdadera generación de neo-cardiomiocitos. Esto crea complicaciones únicas porque la replicación del ADN y la expresión de las quinasas dependientes de ciclina en cardiomiocitos no demuestran exclusivamente división celular verdadero 9,10.
Para ayudar en la cuantificación de la generación de neo-cardiomiocitos, hemos combinado una técnica de aislamiento núcleos establecido, y el etiquetado inmunológica de material pericentriolar 1 (PCM1) para la identificación de los núcleos de cardiomiocitos como se describe por Bergmann et al. 7,11 con nuevos métodos de largo plazo marcaje de ADN y análisis de ploidía. PCM-1 es una proteína de centrosoma que se acumula en la superficie nuclear de diferenciados miocitos, no de ciclismo. Estudios previos han demostrado que los anticuerpos contraPCM-1 etiquetar específicamente núcleos de cardiomiocitos 7,11 y, como tal PCM1 ha sido utilizado por un número de grupos independientes para identificar los cardiomiocitos 1,12,13. Además, hemos demostrado que la expresión PCM1 asigna a núcleos de cardiomiocitos genéticamente etiquetados en el modelo CRE-TnT ratón transgénico 14 (Figura 1).
El protocolo descrito aquí permite la identificación precisa y sensible de la generación de cardiomiocitos núcleos neo en el corazón de ratón, independientemente de los orígenes celulares (Figura 1A y B) mientras que excluye simultáneamente etiquetado nucleósido debido a polyploidization del análisis (Figura 1C y D). Aunque este método no puede controlar para binucleación cardiomiocitos, permite una cuantificación rápida y robusta de núcleos neo-cardiomiocitos que se requiere para la cuantificación precisa de la rotación de los cardiomiocitos. Además,proporciona una herramienta de detección rápida para evaluar los posibles cambios en la dinámica de generación de cardiomiocitos.
Si bien el etiquetado de ADN implica generalmente 5-bromo-2'-desoxiuridina (BrdU) como el análogo de la timidina, el protocolo descrito aquí utiliza un ensayo de 5-etinil-2 'desoxiuridina (EDU), basado ya que requiere menos etapas de procesamiento para una más rápida a través de entradas y no requiere desnaturalización del ADN para la inmuno-detección, por lo que es compatible con otros protocolos de inmunotinción y aumentando así las posibles aplicaciones posteriores del método.
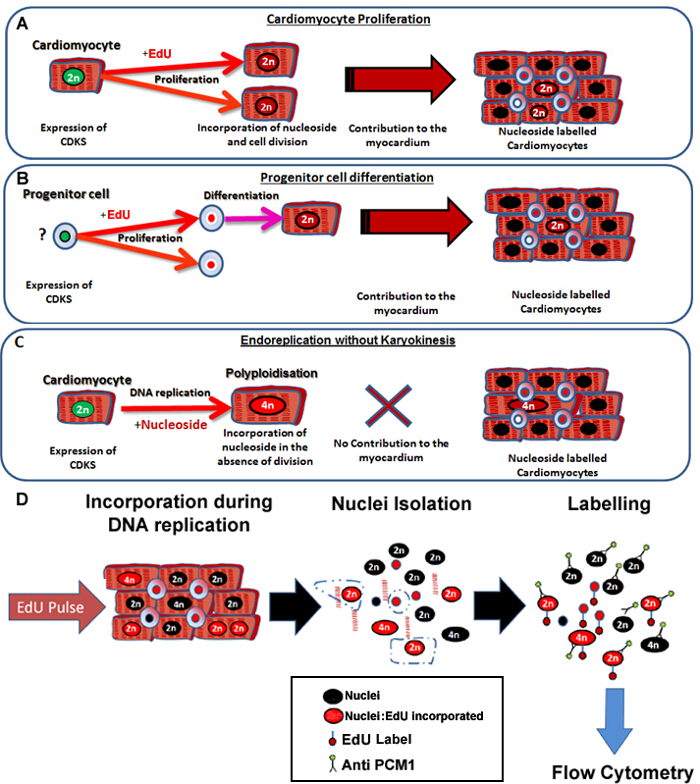
Figura 1: pulsante continuo con etiquetas EdU neo-cardiomiocitos, independientemente de sus progenitores. (A) EdU se incorpora en el ADN de los cardiomiocitos durante la división celular. La proliferación de los cardiomiocitos en la población será Result en un aumento de, o la sustitución de los cardiomiocitos y es por lo tanto la síntesis de ADN productivo (contribuye al mantenimiento y reparación de tejidos). (B) EdU se incorpora en el ADN de las células progenitoras cardiacas durante la división celular. Este será retenido en la célula durante la diferenciación al linaje de los cardiomiocitos. Esta diferenciación de células madre también dará lugar a un aumento en el número de cardiomiocitos y por lo tanto contribuye al mantenimiento y reparación de tejidos. (C) Los cardiomiocitos tienen la posibilidad de someterse a la replicación del ADN "no productivo" que resulta en aumento de ploidía de los cardiomiocitos, que está asociada con la hipertrofia de los cardiomiocitos y la remodelación del miocardio, pero no sustituye a los cardiomiocitos perdidos. El proceso de polyploidization difiere de binucleación ya que resulta en una cardiomiocitos con un solo núcleo, que contiene cuatro o más conjuntos de dos cromosomas homólogos (> 2N). (D) A raíz de una núcleos continua pUlse, este protocolo describe el aislamiento y la identificación de los núcleos de los núcleos de cardiomiocitos por expresión PCM1 para permitir la cuantificación de ambos ploidía de los cardiomiocitos y la incorporación EdU. PCM1 expresión y Edu incorporación detectado mediante citometría de flujo. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Para cuantificar con precisión el volumen de negocios de los cardiomiocitos y ensayos de regeneración deben distinguir entre la verdadera generación de cardiomiocitos y la división de ADN no productiva. Muchos estudios siguen ignorando estos hechos simplemente no productivas, la cuantificación de la proliferación de cardiomiocitos únicamente a través de la expresión de ciclina kinesis y marcadores del ciclo celular. Hasta la fecha, un único método que permite la cuantificación precisa de la rotación de los cardiomiocitos mientras que el control de estos eventos no productivos sigue siendo alusiva. En particular, sigue siendo difícil dar cuenta de los cardiomiocitos polyploidization que contribuye hasta ~ 65% de la replicación del ADN de los cardiomiocitos 13. Por lo tanto para ayudar en la cuantificación exacta de la generación de cardiomiocitos hemos desarrollado un protocolo que permite la cuantificación robusta de los tipos de núcleos de cardiomiocitos neo excluyendo la replicación del ADN que resulta en un incremento de ploidía. Aunque este protocolo puede no discriminación entre los géneros neo-cardiomiocitosción y cardiomiocitos bi-nucleación, que pueden ser utilizados rápidamente y calcular con precisión el límite superior (que representan el ploidía) de generación de cardiomiocitos. Por lo tanto, este protocolo proporciona una herramienta de evaluación para evaluar los posibles cambios en las tasas de generación de cardiomiocitos y polyploidization en modelos de enfermedad o para evaluar la eficacia de la terapéutica potencial. Una vez que los cambios en la velocidad de generación de núcleos neo-cardiomiocitos se identifican utilizando este protocolo estudios posteriores se pueden utilizar para determinar si esto debido a cambios en la generación de cardiomiocitos de número de cardiomiocitos de nucleación, como se describió anteriormente 2,13,17,18. Estos incluyen el uso de la dinámica de nucleación cuantificación cardiomiocitos histológicos durante el período de pulso o análisis de secciones de tejido obtenidos a partir de animales EdU pulsados en comparar EdU incorporación en las poblaciones de cardiomiocitos mononucleadas y multinucleadas.
Debido a los bajos niveles de los cardiomiocitos rotación de esteprotocolo utiliza múltiples inyecciones de EdU lo largo de un período de 7 días. Esto también permite que la "persecución" de todas las posibles fuentes celulares de la generación de cardiomiocitos y permite la cuantificación de la generación de cardiomiocitos núcleos acumulada durante este período de tiempo. Dependiendo del estudio, este periodo de tiempo se puede ajustar para adaptarse a los niveles predichos de generación de cardiomiocitos. Para la cuantificación exacta de EdU incorporación en los núcleos de cardiomiocitos, es imperativo que no hay etiquetado no específica de los núcleos con el anticuerpo secundario usado para detectar PCM-1 reactividad. Por tanto, sería prudente para llevar a cabo experimentos de titulación de anticuerpos secundarios adicionales con el fin de optimizar este aspecto del protocolo, en particular si un anticuerpo secundario que no sea que se sugiere en este protocolo se va a usar. El protocolo descrito aquí utiliza PCM-1 de expresión para identificar los núcleos de cardiomiocitos. Si bien este es un marcador de cardiomiocitos establecido, marcadores alternativos se pueden utilizar para validardatos; estos incluyen anticuerpos específicos para la troponina T cardiaca que se ha identificado como parte localizada en los núcleos de cardiomiocitos 1. Del mismo modo, las proteínas localizadas nucleares alternativos pueden ser utilizados para identificar y cuantificar EdU incorporación en las poblaciones de núcleos distintos de los de los cardiomiocitos. Es importante que todos los núcleos de cardiomiocitos que están experimentando activamente mitosis se excluyen del análisis, ya que se desconoce el destino de esta síntesis de ADN y puede resultar en la división celular o el aumento de ploidía. PCM1 se desmonta durante la fase M del ciclo celular, por lo tanto cardiomiocitos mitosis no será identificado por la expresión PCM1. Además, todos los núcleos en la fase s del ciclo celular deben ser excluidos de análisis posteriores. Esto se puede lograr por Supresión todos los núcleos con una intensidad DAPI por encima de la población 2N incluyendo aquellos con una intensidad DAPI entre el 2N y 4N poblaciones.
Aunque es cada vezaceptado que el corazón tiene la capacidad de sustituir los cardiomiocitos durante el envejecimiento normal y después de una lesión aguda, la fuente y el grado de este potencial sigue siendo controvertido. Además, las tasas dispares de rotación de los cardiomiocitos se han reportado 1,7,20-22. Esto puede ser debido en parte a las dificultades en la identificación y la cuantificación de la generación de neo-cardiomiocitos 19 con precisión. Hasta la fecha la mayoría de los estudios se han basado sólo en el uso del análisis histológico y la identificación de los cardiomiocitos a través de la expresión de proteínas citoplasmáticas, incluyendo proteínas del sarcómero, para la cuantificación del volumen de negocio de los cardiomiocitos y la renovación 2,4,23,24. El uso de estos métodos para detectar la expresión de marcadores de proliferación, o como se ha demostrado aquí, la incorporación de análogos de timidina puede resultar fácilmente en el error de identificación de otros tipos de células cardiaca como cardiomiocitos. Mientras que el uso de la imagen confocal 3D puede ayudar a aliviar estos problemas thESE métodos son caros y lentos. Curiosamente, el protocolo descrito aquí demuestra neo-cardiomiocitos generación núcleos se produce a un ritmo de 0,17% por semana. Esto es consistente con otra citometría de flujo que demuestran los estudios basados tasas de rotación semanales de hasta 0,13% 5. Aunque es tentador para extrapolar las tasas de rotación anual sobre la base de estos datos, al igual que en estudios previos 2,5,25,26, esto no es apropiado como las tasas de rotación son dinámicos durante el tiempo de vida de un animal 13.
Este método tiene un número de aplicaciones potenciales, incluyendo la evaluación de los agentes terapéuticos potenciales para mejorar la regeneración de los cardiomiocitos o la investigación de los efectos de la enfermedad cardiaca en el recambio de los cardiomiocitos y las tasas de polyploidization cardiomiocitos.
Divulgaciones
The authors have nothing to disclose.
Acknowledgements
This work was supported by the British Heart Foundation, project grant PG/13/69/30454.
Materials
| 0.32 M sucrose | Sigma | 84100 | |
| 10 mM Tris-HCl (pH = 8) | Sigma | T3253 | |
| 5 mM CaCl2 | Sigma | c5086 | |
| 5 mM magnesium acetate | sigma | M-5661 | |
| 2.0 mM EDTA | Sigma | E5134 | |
| 0.5 mM EGTA | Sigma | 63779 | |
| 1 mM DTT | Sigma | D0632 | |
| 70 mM KCl | Sigma | P9541 | |
| 10 mM MgCl2 | Sigma | M8266 | |
| 1.5 mM spermine | Sigma | 85590 | |
| Isotype rabbit IgG- ChIP Grade | abcam | abc7415 | |
| Rabbit anti-PCM-1 antibody | Sigma | HPA023374 | |
| Alexa Fluor 488 F(ab')2 Fragment of Goat Anti-Rabbit IgG (H+L) Antibody | Life technologies | A-11070 | |
| cell strainers 70 μm and 100μm | Fisher scientific | 11597522, 11517532 | |
| Glass dounce (40 ml) and pestle large clearance | Sigma | D9188-1SET | |
| EdU (5-ethynyl-2’-deoxyuridine) | Life technologies | A10044 | |
| Click-iT Plus EdU Alexa Fluor 647 Flow Cytometry Assay Kit | Life technologies | C10634 | This kit inlcudes reagents required for section, EdU reaction buffer, EdU fixative, saponin-based permeabilization solution and the reagents required for the EdU labelling cocktail. |
| CyStain DNA 2 step kit, | Sysmex Partec | 05 5005 | This kit inlcudes reagents required for DAPI labelling (DNA staining solution) |
| Probe homogeniser e.g. TissueRuptor | Qiagen | 9001273 | |
| TissueRuptor Disposable Probes | Qiagen | 990890 | |
| ultracentrifuge | Sorvall | ||
| Facscanto II | BD Biosciences | ||
| Ultracentrifuge Tube, Thinwall, Polypropylene. 38.5 mL, 25 x 89 mm | Beckman Coulter | 326823 | |
| Bovine serum albumin | Sigma | A2153 |
Referencias
- Bergmann, O., et al. Evidence for cardiomyocyte renewal in humans. Science. 324, 98-102 (2009).
- Senyo, S. E., et al. Mammalian heart renewal by pre-existing cardiomyocytes. Nature. 493, 433-436 (2013).
- Soonpaa, M. H., Rubart, M., Field, L. J. Challenges measuring cardiomyocyte renewal. Biochim Biophys Acta. 1833, 799-803 (2013).
- Loffredo, F. S., Steinhauser, M. L., Gannon, J., Lee, R. T. Bone marrow-derived cell therapy stimulates endogenous cardiomyocyte progenitors and promotes cardiac repair. Cell Stem Cell. 8, 389-398 (2011).
- Malliaras, K., et al. Cardiomyocyte proliferation and progenitor cell recruitment underlie therapeutic regeneration after myocardial infarction in the adult mouse heart. EMBO Mol Med. 5, 191-209 (2013).
- Hsieh, P. C., et al. Evidence from a genetic fate-mapping study that stem cells refresh adult mammalian cardiomyocytes after injury. Nat Med. 13, 970-974 (2007).
- Bergmann, O., et al. Identification of cardiomyocyte nuclei and assessment of ploidy for the analysis of cell turnover. Exp Cell Res. 317, 188-194 (2011).
- Herget, G. W., Neuburger, M., Plagwitz, R., Adler, C. P. DNA content, ploidy level and number of nuclei in the human heart after myocardial infarction. Cardiovasc Res. 36, 45-51 (1997).
- Scholzen, T., Gerdes, J. The Ki-67 protein: from the known and the unknown. J Cell Physiol. 182, 311-322 (2000).
- Carmena, M., Earnshaw, W. C. The cellular geography of aurora kinases. Nat Rev Mol Cell Biol. 4, 842-854 (2003).
- Bergmann, O., Jovinge, S. Isolation of Cardiomyocyte Nuclei from Post-mortem Tissue. J. Vis. Exp. , e4205 (2012).
- Gilsbach, R., et al. Dynamic DNA methylation orchestrates cardiomyocyte development, maturation and disease. Nat Commun. 5, 5288 (2014).
- Richardson, G., Laval, S., Owens, W. A. Cardiomyocyte regeneration in the mdx mouse model of non-ischemic cardiomyopathy. Stem Cells Dev. , (2015).
- Jiao, K., et al. An essential role of Bmp4 in the atrioventricular septation of the mouse heart. Genes Dev. 17, 2362-2367 (2003).
- Bergmann, O., et al. Cardiomyocyte renewal in humans. Circ Res. 110, e17-e18 (2012).
- Prigge, J. R., et al. Nuclear double-fluorescent reporter for in vivo and ex vivo analyses of biological transitions in mouse nuclei. Mamm Genome. , (2013).
- Naqvi, N., et al. A proliferative burst during preadolescence establishes the final cardiomyocyte number. Cell. 157, 795-807 (2014).
- Liu, Z., Yue, S., Chen, X., Kubin, T., Braun, T. Regulation of cardiomyocyte polyploidy and multinucleation by CyclinG1. Circ Res. 106, 1498-1506 (2010).
- Ang, K. L., et al. Limitations of conventional approaches to identify myocyte nuclei in histologic sections of the heart. Am J Physiol Cell Physiol. 298, C1603-C1609 (2010).
- Kajstura, J., et al. Myocyte turnover in the aging human heart. Circ Res. 107, 1374-1386 (2010).
- Kajstura, J., et al. Cardiomyogenesis in the adult human heart. Circ Res. 107, 305-315 (2010).
- Walsh, S., Ponten, A., Fleischmann, B. K., Jovinge, S. Cardiomyocyte cell cycle control and growth estimation in vivo–an analysis based on cardiomyocyte nuclei. Cardiovasc Res. 86, 365-373 (2010).
- Anversa, P., Leri, A., Kajstura, J. Cardiac regeneration. J Am Coll Cardiol. 47, 1769-1776 (2006).
- Gonzalez-Valdes, I., et al. Bmi1 limits dilated cardiomyopathy and heart failure by inhibiting cardiac senescence. Nat Commun. 6, 6473 (2015).
- Kimura, W., et al. Hypoxia fate mapping identifies cycling cardiomyocytes in the adult heart. Nature. 523, 226-230 (2015).
- Soonpaa, M. H., Field, L. J. Assessment of cardiomyocyte DNA synthesis in normal and injured adult mouse hearts. Am J Physiol. 272, H220-H226 (1997).

