Визуализация взаимодействия белков ДНК по иммунофлюоресценции
Summary
После повреждения ДНК, человеческие клетки активируют необходимые пути восстановления для восстановления целостности их генома. Здесь мы описываем метод косвенного иммунофторесценции как средство обнаружения белков восстановления ДНК, анализа их пространственного и временного набора, а также помогаем допросить взаимодействие белково-белкового белка в местах повреждения ДНК.
Abstract
Клетки млекопитающих постоянно подвергаются воздействию химических веществ, излучению и естественным метаболическим побочным продуктам, которые создают специфические типы оскорблений ДНК. Генотоксические агенты могут повредить позвоночник ДНК, сломать его или изменить химический характер отдельных оснований. После оскорбления ДНК активируются пути реакции повреждения ДНК (DDR) и набираются белки, участвующие в ремонте. Множество факторов участвуют в зондировании типа повреждения и активации соответствующего ответа на ремонт. Неспособность правильно активировать и набирать факторы DDR может привести к геномной нестабильности, которая лежит в основе многих человеческих патологий, включая рак. Исследования белков DDR могут дать представление о генотоксической реакции препарата и клеточных механизмах лекарственной устойчивости.
Есть два основных способа визуализации белков in vivo:прямое наблюдение, пометки белка интереса с флуоресцентным белком и после него живой визуализации, или косвенные иммунофторесценции на фиксированных образцах. В то время как визуализация флуоресцентно помеченных белков позволяет точно контролировать с течением времени, прямые пометки в N- или C-термине могут помешать локализации или функции белка. Предпочтительнее наблюдение за белками в их немодифицированном эндогенном варианте. Когда белки восстановления ДНК набираются на оскорбление ДНК, их концентрация увеличивается локально, и они образуют группы, или “фоки”, которые могут быть визуализированы косвенным иммунофторесценцией с использованием конкретных антител.
Хотя обнаружение белковых очагов не является окончательным доказательством прямого взаимодействия, ко-локализация белков в клетках указывает на то, что они перегруппируются к месту повреждения и могут информировать о последовательности событий, необходимых для сложного образования. Тщательный анализ очагов пространственного перекрытия в клетках, выражаюющих дикий тип или мутантные версии белка, может дать драгоценные подсказки о функциональных областях, важных для функции восстановления ДНК. Наконец, ко-локализация белков указывает на возможные прямые взаимодействия, которые могут быть проверены путем ко-иммунопреципиентации в клетках, или прямого выдвижного использования очищенных белков.
Introduction
Клетки человека постоянно подвергаются воздействию различных ДНК-повреждающих веществ различного происхождения. Экзогенные источники в основном состоят из воздействия радиации, химических веществ (включая химиотерапевтические агенты и некоторые антибиотики) и вирусов, в то время как основные эндогенные источники включают ошибки в репликации ДНК и окислительного стресса. Прямые последствия генотоксического воздействия могут варьироваться от модифицированной базы до потенциально смертельного разрыва двойной нити ДНК (DSB), в зависимости от стресса и дозы воздействия. В конечном счете, невосстановимые или неправильно отремонтированные повреждения ДНК могут привести к накоплению мутаций, геномных перестановок, нестабильности генома и в конечном итоге привести кканцерогенеза 1. Клетки млекопитающих развились сложные пути для распознавания конкретных типовповреждения ДНК 2,3 и ремонта их своевременно, синхронизированы с прогрессированием клеточного цикла.
Ионизирующее излучение (ИК) повреждает двойную спираль ДНК и создает двойные разрывы (DSB), одну из самых вредных форм повреждения ДНК. Комплекс MRN (MRE11, RAD50, NBS1) функционирует как датчик конца ДНК и активирует белок киназы атаксия телангиектазия мутировавшая(ATM) 4,5. После первоначальной активации банкомата по ДНК заканчивается, банкомат запускает каскад событий DDR в месте разрыва, инициируя с ключевым событием, фосфорилирование гистона вариант H2AX6. Фосфорилирование H2AX на остатке S139 активирует его в QH2AX, охватывающих регионы до мегабазы вокругпоражения ДНК 6,,7,,8,,9. Это событие повышает доступность ДНК, что приводит к набору и накоплению других белков восстановленияДНК 7. Из-за того, что ЗГ2АКС обильно и специально индуцирован окружающими DSB, его можно легко визуализировать с помощью специфических антител, и он обычно используется в качестве суррогатного маркера для DSBs в области восстановления ДНК. Как только разрыв сигнализируется, клетки активируют пути восстановления ДНК и обработать повреждение ДНК. Белок MDC1 (посредник белка контрольно-пропускного пункта повреждения ДНК 1) напрямую связывает qH2AX10,взаимодействуетс банкоматом 11, а также с NBS112,,13. Это способствует увеличению концентрации комплекса MRN в DSB и инициированию положительного цикла обратной связи банкоматов. После ремонта разрыв быстро удаляется, что позволяет контролировать клиренс DSB. Вслед за микроскопией, снижение йH2AX с течением времени обеспечивает косвенное измерение остаточных разрывов и эффективности восстановления ДНК.
Эукариотические клетки могут ремонтировать DSBs несколькими путями, два основных из которых являются неомологическими конца присоединения (NHEJ) и гомологичной рекомбинации (HR) (Рисунок 1). NHEJ по существу лигаты ДНК двойной пряди заканчивается без использования расширенной гомологии и работает на протяжении всегоклеточного цикла 14,15. HR становится преобладающим во время фаз S и G2, и в противном случае подавляется, так как он требует сестра хроматид в качестве гомологовогошаблона для ремонта 14,16. Выбор пути между NHEJ и HR зависит не только от физической близости сестры хроматид, но и от расширения ДНК конца ресекции17, который подавляет NHEJ.
Гомологиологически-зависимый ремонт DSB инициирует нуклеолитической деградацией 5′ нитей от концов разрыва для генерации 3′ однотяговых ДНК (ssDNA) хвостов, процесс, называемый 5′-3′ resection. Комплекс MRN инициирует ресекцию конца ДНК и дальнейшая ресекция обрабатывается в сочетании с BLM/EXO1 (белок синдрома Блума/экзонуклеаза 1) или BLM/DNA2 (репликация ДНК АТФ-зависимого геликайса/нуклеазы)18,19,20,21,22. Ресекция конца ДНК усиливается CtIP (CtBP-взаимодействующий белок) через его прямое взаимодействие с MRNкомплекс 23 и набор BRCA1 (белок восприимчивости рака молочной железы типа1) 24,25. Репликационный белок A (RPA) быстро связывается с ssDNA подвергается, а затем вытесняется рекомбиназы белка RAD51 сформировать нуклеопротеин нить, которая катализа гомологичный поиск и вторжениепряди 26,27,28.
Инициирование ресекции является важным шагом для выбора пути ремонта. После того, как ресекция началась, концы ДНК становятся бедными субстратами для связывания гетенодимером Ku70/Ku80 (компонент пути NHEJ), а клетки привержены HR17,,29,30. Ku70/Ku80 гетенодимер связывается с концами DSB, вербовка ДНК-PKcs и p53 Связывающий белок 1 (53BP1)29,30. 53BP1 выступает в качестве барьера для resection в G1, тем самым блокируя HR при продвижении NHEJ31,32, но он удаляется в BRCA1-зависимой манере в фазе S, следовательно, позволяя resectionпроизойти 33,34. Таким образом, 53BP1 и BRCA1 играют противоположную роль в ремонте DSB, с 53BP1 является посредником NHEJ в то время как BRCA1 действует позволяет перерывы для ремонта через HR.
В лаборатории образование DSB может быть вызвано ионизирующим излучением (ИК). Хотя этот пример использует высокую дозу 4 Gy, 1 Gy и 2 Gy также создать значительное количество DSBs, подходит для анализа образования очагов обильными белками. Важно отметить, что тип и доза используемого излучения может привести к различным поражениям в ДНК и в клетке: в то время как ИК индуцирует DSBs, он также может вызвать одиночные разрывы нити или изменения базы(см. 35,36 для справки об облучении линейной передачи энергии (LET) и тип повреждения ДНК). Для определения кинетики ионизирующего радиационно-индуцированного образования очагов (IRIF) и их расчистки, которые указывают на ремонт повреждений и разворот активированного DDR8,,9,,37,,38,образование очагов можно контролировать в разные моменты времени после ионизирующего излучения. Сроки активации и очистки всех основных белков поврежденияДНК известно 39, и многие из них расследуются как суррогатные маркеры ключевых событий. Например, pRPA, который обладает высокой сродством к ssDNA используется в качестве суррогата ресекции разрыва, белки MRN (MRE11, RAD50, NBS1) и экзонуклеазы могут быть использованы для оценки эффективности ресекции тоже. В то время как RAD51, BRCA1, BRCA2 (белок восприимчивости к раку молочной железы 2 типа) и PALB2 (партнер и локализатор BRCA2) могут быть проверены для оценки эффективности HR, наличие белков Ku или 53BP1, используются в качестве маркеров NHEJ (Рисунок 1).
По мере того как протеины машин ремонта ДНАА вербуют один другого к пролому и собирают в супер-комплексах, взаимодействия дна-белка и протеина-протеина можно сделать вывод путем следовать за их индивидуальной локализацией над временем и анализировать co-локализацию протеинов, как визуализировано перекрывая сигналамив клетке 40,41,42. В клеточных линиях введение точечных мутаций или удаление в конкретных генах восстановления ДНК либо путем редактирования генома, либо путем переэкспрессии плазмидных мутантов, позволяет изутовить специфические остатки и их возможную роль в признании повреждения ДНК (например, совместной локализации с помощью QH2AX) или сложной сборки (совместной локализации с другим, или несколькими белками), а также их влияния на ДНК. Здесь мы используем косвенное иммунофторесценцию как среднее для исследования образования и разрешения DSBs, следуя фоклюсу NO2AX с течением времени. Мы также представляем один пример анализа формирования очагов и совместной локализации крупным игроком в ремонте DSB: p53 Binding Protein 1 (53BP1)32. Как упоминалось ранее, 53BP1 считается центральным для выбора пути восстановления ДНК. После накопления 53BP1 и его совместной локализации с хХ2АКС предоставляет ценную информацию о фазе клеточного цикла, накоплении повреждений ДНК и пути, используемом для ремонта DSB. Целью косвенной иммунолокализации является оценка эффективности восстановления повреждений ДНК в клеточных линиях, после ИК, как в данном исследовании, или после воздействия различных напряжений в клетке, от перекрестного пересечения ДНК до блокировки вилки репликации (список повреждающих ДНК агентов приведен в таблице 1).
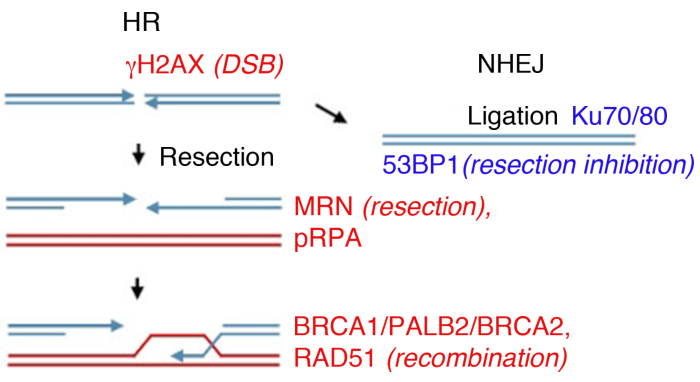
Рисунок 1: ДНК двойные нити перерывов (DSB) ремонт путей.
Ремонт DSB включает в себя два основных пути: гомологичная рекомбинация (HR, слева) и не гомологичный end-Joining (NHEJ, справа). После перерыва, белки активируются, чтобы отметить разрыв (NOH2AX), участвовать в конце ресекции (MRN), пальто resected ssDNA (pRPA), способствовать рекомбинации (BRCA1, PALB2, BRCA2, RAD51) или ограничить ресекцию и содействовать NHEJ (53BP1). Другие белки участвуют в ремонте повреждений, но перечисленные белки обычно следуют косвенные иммунофлуоресценции. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
| Повреждающее ДНК агент | Механизм действия | Рекомендуемая доза |
| К-лучи/рентгеновские лучи | Излучения Формирование двухместных разрывов с некоторыми неконтролируемыми клеточными эффектами |
1-4 Gy |
| 36 Лет Ар ионы | Излучения Формирование двухместных перерывов |
270 кВ/мкм |
| К-частицы | Излучения Формирование двухместных перерывов |
116 кВ/мкм |
| Блеомицин | Ингибитор синтеза ДНК | 0,4-2 мкг/мл |
| Камптотецин | Ингибитор топоизомеразы I | 10-200 нм |
| Цисплатин | Алкилайт агент (индуцирующие внутристрандовые перекрестные ссылки) |
0,25-2 МКМ |
| Доксорубицин | Интеркалирующий агент Ингибитор топоизомеразы II |
10-200 нм |
| Этопозид | Ингибитор топоизомеразы II | 10 МКМ |
| Гидроксиуреа | Ингибитор синтеза ДНК (по рибонуклеотидной редуказе) |
10-200 МКМ |
| Метил метансульфонат | Алкилайт агент | 0,25-2 мММ |
| Митомицин C | Алкилайт агент | 0,25-2 МКМ |
| Ультрафиолетовый (УФ) свет | Формирование димеров тимидин (генерация искажения цепочки ДНК) |
50-100 мДж/см2 |
Таблица 1: Генотоксические агенты. Примеры повреждающих ДНК агентов, их механизм действия и ущерб, индуцированный на основе предложенной рабочей концентрации.
Protocol
Representative Results
Discussion
Анализ сроков и эффективности ремонта повреждений ДНК с помощью микроскопии оказался необходимым для понимания того, как функционирует механизм восстановления ДНК, в нормальных клетках и в патологиях человека, таких как рак.
Разработка специфических антител, позволяющ…
Divulgaciones
The authors have nothing to disclose.
Acknowledgements
Эта работа была поддержана грантом Фонда района Сан-Антонио. Онкологический центр Мэйс поддерживается NCI онкологический центр поддержки основной грант P30 CA054174. Мы хотели бы поблагодарить Стивена Холлоуэя за помощь в поиске реагентов, а также лабораторию Sung за предоставление пространства и микроскопии.
Materials
| 16% (v/v) paraformaldehyde (PFA) aqueous solution | Electron Microscopy Sciences | 15710 | Microscopy quality of the PFA ensures best images. If using "home-made PFA", filter prior to use. |
| Bovine serum albumin (BSA) | Sigma-Aldrich | A3059 | Heat-shock fraction is recommended, to avoid precipitation/background. |
| Coverglass #1, 18 mm round (coverslips) | Neuvitro | NC0308920 | Coverslips need to be cleaned and sterilized prior using, with HCl or ethanol. |
| DMEM, High Glucose [(+) 4.5 g/L D-Glucose, (+) L-Glutamine, (-) Sodium Pyruvate] | Gibco | 11965118 | Adjust the growing media to the needs of cell line used. |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | PBS for tissue culture. |
| Ethylene glycol-bis(β-aminoethyl ether)-N,N,N′,N′-tetraacetic acid (EGTA) | Research Products International | E57060 | Nuclear extraction buffer. |
| Fetal Bovine Serum (FBS) | Life Technologies | 104370028 | The quality of FBS can be assessed by testing gH2AX foci formation. If traces of genotoxic endotoxin are present in the batch, gH2AX will be positive in the absence of stress. |
| Magnesium chloride (MgCl2) | Research Products International | M24000 | Nuclear extraction buffer. |
| Piperazine-N,N′-bis(2-ethanesulfonic acid) (PIPES) | Research Products International | P40150 | Nuclear extraction buffer. |
| SlowFade Diamond Antifade Mountant with DAPI | Invitrogen | S36973 | 300 nM DAPI with VECTASHIELD Antifade Mounting Medium can be used instead. |
| Sodium chloride (NaCl) | Research Products International | S23020 | Nuclear extraction buffer. |
| Sucrose | Research Products International | S24060 | Nuclear extraction buffer. |
| Superfrost Plus Microscope Slides | Fisherbrand | 1255015 | Polysine Slides can be used instead. |
| TC-Treated Multiple Well Plates, size 12 wells | Costar | 3513 | Seeding on coverslips is done in 12-wells plate. |
| Triton X-100 | AmericanBio | AB02025 | Nuclear extraction buffer. |
| TrypLE Express Enzyme (1X), No Phenol Red | Gibco | 12604021 | Trypsin-EDTA can be used instead. |
| Trypsin-EDTA (0.5%), No Phenol Red | Gibco | 15400054 | TrypLE can be used instead. |
Referencias
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor Perspectives in Biology. 7 (4), 016600 (2015).
- Jalan, M., Olsen, K. S., Powell, S. N. Emerging Roles of RAD52 in Genome Maintenance. Cancers (Basel). 11 (7), (2019).
- Oh, J., Symington, L. S. Role of the Mre11 Complex in Preserving Genome Integrity. Genes (Basel). 9 (12), (2018).
- Uziel, T., et al. Requirement of the MRN complex for ATM activation by DNA damage. The EMBO Journal. 22 (20), 5612-5621 (2003).
- Lee, J. H., Paull, T. T. ATM activation by DNA double-strand breaks through the Mre11-Rad50-Nbs1 complex. Science. 308 (5721), 551-554 (2005).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. Journal of Biological Chemistry. 273, 5858-5868 (1998).
- Kinner, A., Wu, W., Staudt, C., Iliakis, G. γ-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Research. 36 (17), 5678-5694 (2008).
- Martin, O. A., Pilch, D. R., Redon, C., Bonner, W. M. Histone H2AX in DNA damage repair. Cancer Biology & Therapy. 2 (3), 233-235 (2003).
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. Journal of Cell Biology. 146 (5), 905-916 (1999).
- Stucki, M., et al. MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks. Cell. 123 (7), 1213-1226 (2005).
- Lou, Z., et al. MDC1 maintains genomic stability by participating in the amplification of ATM-dependent DNA damage signals. Molecular Cell. 21 (2), 187-200 (2006).
- Chapman, J. R., Jackson, S. P. Phospho-dependent interaction between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage. EMBO Reports. 9 (8), 795-801 (2008).
- Melander, F., et al. Phosphorylation of SDT repeats in the MDC1 N terminus triggers retention of NBS1 at the DNA damage-modified chromatin. Journal of Cell Biology. 181 (2), 213-226 (2008).
- Branzei, D., Foiani, M. Regulation of DNA repair throughout the cell cycle. Nature Review. Molecular Cell Biology. 9 (4), 297-308 (2008).
- Chiruvella, K. K., Liang, Z., Wilson, T. E. Repair of double-strand breaks by end joining. Cold Spring Harbor Perspectives in Biology. 5 (5), 012757 (2013).
- Mehta, A., Haber, J. E. Sources of DNA double-strand breaks and models of recombinational DNA repair. Cold Spring Harbor Perspectives in Biology. 6 (9), 016428 (2014).
- Symington, L. S., Gautier, J. Double-strand break end resection and repair pathway choice. Annual Review of Genetics. 45, 247-271 (2011).
- Huertas, P. DNA resection in eukaryotes: deciding how to fix the break. Nature Structural & Molecular Biology. 17 (1), 11-16 (2010).
- Nimonkar, A. V., et al. BLM-DNA2-RPA-MRN and EXO1-BLM-RPA-MRN constitute two DNA end resection machineries for human DNA break repair. Genes & Development. 25 (4), 350-362 (2011).
- Garcia, V., Phelps, S. E. L., Gray, S., Neale, M. J. Bidirectional resection of DNA double-strand breaks by Mre11 and Exo1. Nature. 479 (7372), 241-244 (2011).
- Sturzenegger, A., et al. DNA2 cooperates with the WRN and BLM RecQ helicases to mediate long-range DNA end resection in human cells. Journal of Biological Chemistry. 289 (39), 27314-27326 (2014).
- Daley, J. M., Niu, H., Miller, A. S., Sung, P. Biochemical mechanism of DSB end resection and its regulation. DNA Repair. 32, 66-74 (2015).
- Sartori, A. A., et al. Human CtIP promotes DNA end resection. Nature. 450 (7169), 509-514 (2007).
- Chen, L., Nievera, C. J., Lee, A. Y. L., Wu, X. Cell cycle-dependent complex formation of BRCA1-CtIP-MRN is important for DNA double-strand break repair. Journal of Biological Chemistry. 283, 7713-7720 (2008).
- Yun, M. H., Hiom, K. CtIP-BRCA1 modulates the choice of DNA double-strand break repair pathway throughout the cell cycle. Nature. 459 (7245), 460-463 (2009).
- Sung, P., Klein, H. Mechanism of homologous recombination: mediators and helicases take on regulatory functions. Nature Review. Molecular Cell Biology. 7, 739-750 (2006).
- San Filippo, J., Sung, P., Klein, H. Mechanisms of eukaryotic homologous recombination. Annual Review of Biochemistry. 77, 229-257 (2008).
- Jasin, M., Rothstein, R. Repair of strand breaks by homologous recombination. Cold Spring Harbor Perspectives in Biology. 5 (11), 012740 (2013).
- Dynan, W. S., Yoo, S. Interaction of Ku protein and DNA-dependent protein kinase catalytic subunit with nucleic acids. Nucleic Acids Research. 26 (7), 1551-1559 (1998).
- Lieber, M. R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annual Review of Biochemistry. 79, 181-211 (2010).
- Cejka, P. DNA end resection: nucleases team up with the right partners to initiate homologous recombination. Journal of Biological Chemistry. 290 (38), 22931-22938 (2015).
- Mirman, Z., de Lange, T. 53BP1: a DSB escort. Genes & Development. 34, 7-23 (2020).
- Cao, L., et al. A selective requirement for 53BP1 in the biological response to genomic instability induces by BRCA1 deficiency. Molecular Cell. 35 (4), 534-541 (2009).
- Zimmermann, M., de Lange, T. 53BP1: Pro choice in DNA repair. Trends in Cell Biology. 24 (2), 108-117 (2014).
- Mavragani, I. V., Nikitaki, Z., Kalospyros, S. A., Georgakilas, A. G. Ionizing Radiation and Complex DNA Damage: From Prediction to Detection Challenges and Biological Significance. Cancers (Basel). 11 (11), (2019).
- Nikitaki, Z., et al. Measurement of complex DNA damage induction and repair in human cellular systems after exposure to ionizing radiations of varying linear energy transfer (LET). Free Radical Research. 50, 64-78 (2016).
- Redon, C., et al. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. 12 (2), 162-169 (2002).
- Fernandez-Capetillo, O., Lee, A., Nussenzweig, M., Nussenzweig, A. H2AX: the histone guardian of the genome. DNA Repair. 3 (8-9), 959-967 (2004).
- Paull, T. T., et al. A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage. Current Biology. 10 (15), 886-895 (2000).
- Sy, S. M. H., Huen, M. S. Y., Chen, J. PALB2 is an integral component of the BRCA complex required for homologous recombination repair. Proceedings of the National Academy of Sciences. 106 (17), 7155-7160 (2009).
- Buisson, R., Masson, J. Y. PALB2 self-interaction controls homologous recombination. Nucleic Acids Research. 40 (20), 10312-10323 (2012).
- Belotserkovskaya, R., et al. PALB2 chromatin recruitment restores homologous recombination in BRCA1-deficient cells depleted of 53BP1. Nature Communications. 11 (1), 819 (2020).
- Betts, J. A., et al. Long noncoding RNAs CUPID1 and CUPID2 mediate breast cancer risk at 11q13 by modulating the response to DNA damage. American Journal of Human Genetics. 101 (2), 255-266 (2017).
- Dray, E., et al. Molecular basis for enhancement of the meiotic DMC1 recombinase by RAD51 associated protein 1 (RAD51AP1). Proceedings of the National Academy of Sciences. 108 (9), 3560-3565 (2011).

