通过中子自旋回声光谱研究蛋白质动力学
Summary
本方案描述了研究在人体健康中具有重要作用的两种模型蛋白的结构和动力学的方法。该技术将台式生物物理表征与中子自旋回声光谱相结合,以访问与蛋白质域间运动相关的时间和长度尺度的动力学。
Abstract
大多数人体蛋白质的活性和功能都与蛋白质晶体结构内整个子结构的构型变化有关。晶体结构为描述蛋白质结构或动力学的任何计算奠定了基础,大多数时候具有很强的几何限制。然而,这些来自晶体结构的限制在溶液中并不存在。由于环或子域在微微到纳秒的时间尺度(即内部蛋白质动力学时间状态)上的重排,溶液中蛋白质的结构可能与晶体不同。本著作描述了如何使用中子散射来访问几十纳秒时间尺度上的慢动作。特别是,两种主要的人类蛋白质的动态表征,一种是缺乏明确定义的二级结构的内在无序蛋白质,另一种是经典的抗体蛋白,通过中子自旋回声波谱(NSE)结合各种实验室表征方法来解决。使用数学建模来描述实验中子数据并确定组合扩散和内部蛋白质运动之间的交叉,从而进一步了解蛋白质结构域动力学。提取从NSE获得的对中间散射函数的内部动态贡献,包括各种运动的时间尺度,可以进一步了解单个蛋白质的机械性质以及蛋白质在拥挤蛋白质溶液中几乎自然环境中的柔软度。
Introduction
用中子探测软物质的动力学
研究蛋白质和肽的动力学特性是生物物理研究的主要部分,并且今天存在许多开发良好的方法来访问广泛的能量景观1。将实验揭示的蛋白质动力学与其生物学功能联系起来是一项更加困难的任务,需要复杂的数学模型和计算机辅助动力学模拟。中子光谱学在蛋白质运动分析中的重要性已经在几项广受好评和广泛认可的研究1,2,3,4,5中得到强调。在探索内部蛋白质动力学的多样化能量景观之前,需要简要概述软物质中的动力学过程以及中子如何访问它们。
中子对同位素构型的敏感性以及它们与软物质相互作用的类型使中子散射成为最通用的研究技术之一6.中子可以访问广泛的相关长度尺度和相关时间,从核激发和原子振动到集体运动和缓慢松弛过程,如各向同性旋转和扩散运动。当研究散射中子的能量转移时,可以区分三个主要相互作用:弹性散射,其中入射中子和样品中的粒子之间没有能量交换;非弹性散射,中子和粒子之间具有大的,可量化的能量交换;以及准弹性散射的特殊情况,与入射中子能量1,7相比,它表示非常小的能量转移。这些相互作用提供了有关所研究材料的精确信息,并构成了各种中子散射技术的理论基础。
在弹性散射中,探测器将中子的方向记录为衍射图案,该衍射图谱显示样品原子相对于彼此的位置。获取有关原子位置相关性的信息(即,有关动量转移Q的积分强度 S( Q),仅与结构信息有关)。这个原理构成了中子衍射8的基础。
当由于样品材料中的激发和内部波动而导致能量转移不再为零时,复杂性就会出现。这构成了中子光谱学的基础,其中散射中子被研究为能量转移 E 和动量转移 Q的函数。获得动态和结构信息。中子光谱测量能量转移的相同综合强度 S(Q)(即由于样品散射引起的中子的速度变化, S(Q,ω) = S(Q,E),也称为动态结构因子)9。
为了计算材料的散射,使用对相关函数7,10更合适。在衍射情况下,静态对相关函数 G(r) 给出了在给定距离 r 处找到一个粒子中心与另一个粒子中心的概率。光谱学推广了静态对相关函数,并在散射方程中包括能量/频率/时间。对相关函数 G(r) 成为时间 G(r, t) 的函数,它可以分解为不同的原子对相关函数 GD(r, t) 和自相关函数 GS(r, t)。这些描述了两种类型的相关性:控制相干散射的原子的对相关运动,以及控制非相干散射10的自相关。
相干散射是从“平均值”散射,取决于散射波的相对相位。在小角度散射状态下,来自不同散射中心(不同原子)的散射中子波建设性地干涉(具有相似的相位),并且观察到原子的集体运动具有很强的强度增强。相干散射本质上描述了来自样品10中所有原子核的单个中子的散射。
当来自不同中心的散射中子波之间没有发生相长干涉时,及时跟踪单个原子,并观察到时间 t = 0时原子的位置与时间 t 处的同一原子之间的自相关。因此,关于原子相对位置的信息丢失了,焦点只放在局部波动上。局部波动的散射支配着不相干的散射。非相干散射是各向同性的,有助于背景信号,并降低信噪比10,11。
结合上述所有内容,我们区分了四个主要的中子散射过程10:(1)弹性相干(测量原子位置的相关性),(2)非弹性相干(测量原子的集体运动),(3)弹性不相干(有助于背景,通过Debye-Waller因子(DWF)降低散射强度并测量弹性不相干结构因子(EISF),描述受限几何中扩散运动的几何形状, (4)非弹性非相干(测量单原子动力学和自相关)。
中子在生物学中可以进入的动力学过程范围从低频原子和分子振动的阻尼,溶剂分子与生物表面的相互作用,以及大分子和受限几何形状的水化层中的扩散过程,到短程平移,旋转和翻滚扩散运动,以及蛋白质结构域和变构运动1.用于测量蛋白质动力学的中子方法和仪器的广泛多样性是基于如何实现入射或流出中子束的色变化以及如何进行散射中子的能量分析。从三轴到飞行时间,反向散射和自旋回波光谱仪,人们可以探索特征时间在1 x 10-14 s和1 x 10-6 s(飞秒到微秒)之间的动态过程12。
橡树岭国家实验室拥有两个著名的中子源,散裂中子源 – SNS13 和高同位素通量反应堆 – HFIR14,拥有用于研究生物材料动力学的最佳光谱仪套件之一。一些最雄辩的例子包括在SNS15上使用冷中子斩波光谱仪(CNCS)来研究溶液16 中绿色荧光蛋白周围水合水的动态扰动或几种蛋白质17的亚皮秒集体振动。非弹性中子散射研究的一个反复出现的问题是,一些生物过程太慢而无法观察到。在没有导致中子强度巨大损失的极端设置的情况下,飞行时间光谱仪被限制为10μeV的能量分辨率,相当于最大时间尺度约为200 ps10,11。这不足以观察蛋白质的大规模运动。因此,通常需要具有更高能量分辨率的仪器,如反向散射光谱仪。结合飞行时间和反向散射技术已被证明对于研究细胞色素P450cam(CYP101)的内部动力学变化非常有效,细胞色素P450cam是一种催化羟基化樟脑18的酶。
SNS-BASIS19 的反向散射光谱仪测量的微观扩散率出奇地定义得很好,可以分为水的扩散性(水合作用,细胞质和块状水)和平面扁虫中细胞成分的扩散性,扁平扁虫是第一个通过中子散射20研究的活体动物.反向散射是一种高分辨率光谱技术,但它也仅限于几μeV = 几纳秒,而生物材料中的缓慢动力学也表现为原子位置或自旋取向之间相关性的生存时间(例如,弛豫过程,通常在十到数百纳秒的时间范围内发生)。
中子自旋回波光谱(NSE)是唯一达到如此高分辨率的中子散射技术。与其他中子技术不同,NSE不需要对光束进行消色变,因为它使用中子的量子力学相,即它们的磁矩。磁矩的操纵允许使用宽的中子束波长分布,而该技术对非常小的中子速度变化(1 x 10-4)敏感。NSE已成功用于研究许多蛋白质溶液中蛋白质的缓慢动力学。在这些众多的先驱研究中,我们承认对猪免疫球蛋白21的节段灵活性的研究;Taq聚合酶22中的耦合结构域运动;酵母醇脱氢酶23的四聚体中的结构域运动;底物结合3时磷酸甘油酯激酶构象的变化;Na+/H+交换调节辅因子1(NHERF1)蛋白4,24,25中结构域运动的激活和变构信号的动态传播;汞离子还原酶26的紧致状态的动力学;以及血红蛋白在红细胞中的扩散27。最近两项关于蛋白质动力学的研究揭示了人抗体免疫球蛋白G(IgG)作为熵弹簧28的柔韧性,以及溶剂对内在无序髓鞘碱性蛋白(MBP)动力学的贡献特征5。
本文解释了NSE的基本原理,推荐用于彻底蛋白质动力学研究的多种制备方法,以及SNS,SNS-NSE的NSE光谱仪NSE数据采集的方法和实验方案。该方案表征了两种蛋白质:IgG,一种常规的人抗体蛋白,以及内在无序的蛋白质MBP。简要讨论了生物物理意义,示例的研究相关性以及该技术的局限性。
NSE光谱,慢速动力学测量的方法
NSE是一种极化技术,它使用中子飞行时间来测量由于样品中中子和原子之间的准弹性相互作用而导致的能量交换(极化损失)。NSE光谱学的核心是两个基本原理:(1)中子自旋以与磁强度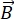 成正比的频率在磁场中进动的能力,即拉莫尔频率29,以及(b)自旋回波或Hann回波,表示在施加一系列射频脉冲30时对偏振信号的操纵和重新对焦。
成正比的频率在磁场中进动的能力,即拉莫尔频率29,以及(b)自旋回波或Hann回波,表示在施加一系列射频脉冲30时对偏振信号的操纵和重新对焦。
NSE 过程的基础知识可以用几个简单的步骤6,11 使用 图 1 进行总结。(1)源(位置1)产生的中子束被偏振(位置2),引导并传输(位置3),并到达NSE光谱仪的入口,在那里它被第一个pi-half鳍(位置4)旋转90°。(2)偏振光束(例如中子磁矩)垂直于第一磁体的磁力线(第一进动区,位置5)并开始进动。(3)在磁体的末端,中子自旋积累一定的进动角,与磁场强度和内部的飞行时间成正比(基本上与中子速度成反比)。单个中子速度在第一个进动区末端的进动角内编码。(4)靠近样品位置,pi-flipper(位置6)将自旋的方向反转180°,改变进动角的符号。(5)中子与样品的分子(位置7)相互作用并散射。(6)散射中子进入第二进动区(位置8)并进动,但变为反向定向。(7) 另一个圆周率半鳍(位置 9)用于将自旋的方向从垂直于水平方向旋转。这将停止进动,将进动角φ转换为与cos(φ)成比例的偏振。(8) 分析仪(位置 10)根据一个方向选择中子。如果与样品的相互作用是弹性的,则中子的速度不会改变。中子将花费相同的时间在第一和第二进动区飞行,并且累积的进动角被完全恢复。探测器(位置11)上的全偏振恢复为原始偏振的回波(即自旋回波)。(9)然而,在NSE中,散射是准弹性的,因此中子和样品分子之间的小能量交换导致样品散射后出现不同的中子速度。由于速度不同,中子将花费额外的时间飞过第二个进动区,并且不会正确恢复其进动角。在探测器上检索到部分偏振,并且由于自旋弛豫引起的偏振损失与光谱函数 S(Q,ω)的cos-傅里叶变换成正比,中间散射函数 F(Q,t)。(10)函数 F(Q,t)的时间参数与进动磁场强度成正比。因此,将极化损失作为磁场强度的函数进行扫描会产生一个松弛函数,该函数取决于样品内的动力学过程。

图 1:SNS (SNS-NSE) 的 NSE 光谱仪照片和包含最重要功能组件的中子飞行路径示意图。 从右到左:1 =中子源;2 = 斩波器-弯曲-偏振片-二次快门系统;3 = 光束传输导轨;4 = pi/2 鳍片,用于第一个 90° 自旋转动;5 = 第一进动区;6 = pi 鳍片用于 180° 自旋转动;7 = 样品面积和样品环境(此处为冷冻炉);8 = 第二岁差区;9 = pi/2 鳍片,用于第二个 90° 自旋转动;10 = 分析仪;11 = 探测器。(请注意,3部分以及2和1部分位于屏蔽层内部的蓝色墙后面,斩波器被基于反应堆的NSE的速度选择器取代)。 请点击此处查看此图的大图。
Protocol
Representative Results
Discussion
NSE光谱学提供了蛋白质动力学的独特而详细的视图,这是其他光谱技术无法产生的。在延长的时间尺度上进行测量可以观察蛋白质的翻译和旋转扩散,如图所示。节段动力学和其他内部振荡表明,它们在短时间内是相干散射函数 S(Q,t)的强衰减,并且与整体扩散弛豫过程很好地分离。NSE技术的主要局限性是测量时间长,并且获取良好统计信号所需的大量样品。这可能对测量具有低浓度移…
Divulgaciones
The authors have nothing to disclose.
Acknowledgements
这项研究使用了散裂中子源(BL-15,BL-6,生物和化学实验室)的资源,这是由橡树岭国家实验室运营的科学用户设施的美国能源部办公室。这项研究还使用了MLZ-FRM2反应堆加兴(KWS-2,菲尼克斯-J-NSE)和德国尤利希研究中心JCNS1的资源。作者感谢Ralf Biehl博士和Andreas Stadler博士在建模方面的帮助以及他们对IgG和MBP蛋白研究的贡献,Piotr A. Ż ołnierczuk博士为NSE数据减少支持,长武博士对SANS测量的支持,以及朗达·穆迪和凯文·韦斯博士对SNS生物化学实验室的支持。
Materials
| Bovine MBP protein solution | Sigma-Aldrich | M1891 | lyophilized powder reconstituted in D2O |
| D2O – heavy water | Sigma-Aldrich | Product No. 151882 | liquid |
| Dionized water | in house | – | for washing / cleanning cells |
| DLS instrument | Zetasizer Nano ZS, FZ-Jülich | – | dynamic light scattering instrument |
| Elastic scattering standards | SNS-NSE, ORNL | – | Al2O3 and Graphite powders |
| Ethanol | Sigma-Aldrich | 65350-M | 70% ethanol for cleaning cells |
| IgG protein solution | Sigma-Aldrich | I4506 | lyophilized powder reconstituted in D2O |
| KWS-2 instrument | JCNS outstation at the MLZ, Garching, Germany | – | small angle neutron instrument |
| Liquinox dish detergent | Alconox | – | Phosphate-free liquid lab glassware cleaner |
| Na2HPO4·7H2O | Sigma-Aldrich | Product No.S9390 | disodium phosphate heptahydrate salt |
| NaCl | Sigma-Aldrich | Product No.S9888 | sodium chloride salt |
| NaH2PO4·H2O | Sigma-Aldrich | Product No. S9638 | monosodium phosphate monohydrate salt |
| Nanodrop spectrophotometer | Thermo Scientific | Catalog number: ND-2000 | NanoDrop 2000/2000c Spectrophotometer |
| Neutron alignment camera | NeutronOptics, Grenoble | NOG210222 | 100 x 100 mm camera with Sony IMX249 CMOS sensor |
| Parafilm M – wax parafilm | Bemis | Parafilm M – 5259-04LC PM996 | all-purpose laboratory film in cardboard dispenser |
| Phoenix-J-NSE Spectrometer | JCNS outstation at the MLZ, Garching, Germany | – | neutron spectrometer |
| SasView | https://www.sasview.org/ | ||
| SAXSpace, Anton Paar instrument | FZ-Jülich | – | small angle x-ray instrument |
| Slide-A-Lyzer dialysis membranes | Thermo Scientific | 88400-88405 | Slide-A-Lyzer mini dialysis devices tubes of 3.5 K MWCO |
| SNS Remote Analysis Cluster | Neutron Science Remote Analysis (sns.gov) | https://analysis.sns.gov | |
| SNS-NSE spectrometer | ORNL, Oak Ridge, TN, USA | – | neutron spectrometer |
| Sterile syringe filters | VWR | N.A. PN:28145-501 | 0.2 µm pore size filters |
| Temperature Forcing System (TFS) | SP Scientific | Part Number 100004055 | sample environment equipment |
| Urea -d4 | Sigma-Aldrich | Product No. 176087 | deuterated Urea salt |
| Viscometer | FZ-Jülich | – | falling ball viscometer |
Referencias
- Fitter, J., Gutberlet, T., Katsaras, J. . Neutron Scattering in Biology: Techniques and Applications. , (2006).
- Stadler, A., Monkenbusch, M., Biehl, R., Richter, D., Ollivier, J. Neutron spin-echo and TOF reveals protein dynamics in solution. Journal of the Physical Society of Japan. 82, (2013).
- Inoue, R. Large domain fluctuations on 50-ns timescale enable catalytic activity in phosphoglycerate kinase. Biophysical Journal. 99 (7), 2309-2317 (2010).
- Callaway, D. J. E., et al. Controllable activation of nanoscale dynamics in a disordered protein alters binding kinetics. Journal of Molecular Biology. 429 (7), 987-998 (2017).
- Stingaciu, L. R., Biehl, R., Changwoo, D., Richter, D., Stadler, A. M. Reduced internal friction by osmolyte interaction in intrinsically disordered myelin basic protein. Journal of Physical Chemistry Letters. 11 (1), 292-296 (2020).
- Monkenbusch, M., Richter, D. High resolution neutron spectroscopy-a tool for the investigation of dynamics of polymers and soft matter. Comptes Rendus Physique. , (2007).
- Richter, D., Monkenbusch, M., Schwahn, D. Neutron Scattering. Polymer Science: A Comprehensive Reference, 10 Volume Set. , (2012).
- Wilson, C. C. . Single Crystal Neutron Diffraction From Molecular Materials. , (2000).
- Marshall, W. . Theory of thermal neutron scattering. , (1971).
- . Roger Pynn Introduction & Neutron Scattering "Theory" Available from: https://neutrons.ornl.gov/sites/default/files/intro_to_neutron_scattering.pdf (2004)
- Richter, D. Neutron scattering in polymer physics. Physica B: Condensed Matter. 276-278, 22-29 (2000).
- Harroun, T. A., Wignall, G. D., Katsaras, J. Neutron scattering for biology. Neutron Scattering in Biology. , (2006).
- . SNS Available from: https://neutrons.ornl.gov/sna (2020)
- . HFIR Available from: https://neutrons.ornl.gov/hfir (2020)
- . CNCS Available from: https://neutrons.ornl.gov/cncs (2020)
- Perticaroli, S., et al. Description of hydration water in protein (green fluorescent protein) solution. Journal of the American Chemical Society. 139 (3), 1098-1105 (2017).
- Perticaroli, S., Nickels, J. D., Ehlers, G., Sokolov, A. P. Rigidity, secondary structure, and the universality of the boson peak in proteins. Biophysical Journal. 106 (12), 2667-2674 (2014).
- Miao, Y., et al. Coupled flexibility change in cytochrome p450cam substrate binding determined by neutron scattering, NMR, and molecular dynamics simulation. Biophysical Journal. 103 (10), 2167-2176 (2012).
- Mamontov, E., Zamponi, M., Hammons, S., Keener, W. S., Hagen, M., Herwig, K. W. BASIS: A new backscattering spectrometer at the SNS. Neutron News. 19 (3), 22-24 (2008).
- Mamontov, E. Microscopic diffusion processes measured in living planarians. Scientific Reports. 9, 8708 (2018).
- Alpert, Y., Cser, L., Faragó, B., Franěk, F., Mezei, F., Ostanevich, Y. M. Segmental flexibility in pig immunoglobulin G studied by neutron spin-echo technique. Biopolymers. 24 (9), 1769-1784 (1985).
- Bu, Z., Biehl, R., Monkenbusch, M., Richter, D., Callaway, D. J. E. Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (49), 17646-17651 (2005).
- Biehl, R., et al. Direct observation of correlated interdomain motion in alcohol dehydrogenase. Physical Review Letters. 101, 138102 (2008).
- Farago, B., Li, J., Cornilescu, G., Callaway, D. J. E., Bu, Z. Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 99 (10), 3473-3482 (2010).
- Bu, Z., Callaway, D. J. E. Dynamic propagation of long-range allosteric signals by nanoscale protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 100 (3), 223 (2011).
- Hong, L., et al. Structure and dynamics of a compact state of a multidomain protein, the mercuric ion reductase. Biophysical Journal. 107 (2), 393-400 (2014).
- Longeville, S., Stingaciu, L. -. R. Hemoglobin diffusion and the dynamics of oxygen capture by red blood cells. Scientific Reports. 7, 10448 (2017).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Hahn, E. L. Nuclear induction due to free larmor precession. Physical Review. 77, 297 (1950).
- Hahn, E. L. Spin echoes. Physical Review. 80, 580 (1950).
- Svergun, D. I., Koch, M. H. J. Small-angle scattering studies of biological macromolecules in solution. Reports on Progress in Physics. 66 (10), 1735-1782 (2003).
- . Sasview Available from: https://www.sasview.org (2020)
- Zhao, J. K., Gao, C. Y., Liu, D. The extended Q-range small-angle neutron scattering diffractometer at the SNS. Journal of Applied Crystallography. 43, 1068-1077 (2010).
- Ohl, M., et al. The high-resolution neutron spin-echo spectrometer for the SNS with τ ≥ 1 µs. Physica B: Condensed Matter. 350, 147-150 (2004).
- . SNS-NSE web page Available from: https://neutrons.ornl.gov/nse (2022)
- Ohl, M., et al. The spin-echo spectrometer at the Spallation Neutron Source (SNS). Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 696, 85-99 (2012).
- Zolnierczuk, P. A., Holderer, O., Pasini, S., Kozielewski, T., Stingaciu, L. R., Monkenbusch, M. Efficient data extraction from neutron time-of-flight spin-echo raw data. Journal of Applied Crystallography. 52 (5), 1022-1034 (2019).
- . Dr Spine hub Available from: https://jugit.fz-juelich.de/nse/drspine (2022)
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), (2014).
- . FZJ Available from: https://www.fz-juelich.de/portal/EN/AboutUs (2022)
- . KWS2 Available from: https://miz-garching.de/kws-2 (2022)
- Moorhouse, M., Barry, P. The protein databank. Bioinformatics Biocomputing and Perl. , (2005).
- Tria, G., Mertens, H. D. T., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (2), 207-217 (2015).
- Holderer, O., Monkenbusch, M., Schätzler, R., Kleines, H., Westerhausen, W., Richter, D. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 90 (4), 043107 (2008).
- Biehl, R., Monkenbusch, M., Richter, D. Exploring internal protein dynamics by neutron spin echo spectroscopy. Soft Matter. 7 (4), 1299-1307 (2011).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), 6987-6994 (2014).
- Biehl, R., Richter, D. Slow internal protein dynamics in solution. Journal of Physics: Condensed Matter. 26 (50), 503103 (2014).
- Hinsen, K. The molecular modeling toolkit: A new approach to molecular simulations. Journal of Computational Chemistry. 21 (2), 79-85 (2000).
- Uhlenbeck, G. E., Ornstein, L. S. On the theory of the Brownian motion. Physical Review. 36 (5), 823 (1930).
- Wang, M. C., Uhlenbeck, G. E. On the theory of the Brownian motion II. Reviews of Modern Physics. 17, 323 (1945).
- Callaway, D. J. E., Bu, Z. Nanoscale protein domain motion and long-range allostery in signaling proteins-a view from neutron spin echo spectroscopy. Biophysical Reviews. 7 (2), 165-174 (2015).
- Liu, Y. Intermediate scattering function for macromolecules in solution probed by neutron spin echo. Physical Review E. 95, 020501 (2017).
- Liu, Y. Short-time dynamics of proteins in solutions studied by neutron spin echo. Current Opinion in Colloid & Interface Science. 42, 147-156 (2019).

