면역 세포에 의한 일시적인 피코네톤 수용체 힘을 매핑하는 DNA 장력 프로브
Summary
이 논문은 DNA 기반 장력 프로브를 사용하여 면역 세포에 의해 가해지는 수용체 힘을 이미지화하기 위한 자세한 프로토콜을 설명합니다. 이 접근법은 수용체 힘>4.7pN을 실시간으로 매핑할 수 있으며 시간이 지남에 따라 힘을 통합할 수 있습니다.
Abstract
인접한 두 세포 사이의 접합부와 세포와 세포외 기질 사이의 접합부에서 전달되는 기계적 힘은 발달에서 면역학에 이르는 많은 과정을 조절하는 데 중요합니다. 따라서 분자 규모에서 이러한 힘을 연구하는 도구를 개발하는 것이 중요합니다. 우리 그룹은 세포에서 생성되어 특정 리간드로 전달되는 힘을 정량화하고 시각화하기 위해 분자 장력 센서 제품군을 개발했습니다. 가장 민감한 종류의 분자 장력 센서는 핵산 줄기 루프 헤어핀으로 구성됩니다. 이 센서는 형광단-소광제 쌍을 사용하여 힘을 받고 있는 DNA 헤어핀의 기계적 확장 및 풀림을 보고합니다. DNA 헤어핀 장력 센서의 한 가지 문제는 장력이 종료될 때 빠르게 헤어핀을 다시 접을 수 있어 일시적인 힘을 기록하기 어렵다는 것입니다. 이 기사에서는 기계적 정보를 “저장”할 수 있도록 “잠길” 수 있고 다시 접히는 것을 방지할 수 있는 DNA 장력 센서를 준비하기 위한 프로토콜에 대해 설명합니다. 이를 통해 매우 일시적인 피코네톤 힘을 기록할 수 있으며, 이는 잠금 장치를 제거하는 상보적 핵산을 추가하여 이후에 “삭제”할 수 있습니다. 실시간 장력 매핑과 기계적 정보 저장 사이를 전환하는 이 기능은 T 세포가 면역 기능의 일부로 일반적으로 사용하는 약하고 수명이 짧으며 덜 풍부한 힘을 보여줍니다.
Introduction
면역 세포는 항원에 대해 표적 세포의 표면을 지속적으로 크롤링하고 스캔하여 표면을 박음으로써 병원체와 암세포를 방어합니다 1,2. 항원 인식은 T 세포 수용체(TCR)와 표적 세포 표면에서 발현되는 펩타이드-주요 조직적합성 복합체 MHC(pMHC) 복합체 사이의 결합에 따라 시작됩니다. TCR-pMHC 인식은 두 이동 셀 사이의 접합부에서 발생하기 때문에 오랫동안 기계적 힘이 발생하는 것으로 의심되어 왔습니다. 또한, 이것은 TCR 활성화의 기계 센서 모델로 이어졌으며, 이는 TCR 힘이 기능 3,4에 기여한다는 것을 시사합니다. 언제, 어디서, 어떻게 기계적 힘이 T 세포 기능에 기여하는지 이해하려면 T 세포에 의해 전달되는 분자력을 시각화하는 도구를 개발하는 것이 필수적입니다. 전통적으로, 견인력 현미경 (TFM) 및 마이크로 필러 어레이와 같은 방법은 세포력 5,6을 조사하는 데 사용됩니다. 그러나 TFM과 마이크로필러 어레이의 힘 감도는 나노뉴턴(nN) 규모이므로 세포 수용체에 의해 전달되는 분자 피코네톤(pN) 힘을 연구하기에는 불충분한 경우가 많다7. 검출을 위한 힘과 공간 분해능을 개선하기 위해 우리 연구실은 분자 장력 프로브의 개발을 개척했으며, 이 프로브는 초기에 폴리에틸렌 글리콜(PEG) 폴리머를 사용하여 합성되었습니다7. 분자 장력 프로브는 형광단과 소광제 옆에 있는 확장 가능한 분자 “스프링”(PEG, 단백질, DNA)으로 구성되며 표면에 고정되어 있습니다. 프로브의 말단에 가해지는 힘은 프로브의 확장으로 이어져 형광단과 소광제를 분리하여 강력한 형광 신호를 생성합니다(그림 1A)8,9,10.
지난 10년 동안 우리는 핵산(nucleic acids)11, 단백질(proteins) 10 및 폴리머(polymers) 8로 만들어진 스프링 요소(spring elements)를 가진 다양한 종류의 분자 장력 프로브(molecular tension probe) 라이브러리를 개발해 왔습니다. 이 중 DNA 기반 장력 프로브는 가장 높은 신호 대 잡음비와 가장 큰 힘 감도를 제공하며, 이는 몇 pN에서 최대 ~20pN까지 쉽게 조정할 수 있습니다11. 우리는 이러한 실시간 DNA 장력 프로브를 사용하여 섬유아세포, 암세포, 혈소판 및 면역 세포를 포함한 다양한 세포 유형에서 생성되는 분자력을 연구했습니다11,12,13. 이 원고는 기존의 형광 현미경을 사용하여 pN 힘 분해능으로 분자 수용체 힘을 매핑하기 위해 표면에 DNA 장력 프로브를 합성하고 조립하는 프로토콜을 설명합니다. 현재 절차에는 형광 리포터를 도입하기 위한 핵산에 대한 화학적 변형이 포함되지만(그림 1B), 많은 수정 및 정제 단계를 맞춤형 DNA 합성 회사에 아웃소싱할 수 있다는 점에 유의해야 합니다. 따라서 DNA 장력 프로브 기술은 용이하고 광범위한 세포 생물학 및 기계 생물학 커뮤니티에서 접근할 수 있습니다.
간단히 말해서, DNA 장력 센서를 조립하기 위해 DNA 헤어핀을 한쪽 팔의 형광 리간드 가닥과 다른 쪽 팔의 소광제 앵커 가닥에 혼성화한 다음 유리 기판에 고정합니다(그림 1C, 실시간 장력). 기계적 힘이 없으면 머리핀이 닫히고 형광이 소멸됩니다. 그러나 적용된 기계적 힘이 F1/2 (풀림 확률 50%로 이어지는 평형 상태의 힘)보다 크면 헤어핀이 기계적으로 녹고 형광 신호가 생성됩니다.
실시간 DNA 장력 센서를 기반으로 축적된 힘을 매핑하는 프로토콜도 설명하는데, 이는 면역 세포의 수용체와 천연 리간드 간의 상호 작용을 연구하는 데 특히 유용합니다. 이것은 면역 수용체가 종종 수명이 짧은 결합을 나타내기 때문입니다 3,14. 축적된 힘은 열린 DNA 헤어핀에 우선적으로 결합하고 기계적 당김 이벤트와 관련된 형광 신호를 저장할 수 있는 “잠금” 가닥을 사용하여 이미지화됩니다(그림 1C, 고정 장력). 상기 잠금 스트랜드는 기계적으로 유도된 헤어핀의 용융 시 노출되는 비밀 결합 부위를 묶고, 헤어핀 재접힘을 차단하여 헤어핀을 개방 상태로 고정시켜 장력 신호를 저장하고, 축적된 장력 맵을 생성하도록 설계된다. 또한, 잠금 가닥은 8개의 뉴클레오티드 토홀드로 설계되어 완전한 보완인 “잠금 해제” 가닥으로 발가락 매개 가닥 변위 반응을 가능하게 합니다. 잠금 해제 스트랜드가 추가되면 바운드 잠금 스트랜드가 헤어핀 구조에서 벗겨져 저장된 장력 신호가 지워지고 헤어핀이 실시간 상태로 재설정됩니다.
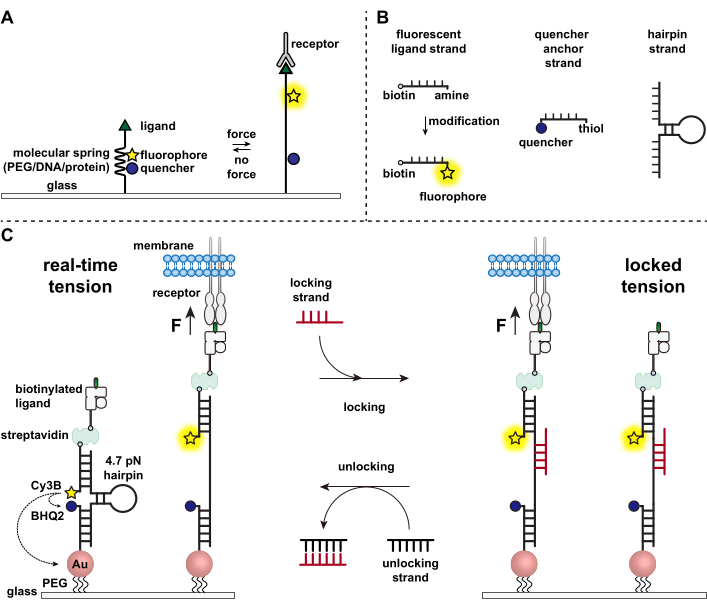
그림 1: 최첨단 분자 장력 프로브의 구성표. (A) 실시간 분자 장력 프로브의 일반 설계, (B) DNA 기반 장력 프로브 구성을 위한 가닥, (C) 설계된 DNA 기반 장력 프로브 및 실시간 상태와 잠금 상태 간의 전환. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
주요 프로토콜은 올리고뉴클레오티드 준비, 표면 처리, 이미징 및 데이터 분석의 네 가지 주요 섹션으로 구성됩니다. 이 프로토콜은 순진하고 활성화된 OT-1 CD8+ T 세포, OT-II CD4+ 세포 및 하이브리도마에서 우리 연구실과 다른 사람들에 의해 성공적으로 입증되었으며 T 세포 수용체, 프로그래밍된 세포 사멸 수용체(PD1) 및 림프구 기능 관련 항원 1(LFA-1) 힘을 포함한 다양한 면역 세포 수용체를 조사하는 데 적용할 수 있습니다. OT-1 CD8+ 나이브 T 세포는 본 논문에서 세포주의 예로 사용됩니다.
Protocol
Representative Results
Discussion
여기에 제공된 자세한 절차를 통해 DNA 헤어핀 장력 프로브 기질을 준비하여 면역 세포에 의해 생성된 수용체 장력을 매핑하고 정량화할 수 있습니다. 세포가 DNA 헤어핀 장력 프로브 기판에 도금되면 수용체가 화학적으로나 기계적으로 리간드를 감지함에 따라 착지, 부착 및 확산되며, 후자는 프로브에 의해 감지됩니다. 그러나 어떤 경우에는 세포가 퍼지지 않을 수 있습니다 (그림…
Divulgaciones
The authors have nothing to disclose.
Acknowledgements
이 작업은 NIH 보조금 R01GM131099, NIH R01GM124472 및 NSF CAREER 1350829의 지원을 받았습니다. pMHC 리간드에 대해 NIH Tetramer Facility에 감사드립니다. 이 연구는 Emory Comprehensive Glycomics Core에 의해 부분적으로 지원되었습니다.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Referencias
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

