Sondas de Tensão de DNA para Mapear as Forças Transitórias do Receptor de Piconewton por Células Imunes
Summary
Este artigo descreve um protocolo detalhado para o uso de sondas de tensão baseadas em DNA para obter imagens das forças receptoras aplicadas por células imunes. Essa abordagem pode mapear forças receptoras >4.7pN em tempo real e pode integrar forças ao longo do tempo.
Abstract
As forças mecânicas transmitidas na junção entre duas células vizinhas e na junção entre as células e a matriz extracelular são críticas para a regulação de muitos processos que vão desde o desenvolvimento até a imunologia. Portanto, desenvolver ferramentas para estudar essas forças em escala molecular é fundamental. Nosso grupo desenvolveu um conjunto de sensores de tensão molecular para quantificar e visualizar as forças geradas pelas células e transmitidas a ligantes específicos. A classe mais sensível de sensores de tensão molecular é composta por grampos de alça de haste de ácido nucleico. Esses sensores usam pares fluoróforo-quencher para relatar a extensão mecânica e o desdobramento de pinos de cabelo de DNA sob força. Um desafio com os sensores de tensão hairpin de DNA é que eles são reversíveis com redobramento rápido do hairpin após o término da tensão e, portanto, as forças transitórias são difíceis de registrar. Neste artigo, descrevemos os protocolos de preparação de sensores de tensão de DNA que podem ser “travados” e impedidos de redobrar para permitir o “armazenamento” de informações mecânicas. Isso permite o registro de forças de piconewton altamente transitórias, que podem ser posteriormente “apagadas” pela adição de ácidos nucleicos complementares que removem a fechadura. Essa capacidade de alternar entre o mapeamento de tensão em tempo real e o armazenamento de informações mecânicas revela forças fracas, de curta duração e menos abundantes, que são comumente empregadas pelas células T como parte de suas funções imunológicas.
Introduction
As células imunes defendem-se contra patógenos e células cancerígenas rastejando e varrendo continuamente as superfícies das células-alvo em busca de antígenos, fixando sua superfície 1,2. O reconhecimento do antígeno é iniciado após a ligação entre o receptor de células T (TCR) e o complexo MHC (pMHC) do complexo peptídeo-principal de histocompatibilidade expresso na superfície das células-alvo. Como o reconhecimento do TCR-pMHC ocorre na junção entre duas células móveis, há muito se suspeita que ele experimente forças mecânicas. Além disso, isso levou ao modelo mecanosensor de ativação do TCR, que sugere que as forças do TCR contribuem para sua função 3,4. Para entender quando, onde e como as forças mecânicas contribuem para o funcionamento das células T, é imperativo desenvolver ferramentas para visualizar as forças moleculares transmitidas pelas células T. Tradicionalmente, métodos como a microscopia de força de tração (MFT) e os arranjos de micropilares são utilizados para investigar as forças celulares 5,6. No entanto, a sensibilidade à força dos arranjos de MTF e micropilares está na escala de nanonewton (nN) e, portanto, muitas vezes é insuficiente para estudar as forças moleculares de piconewton (pN) transmitidas por receptores celulares7. Para melhorar a força e a resolução espacial para detecção, nosso laboratório foi pioneiro no desenvolvimento de sondas de tensão molecular, que foram inicialmente sintetizadas usando polímeros de polietilenoglicol (PEG)7. As sondas de tensão molecular são compostas por uma “mola” molecular extensível (PEG, proteína, DNA) flanqueada por um fluoróforo e quencher e estão ancoradas em uma superfície. Forças aplicadas ao término da sonda levam à sua extensão, separando o fluoróforo e o quencher, gerando um forte sinal de fluorescência (Figura 1A)8,9,10.
Na última década, desenvolvemos uma biblioteca de diferentes classes de sondas de tensão molecular com elementos de mola feitos de ácidos nucléicos11, proteínas10 e polímeros8. Dentre estas, as sondas de tensão baseadas em DNA fornecem a maior relação sinal/ruído e a maior sensibilidade à força, que é facilmente sintonizada de alguns pN até ~20 pN11. Usamos essas sondas de tensão de DNA em tempo real para estudar as forças moleculares geradas por diversos tipos celulares, incluindo fibroblastos, células cancerosas, plaquetas e células imunes11,12,13. Este manuscrito descreverá protocolos para sintetizar e montar sondas de tensão de DNA em uma superfície para mapear forças de receptores moleculares com resolução de força de pN usando um microscópio de fluorescência convencional. Embora o procedimento atual inclua modificações químicas no ácido nucleico para introduzir o repórter fluorescente (Figura 1B), é importante notar que muitas das etapas de modificação e purificação podem ser terceirizadas para empresas de síntese de DNA personalizadas. Portanto, a tecnologia de sondas de tensão de DNA é fácil e acessível para as comunidades mais amplas de biologia celular e mecanobiologia.
Resumidamente, para montar sensores de tensão de DNA, um hairpin de DNA é hibridizado a uma fita fluorescente ligante em um braço e uma fita de âncora quencher no outro braço e, em seguida, imobilizado em um substrato de vidro (Figura 1C, tensão em tempo real). Na ausência de força mecânica, o hairpin é fechado e, assim, a fluorescência é apagada. No entanto, quando a força mecânica aplicada é maior que o F1/2 (a força em equilíbrio que leva a uma probabilidade de 50% de desdobramento), o hairpin derrete mecanicamente e um sinal fluorescente é gerado.
Com base no sensor de tensão do DNA em tempo real, também descrevemos protocolos para mapear forças acumuladas, o que é particularmente útil para estudar interações entre receptores em células imunes e seu ligante natural. Isso ocorre porque os receptores imunes frequentemente apresentam ligações de curta duração 3,14. As forças acumuladas são imageadas usando uma fita de “travamento” que se liga preferencialmente aos pinos de cabelo de DNA abertos e permite o armazenamento de sinais de fluorescência associados a eventos de tração mecânica (Figura 1C, tensão bloqueada). O fio de bloqueio é projetado para ligar um local de ligação críptico que é exposto após o derretimento mecanicamente induzido do hairpin e travar o hairpin no estado aberto, bloqueando a redobração do hairpin, armazenando assim o sinal de tensão e gerando um mapa de tensão acumulado. Além disso, a fita de travamento é projetada com um suporte de oito nucleotídeos, que permite uma reação de deslocamento de fita mediada por dedos dos pés com seu complemento completo, a fita de “destravamento”. Com a adição do fio de desbloqueio, o fio de travamento preso é retirado da construção do hairpin, apagando o sinal de tensão armazenado e redefinindo o hairpin de volta ao estado em tempo real.
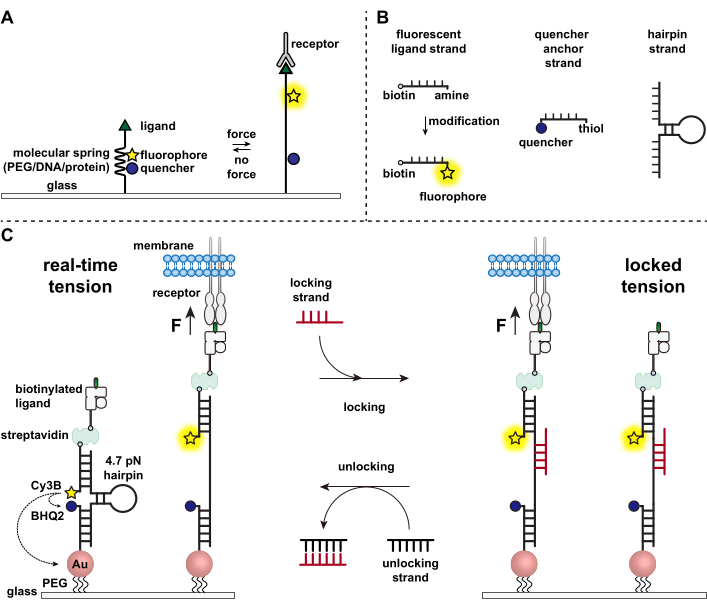
Figura 1: Esquema das sondas de tensão molecular de última geração. (A) Projeto geral de sonda de tensão molecular em tempo real, (B) Fitas para a construção de sonda de tensão baseada em DNA e (C) Sondas de tensão baseadas em DNA projetadas e sua alternância entre o estado em tempo real e o estado bloqueado. Clique aqui para ver uma versão maior desta figura.
O protocolo principal consiste em quatro seções principais – preparação de oligonucleotídeos, preparação de superfície, imagem e análise de dados. Este protocolo foi demonstrado com sucesso por nosso laboratório e outros em células T CD8+ OT-1 virgens e ativadas, células CD4+ OT-II, bem como hibridomas, e pode ser aplicado para interrogar diferentes receptores de células imunes, incluindo receptor de células T, receptor de morte celular programada (PD1) e forças do antígeno 1 associado à função linfocitária (LFA-1). Células T virgens de OT-1 CD8+ são usadas como um exemplo de linhagem celular neste artigo.
Protocol
Representative Results
Discussion
Com os procedimentos detalhados fornecidos aqui, pode-se preparar substratos de sonda de tensão de DNA hairpin para mapear e quantificar a tensão do receptor produzida pelas células imunes. Quando as células são plaqueadas no substrato da sonda de tensão do pino de DNA, elas pousam, se ligam e se espalham à medida que os receptores detectam os ligantes química e mecanicamente, o último dos quais é detectado por nossas sondas. No entanto, em alguns casos, as células podem não se espalhar (…
Divulgaciones
The authors have nothing to disclose.
Acknowledgements
Este trabalho foi apoiado pelos NIH Grants R01GM131099, NIH R01GM124472 e NSF CAREER 1350829. Agradecemos ao NIH Tetramer Facility pelos ligantes pMHC. Este estudo foi apoiado, em parte, pelo Emory Comprehensive Glycomics Core.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Referencias
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

