RNA-seq Verilerinde Alternatif Ekleme ve Poliadenilasyonun Tanımlanması
Summary
Alternatif ekleme (AS) ve alternatif poliadenilasyon (APA), transkript izoformlarının ve ürünlerinin çeşitliliğini genişletir. Burada, deneysel koşullar arasında değişen AS ve APA’yı tespit etmek ve görselleştirmek için toplu RNA-seq ve 3′ uç dizileme testlerini analiz etmek için biyoinformatik protokolleri açıklıyoruz.
Abstract
Deneysel / biyolojik koşullar boyunca diferansiyel gen ekspresyonunu (DGE) ölçmek için RNA-Seq’in tipik analizinin yanı sıra, RNA-seq verileri ekzon seviyesindeki diğer karmaşık düzenleyici mekanizmaları keşfetmek için de kullanılabilir. Alternatif ekleme ve poliadenilasyon, transkripsiyon sonrası seviyede gen ekspresyonunu düzenlemek için farklı izoformlar üreterek bir genin fonksiyonel çeşitliliğinde çok önemli bir rol oynar ve analizleri tüm gen seviyesine sınırlamak bu önemli düzenleyici katmanı kaçırabilir. Burada, Bioconductor ve DEXSeq, Limma paketinden diffSplice ve rMATS dahil olmak üzere diğer paketleri ve fonksiyonları kullanarak, koşullar arasında diferansiyel ekzon ve poliadenilasyon sahası kullanımının tanımlanması ve görselleştirilmesi için ayrıntılı adım adım analizler gösteriyoruz.
Introduction
RNA-seq, yıllar boyunca tipik olarak diferansiyel gen ekspresyonunu ve gen keşfini tahmin etmek için yaygın olarak kullanılmıştır1. Ek olarak, farklı izoformları ifade eden gen nedeniyle değişen ekzon seviyesi kullanımını tahmin etmek için de kullanılabilir, böylece transkripsiyon sonrası seviyede gen düzenlemesinin daha iyi anlaşılmasına katkıda bulunur. Ökaryotik genlerin çoğunluğu, mRNA ekspresyonunun çeşitliliğini arttırmak için alternatif ekleme (AS) ile farklı izoformlar üretir. AS olayları farklı kalıplara ayrılabilir: bir (“kaset”) ekzonun yan tarafındaki intronlarla birlikte transkriptten tamamen çıkarıldığı tam ekzonların (SE) atlanması; ekzonun her iki ucunda iki veya daha fazla ekleme bölgesi bulunduğunda alternatif (donör) 5′ ekleme yeri seçimi (A5SS) ve alternatif 3′ (alıcı) ekleme yeri seçimi (A3SS); Bir intron olgun mRNA transkriptinde tutulduğunda intronların (RI) tutulması ve mevcut iki ekzondan sadece birininbir seferde tutulabildiği ekzon kullanımının (MXE) karşılıklı dışlanması 2,3. Alternatif poliadenilasyon (APA), tek bir transkript4’ten çoklu mRNA izoformları üretmek için alternatif poli (A) bölgeleri kullanarak gen ekspresyonunun düzenlenmesinde de önemli bir rol oynar. Çoğu poliadenilasyon bölgesi (pAs), 3′ çevrilmemiş bölgede (3′ UTR’ler) bulunur ve çeşitli 3′ UTR uzunluklarına sahip mRNA izoformları üretir. 3′ UTR, düzenleyici unsurları tanımak için merkezi merkez olduğundan, farklı 3′ UTR uzunlukları mRNA lokalizasyonunu, kararlılığını ve translasyonunu etkileyebilir5. Protokol6’nın ayrıntılarında farklılık gösteren APA’yı tespit etmek için optimize edilmiş bir 3′ uç sıralama tahlilleri sınıfı vardır. Burada açıklanan boru hattı PolyA-seq için tasarlanmıştır, ancak açıklandığı gibi diğer protokoller için uyarlanabilir.
Bu çalışmada, ekzon bazlı (DEXSeq9, diffSplice10) ve olay tabanlı (Multivariate Analysis of Transcript Splicing (rMATS)11) olmak üzere iki geniş kategoriye ayrılabilen diferansiyel ekzon analiz yöntemleri 7,8 (Şekil 1) boru hattını sunuyoruz. Ekzon tabanlı yöntemler, bireysel ekzonların koşulları arasındaki kıvrım değişimini, farklı şekilde ifade edilen ekzon kullanımını çağırmak için genel gen kıvrım değişiminin bir ölçüsüyle karşılaştırır ve bundan AS aktivitesinin gen düzeyinde bir ölçüsünü hesaplar. Olay tabanlı yöntemler, ekzon atlama veya intronların tutulması gibi belirli ekleme olaylarını algılamak ve sınıflandırmak için ekzon intronunu kapsayan bağlantı okumalarını kullanır ve çıktı3’teki bu AS türlerini ayırt eder. Bu nedenle, bu yöntemler AS12,13’ün tam bir analizi için tamamlayıcı görüşler sağlar. Diferansiyel ekleme analizi için en yaygın kullanılan paketler arasında yer aldıkları için çalışma için DEXSeq (DESeq214 DGE paketine dayanarak) ve diffSplice (Limma10 DGE paketine dayanarak) seçtik. rMATS, olay tabanlı analiz için popüler bir yöntem olarak seçildi. Bir başka popüler olay tabanlı yöntem MISO (İzoform Karışımı)1’dir. APA için ekzon tabanlı yaklaşımı uyarlıyoruz.
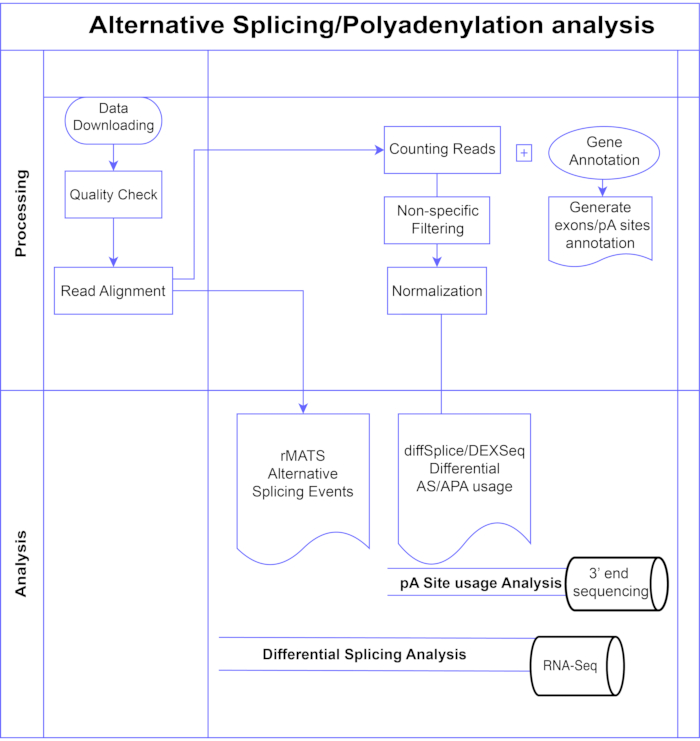
Şekil 1. Analiz işlem hattı. Analizde kullanılan adımların akış şeması. Adımlar şunları içerir: verileri elde etmek, kalite kontrolleri yapmak ve okuma hizalaması, ardından bilinen ekzonlar, intronlar ve pA siteleri için ek açıklamalar kullanarak okumaları saymak, düşük sayıları kaldırmak için filtreleme ve normalleştirme. PolyA-seq verileri diffSplice/DEXSeq yöntemleri kullanılarak alternatif pA bölgeleri için, bulk RNA-Seq diffSplice/DEXseq yöntemleri ile ekzon düzeyinde alternatif ekleme için ve AS olayları rMATS ile analiz edilmiştir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Bu araştırmada kullanılan RNA-seq verileri, Gen İfade Omnibus’undan (GEO) (GSE138691)15 elde edilmiştir. Bu çalışmadan elde edilen fare RNA-seq verilerini iki koşul grubuyla kullandık: vahşi tip (WT) ve her biri üç kopya ile Kas körü benzeri tip 1 nakavt (Mbnl1 KO). Diferansiyel poliadenilasyon alanı kullanım analizini göstermek için, fare embriyo fibroblastları (MEF’ler) PoliA-seq verilerini elde ettik (GEO Katılımı GSE60487)16. Verilerin dört koşul grubu vardır: Wild-type (WT), Kas körü benzeri tip1/tip 2 çift nakavt (Mbnl1/2 DKO), Mbnl3 knockdown (KD) ile Mbnl 1/2 DKO ve Mbnl3 kontrollü Mbnl1/2 DKO (Ctrl). Her koşul grubu iki çoğaltmadan oluşur.
| GEO Katılımı | SRA Çalıştırma numarası | Örnek adı | Koşul | Çoğaltmak | Doku | Sıralama | Okuma uzunluğu | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 nakavt | Temsilci 1 | Timus | Eşleştirilmiş uç | 100 bp |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 nakavt | Temsilci 2 | Timus | Eşleştirilmiş uç | 100 bp | |
| GSM4116220 göster | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 nakavt | Temsilci 3 | Timus | Eşleştirilmiş uç | 100 bp | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Vahşi tip | Temsilci 1 | Timus | Eşleştirilmiş uç | 100 bp | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Vahşi tip | Temsilci 2 | Timus | Eşleştirilmiş uç | 100 bp | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Vahşi tip | Temsilci 3 | Timus | Eşleştirilmiş uç | 100 bp | |
| 3P-Seks | GSM1480973 | SRR1553129 | WT_1 | Vahşi tip (WT) | Temsilci 1 | Fare embriyonik Fibroblastları (MEF’ler) | Tek uçlu | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | Vahşi tip (WT) | Temsilci 2 | Fare embriyonik Fibroblastları (MEF’ler) | Tek uçlu | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 çift nakavt (DKO) | Temsilci 1 | Fare embriyonik Fibroblastları (MEF’ler) | Tek uçlu | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 çift nakavt (DKO) | Temsilci 2 | Fare embriyonik Fibroblastları (MEF’ler) | Tek uçlu | 40 bp | |
| GSM1480977 göster | SRR1553133 | DKOsiRNA_1 | Mbnl 3 siRNA (KD) ile Mbnl 1/2 çift nakavt | Temsilci 1 | Fare embriyonik Fibroblastları (MEF’ler) | Tek uçlu | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 3 siRNA (KD) ile Mbnl 1/2 çift nakavt | Temsilci 2 | Fare embriyonik Fibroblastları (MEF’ler) | Tek uçlu | 36 bg | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 çift nakavt, hedeflemeyen siRNA (Ctrl) ile | Temsilci 1 | Fare embriyonik Fibroblastları (MEF’ler) | Tek uçlu | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 çift nakavt, hedeflemeyen siRNA (Ctrl) ile | Temsilci 2 | Fare embriyonik Fibroblastları (MEF’ler) | Tek uçlu | 40 bp |
Tablo 1. Analiz için kullanılan RNA-Seq ve PolyA-seq veri setlerinin özeti.
Protocol
Representative Results
Discussion
Bu çalışmada, toplu RNA-Seq ve 3′ uç dizileme verilerinde AS ve APA’yı saptamak için ekzon tabanlı ve olay tabanlı yaklaşımlar değerlendirildi. Ekzon tabanlı AS yaklaşımları, hem diferansiyel olarak eksprese edilen ekzonların bir listesini hem de genel gen seviyesi diferansiyel ekleme aktivitesinin istatistiksel önemine göre sıralanmış bir gen seviyesi sıralaması üretir (Tablo 1-2, 4-5). Diferansiyel kullanım, bir ekzonun diferansiyel log kıvrım değişimini aynı gen içindeki…
Divulgaciones
The authors have nothing to disclose.
Acknowledgements
Bu çalışma, Avustralya Araştırma Konseyi (ARC) Future Fellowship (FT16010043) ve ANU Futures Scheme tarafından desteklenmiştir.
Referencias
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3′ end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).

