Hochdurchsatz-Screening mikrobieller Isolate mit Auswirkungen auf die Gesundheit von Caenorhabditis elegans
Summary
Darmmikroben können die Gesundheit ihres Wirts über spezifische oder konservierte Mechanismen positiv oder negativ beeinflussen. Caenorhabditis elegans ist eine bequeme Plattform, um nach solchen Mikroben zu suchen. Das vorliegende Protokoll beschreibt das Hochdurchsatz-Screening von 48 Bakterienisolaten auf Auswirkungen auf die Nematoden-Stressresistenz, die als Proxy für die Gesundheit von Würmern verwendet werden.
Abstract
Mit seiner geringen Größe, kurzen Lebensdauer und einfachen Genetik bietet Caenorhabditis elegans eine bequeme Plattform, um die Auswirkungen mikrobieller Isolate auf die Wirtsphysiologie zu untersuchen. Es fluoresziert auch blau, wenn es stirbt, was eine bequeme Möglichkeit bietet, den Tod zu lokalisieren. Diese Eigenschaft wurde genutzt, um markierungsfreie C. elegans-Überlebenstests (LFASS) mit hohem Durchsatz zu entwickeln. Dabei handelt es sich um die Zeitraffer-Fluoreszenzaufzeichnung von Wurmpopulationen in Multiwell-Platten, aus denen die mediane Todeszeit der Population abgeleitet werden kann. Die vorliegende Studie wendet den LFASS-Ansatz an, um mehrere mikrobielle Isolate gleichzeitig auf die Auswirkungen auf die Anfälligkeit von C. elegans gegenüber starker Hitze und oxidativem Stress zu untersuchen. Eine solche mikrobielle Screening-Pipeline, die insbesondere zum Vorscreening von Probiotika verwendet werden kann, wobei schwere Stressresistenz als Proxy für die Gesundheit des Wirts verwendet wird, wird hier berichtet. Das Protokoll beschreibt, wie sowohl C. elegans-Darmmikrobiota-Isolatsammlungen als auch synchrone Wurmpopulationen in Multiwell-Arrays gezüchtet werden, bevor sie für die Assays kombiniert werden. Das vorgelegte Beispiel umfasst die Prüfung von 47 Bakterienisolaten und einem Kontrollstamm auf zwei Wurmstämme in zwei parallelen Stressassays. Die Ansatzpipeline ist jedoch leicht skalierbar und auf das Screening vieler anderer Modalitäten anwendbar. Somit bietet es ein vielseitiges Setup, um schnell eine multiparametrische Landschaft biologischer und biochemischer Bedingungen zu untersuchen, die sich auf die Gesundheit von C. elegans auswirken.
Introduction
Der menschliche Körper beherbergt schätzungsweise 10-100 Billionen lebende mikrobielle Zellen (Bakterien, Archaeenpilze), die hauptsächlich in der Darm-, Haut- und Schleimhautumgebung vorkommen1. In einem gesunden Zustand bieten diese ihrem Wirt Vorteile, einschließlich Vitaminproduktion, Reifung des Immunsystems, Stimulation angeborener und adaptiver Immunantworten auf Krankheitserreger, Regulierung des Fettstoffwechsels, Modulation von Stressreaktionen und mehr, mit Auswirkungen auf Wachstum und Entwicklung, Krankheitsbeginn und Alterung 2,3,4,5 . Die Darmmikrobiota entwickelt sich auch im Laufe des Lebens erheblich. Die drastischste Entwicklung findet im Säuglingsalter und in der frühen Kindheit statt6, aber auch mit zunehmendem Alter treten signifikante Veränderungen auf, einschließlich einer Abnahme der Bifidobacterium-Häufigkeit und einer Zunahme von Clostridium, Lactobacillus, Enterobacteriaceae und Enterococcus-Arten 7. Der Lebensstil kann die mikrobielle Zusammensetzung des Darms weiter verändern, was zu Dysbiose führt (Verlust nützlicher Bakterien, übermäßiges Wachstum opportunistischer Bakterien), was zu verschiedenen Pathologien wie entzündlichen Darmerkrankungen, Diabetes und Fettleibigkeit führt5, aber auch zu Alzheimer und Parkinson beiträgt 8,9,10,11.
Diese Erkenntnis hat entscheidend dazu beigetragen, das Konzept der Darm-Hirn-Achse (GBA) zu verfeinern, bei der Wechselwirkungen zwischen der Darmphysiologie (die jetzt die darin enthaltenen Mikroben einschließt) und dem Nervensystem als Hauptregulator des tierischen Stoffwechsels und der physiologischen Funktionen angesehen werden12. Die genaue Rolle der Mikrobiota bei der Darm-Hirn-Signalübertragung und die damit verbundenen Wirkmechanismen sind jedoch noch lange nicht vollständig verstanden13. Da die Darmmikrobiota eine Schlüsseldeterminante für gesundes Altern ist, ist die Art und Weise, wie Bakterien den Alterungsprozess modulieren, Gegenstand intensiver Forschung und Kontroversen geworden 6,14,15.
Mit dem Nachweis, dass der Spulwurm Caenorhabditis elegans eine bonafide Darmmikrobiota beherbergt, die – wie bei anderen Arten – von Bacteroidetes, Firmicutes und Actinobacteria dominiert wird 16,17,18,19,20, sein rasanter Aufstieg als experimentelle Plattform zur Untersuchung kommensaler Interaktionen zwischen Wirt und Darm21,22,23,24 ,25,26 hat unser Ermittlungsarsenal 26,27,28,29 erheblich erweitert. Insbesondere Hochdurchsatz-experimentelle Ansätze, die für C. elegans zur Verfügung stehen, um Gen-Diät-, Gen-Medikament-, Gen-Pathogen-Interaktionen usw. zu untersuchen, können angepasst werden, um schnell zu untersuchen, wie bakterielle Isolate und Cocktails die Gesundheit und das Altern von C. elegans beeinflussen.
Das vorliegende Protokoll beschreibt eine experimentelle Pipeline, um sofort Arrays von Bakterienisolaten oder Mischungen in Multiwell-Platten auf Auswirkungen auf die Stressresistenz von C. elegans als Proxy für die Gesundheit zu untersuchen, die zur Identifizierung von Probiotika verwendet werden können. Es wird detailliert beschrieben, wie große Wurmpopulationen gezüchtet und bakterielle Arrays in 96- und 384-Well-Plattenformaten gehandhabt werden, bevor Würmer für die automatisierte Stressresistenzanalyse mit einem Fluoreszenzplattenleser verarbeitet werden (Abbildung 1). Der Ansatz basiert auf markierungsfreien automatisierten Überlebensassays (LFASS)30, die das Phänomen der Todesfluoreszenz31 ausnutzen, wobei sterbende Würmer einen Ausbruch blauer Fluoreszenz erzeugen, mit dem der Zeitpunkt des Todes bestimmt werden kann. Blaue Fluoreszenz wird von Glucosylestern der Anthranilsäure emittiert, die in C. elegans-Darmgranulaten (einer Art Lysosomen-verwandter Organelle) gespeichert sind, die platzen, wenn eine nekrotische Kaskade im Wurmdarm nach dem Tod ausgelöstwird 31.
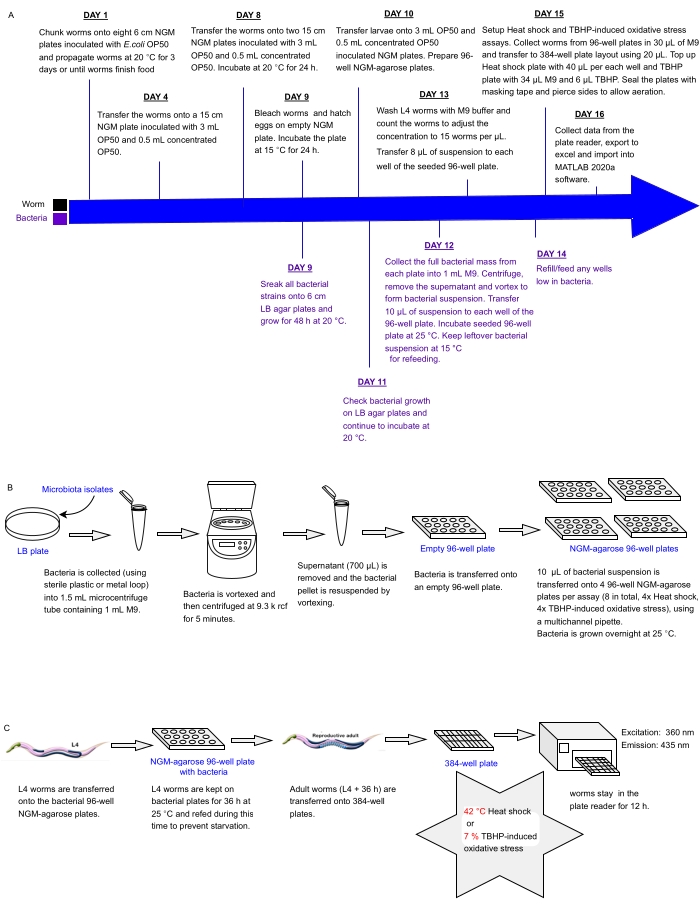
Abbildung 1: Experimenteller Workflow für das Hochdurchsatz-Screening von Bakterienisolaten mit Auswirkungen auf die Stressresistenz von C. elegans . (A) Zeitplan für die Erhaltung von Würmern und Bakterien und den Aufbau des Assays. (B) 96-Well-Aufbau und Handhabung bakterieller Plattenarrays. (C) 384-Well-Schneckenplatten-Setup. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protocol
Representative Results
Discussion
C. elegans bietet aufgrund seiner geringen Größe, Transparenz, schnellen Entwicklung, kurzen Lebensdauer, kostengünstigen und einfachen Handhabung viele Vorteile für das schnelle Screening mehrerer experimenteller Parameter auf einmal. Sein wesentlich einfacheres Genom, Körperplan, Nervensystem, Darm und Mikrobiom, aber komplex und ähnlich genug wie der Mensch, machen es zu einem leistungsstarken präklinischen Modell, in dem mechanistische Erkenntnisse gewonnen werden können, während bioaktive Wirksamke…
Divulgaciones
The authors have nothing to disclose.
Acknowledgements
Wir danken dem CGC Minnesota (Madison, USA, NIH – P40 OD010440) für die Bereitstellung von Wurmstämmen und OP50 und Prof. Hinrich Schulenburg (CAU, Kiel, Deutschland) für die Bereitstellung aller hier abgebildeten Umweltmikrobiellen Isolate. Diese Arbeit wurde durch einen UKRI-BBSRC-Zuschuss an AB finanziert (BB/S017127/1). JM wird durch ein FHM-PhD-Stipendium der Lancaster University finanziert.
Materials
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
Referencias
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood – Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977 (2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer’s disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98 (2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson’s disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921 (2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095 (2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323 (2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio – The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364 (2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545 (2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849 (2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998 (2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613 (2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer’s disease: a systematic review. Nutrients. 14 (1), 20 (2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson’s disease. Ageing Research Reviews. 69, 101347 (2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -. E. H., Jung, Y., Lee, S. -. J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190 (2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583 (2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398 (2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448 (2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35 (2016).

