RNA Secondary Structure Prediction Mit High-Throughput-SHAPE
Summary
Hochdurchsatz-selektiven 2-Hydroxyl-Acylierung durch Primer-Extension (FORM) analysiert verwendet einen neuartigen chemischen Tastertechnologie, reverse Transkription, Kapillarelektrophorese und Sekundärstrukturvorhersage Software, um die Strukturen der RNAs, die von mehreren hundert bis zu mehreren tausend bestimmen Nukleotide an Einzel-Nukleotid-Auflösung.
Abstract
Das Verständnis der Funktion der RNA in biologischen Prozessen beteiligt erfordert eine gründliche Kenntnis der RNA-Struktur. Zu diesem Zweck wird die Methodik genannt "High-Throughput-selektive 2-Hydroxyl Acylierung durch Primer-Extension analysiert", oder SHAPE ermöglicht Vorhersage von RNA Sekundärstruktur mit Einzel-Nukleotid-Auflösung. Dieser Ansatz nutzt chemische Sondierung Mittel, die Acylierung bevorzugt einzelsträngig oder flexiblen Bereiche der RNA in wässriger Lösung. Seiten der chemischen Modifizierung durch reverse Transkription der RNA modifiziert detektiert und die Produkte dieser Reaktion sind fraktioniert automatisierten Kapillarelektrophorese (CE). Seit Reverse Transkriptase Pausen an diesen RNA Nukleotide durch die Form Reagenzien modifiziert, die resultierende cDNA-Bibliothek indirekt abbildet diese Ribonukleotiden die einzigen im Rahmen der gefalteten RNA gestrandet sind. Mit ShapeFinder Software werden die Elektropherogrammen durch automatisierte CE produziert verarbeitet und in nugonucleotid Reaktivität Tabellen, die sich in Pseudo-Energie-Einschränkungen in der RNAStructure (v5.3) Vorhersage-Algorithmus verwendet wird, umgewandelt. Die zweidimensionalen RNA-Strukturen durch die Kombination FORM Sondieren mit in silico RNA Sekundärstrukturvorhersage erhalten haben, wurde festgestellt, dass viel genauer als Strukturen erhalten mit beiden Methoden allein.
Introduction
Um die Funktionen der katalytischen und nicht-kodierende RNAs bei der Regulation der Spleißen, Übersetzung, Virusreplikation und Krebs beteiligt zu verstehen, ist eine genaue Kenntnis der RNA-Struktur 1,2 erforderlich. Leider stellt genaue Vorhersage der RNA-Faltung eine gewaltige Herausforderung. Klassische Sondieren Mittel leiden viele Nachteile, wie Toxizität, unvollständige Nucleotidsequenz Abdeckung und / oder den Durchsatz beschränkt auf 100-150 Nukleotide pro Versuch. Unaided Sekundärstruktur Vorhersage-Algorithmen sind ähnlich nachteilig wegen Ungenauigkeiten aufgrund ihrer Unfähigkeit, effektiv unter energetisch ähnlichen Strukturen zu unterscheiden. Große RNAs insbesondere auch oft feuerfesten Verfahren zur 3D-Struktur Bestimmung wie Röntgenkristallographie und NMR-Spektroskopie (NMR), die aufgrund ihrer konformativen Flexibilität und große Mengen an hochreinem Proben für diese Techniken erforderlich.
HIGH-Durchsatz SHAPE löst viele dieser Probleme durch eine effektive, einfache Methode, um die Erforschung der Strukturen von großen RNAs bei single-nucleotide Auflösung. Darüber hinaus sind die Reagenzien für SHAPE verwendet sichere, einfach zu handhaben und im Gegensatz zu den meisten anderen chemischen Reagenzien Sondieren, mit allen vier Ribonukleotiden reagieren. Diese Reagenzien können auch zelluläre Membranen eindringen, was es ermöglicht, RNAs in ihre in vivo-Kontext (e) 3 sondieren. Ursprünglich in den Wochen 4 Labor entwickelt wurde, hat SHAPE verwendet worden, um eine Vielzahl von RNAs, die bemerkenswerteste Beispiel ist die Bestimmung der vollständigen sekundären Struktur des ~ 9 kb HIV-1-RNA-Genom 5 zu analysieren. Andere bemerkenswerte Erfolge mit SHAPE umfassen Aufklärung der Strukturen von infektiösen Viroiden 6, menschlichen langen nicht-kodierenden RNAs 7, Hefe Ribosomen 8 und 9 riboswitches sowie zur Identifizierung von Protein-Bindungsstellen in Virion-assoziierten HIV-1-RNA-3. While das Original und High-Throughput-Variationen des SHAPE-Protokoll wurde bereits an anderer Stelle veröffentlicht 10-12, stellt die vorliegende Arbeit eine detaillierte Beschreibung der RNA Sekundärstruktur Bestimmung durch High-Throughput-SHAPE mit fluoreszierenden Oligonukleotiden, die Beckman Coulter CEQ 8000 Genetic Analyzer, und ShapeFinder und RNAStructure (v5.3)-Software. Bisher unveröffentlichte technischen Details und Hinweise zur Fehlersuche sind ebenfalls enthalten.
Variationen von SHAPE
Das Wesen Form und seinen Varianten ist die Exposition von RNA in wässriger Lösung einer elektrophilen Anhydride, die selektiv acyliert 2'-Hydroxy-(2'-OH)-Gruppen Ribose, wodurch sperrige Addukte an den Stellen der Modifikation. Diese chemische Reaktion dient als Mittel zum Abfragen lokalen RNA strukturelle Dynamik, als einzelsträngige Nukleotide anfälliger für Konformationen förderlich elektrophilen Angriff durch diese Reagenzien erlassen sind, während basengepaarte oder architektonisch bebauteained Nukleotide sind weniger reaktiv oder 10. Seiten der Adduktbildung werden durch reverse Transkription initiiert von fluoreszent oder radioaktiv markierten Primer hybridisiert an eine spezifische Stelle auf der modifizierten RNA (die "(+)" Primer-Extension-Reaktion) detektiert. Als reverse Transkriptase (RT), um die acylierten Ribonukleotide durchqueren fehlschlägt, wird ein Pool von cDNA-Produkte hergestellt werden, deren Längen übereinstimmen mit Seiten der Modifikation. Ein Steuersystem, "(-)" Primerverlängerungsreaktion Verwendung RNA, die nicht Reagenz ausgesetzt wurde auch durchgeführt, so dass eine vorzeitige Beendigung der DNA-Synthese (dh "Haltestellen") aufgrund von Struktur, unspezifische RNA-Strang Bruch etc, kann RNA. Pausieren von durch chemische Modifikation hergestellt wurden, unterschieden werden. Schließlich werden zwei Dideoxy-Sequenzierungsreaktionen Einleiten von denselben Primern als Marker verwendet werden, um reaktive Nukleotide mit dem RNA-Primärsequenz nach Elektrophorese korrelieren.
In der ursprünglichen Anwendung SHAP(-), Und zwei Sequenzierungsreaktionen E, gleich 32 P-end-markiertem Primer für die (+), verwendet. Produkte dieser Reaktionen in benachbarten Vertiefungen in einer 5-8% igen Polyacrylamid-Slab-Gel geladen und fraktioniert durch denaturierende Polyacrylamid-Gelelektrophorese (PAGE; Abbildung 1). Die quantitative Analyse der Gel-Bilder durch herkömmliche Form hergestellt werden unter Verwendung SAFA, ein halbautomatisches Footprint-Analyse-Software 13 ist.
Im Gegensatz dazu verwendet Hochdurchsatz-FORM fluoreszenzmarkierten Primer und automatisierten Kapillarelektrophorese. Genauer gesagt, für jede Region von RNA untersucht, ein Satz von vier DNA Primer eine gemeinsame Sequenz, aber unterschiedliche 5'Fluoreszenzmarkierungen müssen synthetisiert oder gekauft werden. Diese unterschiedlich markierten Oligonukleotide dienen prime zwei SHAPE Reaktionen und zwei Sequenzierungsreaktionen, deren Produkte gebündelt und fraktionierte / erkannt durch automatisierte Kapillarelektrophorese (CE). Whereas die Reaktivität Profil von 100-150 nt von RNA aus einem Satz von vier Reaktionen mit dem ursprünglichen Ansatz erreicht werden kann, ermöglicht Hochdurchsatz-SHAPE Auflösung von 300-600 nt aus einer gepoolten Probe 3. Bis zu 8 Sätze von Reaktionen fraktioniert werden gleichzeitig, jedoch nicht weniger als 96 Proben für die Fraktionierung innerhalb von 12 aufeinander folgenden Läufen CE (2) hergestellt. Darüber hinaus ist die ShapeFinder entwickelte Software zur Verarbeitung und Analyse von Daten, die aus dem CEQ und andere genetische Analysatoren, stärker automatisiert und erfordern viel weniger Benutzereingriff als SAFA 13 oder Gel-Analyse-Pakete.
Weiter fortgeschrittene Hochdurchsatz-Methodik wurden kürzlich wie PARS (parallele Analyse von RNA-Struktur) 14 und Frag-Seq (Fragment-Sequenzierung) 15, die Struktur-spezifische Enzyme anstatt Alkylierungsmittel Verwendung in Verbindung mit der nächsten Generation Sequenzierung Techniken zu erhalten entstanden information über RNA-Struktur. Trotz der Attraktivität dieser Techniken, die viele Einschränkungen inhärent Nuklease Sondieren noch 16 bleiben. Diese Probleme können in Form Sequenzierung (Shape-Seq) 17-Protokoll, wobei Sequenzierung der nächsten Generation durch chemische Modifizierung und reverse Transkription der RNA in einer ähnlichen Weise wie in herkömmliche Form durchgeführt vorausgeht umgangen werden. Obwohl diese Methoden die Zukunft der RNA-Struktur Bestimmung darstellen kann, ist es wichtig, sich daran zu erinnern, dass der nächsten Generation Sequenzierung sehr teuer ist und bleibt für viele Labore.
SHAPE Data Analysis
Die Daten in der Genetic Analyzer erzeugt wird in Form einer Elektropherogramm dargestellt, wobei die Fluoreszenzintensität der Probe (n), der durch die Kapillare Detektor wird gegen einen anderen Zeitpunkt aufgetragen. Dieses Grundstück hat die Form von überlappenden Spuren entsprechend den vier Fluoreszenzkanals verwendet, um die verschiedenen Fluorophoren zu erfassen, und wobei jede Spur aus einzelnen Peaks, die cDNA oder Sequenzierungsprodukte besteht. Elektropherogramm Daten aus dem Genetic Analyzer als Tab-getrennte Textdatei exportiert und importiert in ShapeFinder Transformation und Analyse-Software 18.
ShapeFinder zunächst verwendet, um eine Reihe mathematischer Transformationen an den Daten durchführen, um sicherzustellen, dass die Migration Zeiten und Peakvolumina genau die Identität und Menge der Reaktionsprodukte sind. Peaks werden dann ausgerichtet und integriert, und die Ergebnisse zusammen mit dem primären RNA-Sequenz aufgelistet. A "Reaktivitätsprofil" für den betreffenden Abschnitt des RNA wird durch Subtrahieren Steuerwerte aus dem (+) RNA, die mit jedem Nukleotid verbunden ist, und das Normalisieren der Daten, wie unten beschrieben erhalten. Dieses Profil wird in RNAstructure importiert (v5.3)-Software 19,20, die den normierten Reaktivität val wandeltUES in Pseudo-Energie Einschränkungen, die in der RNA-Sekundärstruktur Faltungsalgorithmus eingebaut sind. Das Kombinieren der chemischen Sondieren und Falten Algorithmen auf diese Weise erheblich verbessert die Genauigkeit der Vorhersage Struktur im Vergleich zu beiden Verfahren allein 12,21. Der Ausgang des RNAstructure (v5.3) enthält Bilder von der niedrigsten Energie RNA Sekundärstrukturen mit der Form Reaktivitätsprofil (en), sowie den gleichen Strukturen in textueller dot-Klammern zu farbcodiert. Letzteres kann anschließend auf Software spezialisiert auf die grafische Darstellung der RNA Sekundärstruktur wie Varna 22 und 23 PseudoViewer exportiert werden.
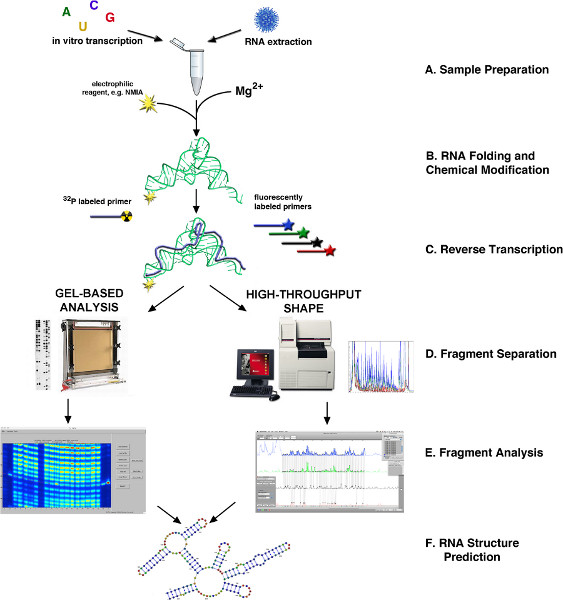
Abbildung 1. Flussdiagramm der RNA-Struktur Bestimmung über SHAPE 4,10. (A) RNA may aus biologischen Proben oder durch in vitro-Transkription erhalten werden. (B) Je nach Quelle wird RNA gefaltet oder anderweitig verarbeitet und modifiziert mit SHAPE Reagenz. (C) reverse Transkription fluoreszierend oder radioaktiv markierte Primer verwenden. (D) cDNA-Produkte sind fraktionierten entweder über Kapillare oder Slab-Gel-basierten Elektrophorese. (E) Fragment-Analyse. (F) RNA-Struktur-Vorhersage. Klicken Sie hier für eine größere Abbildung zu sehen.
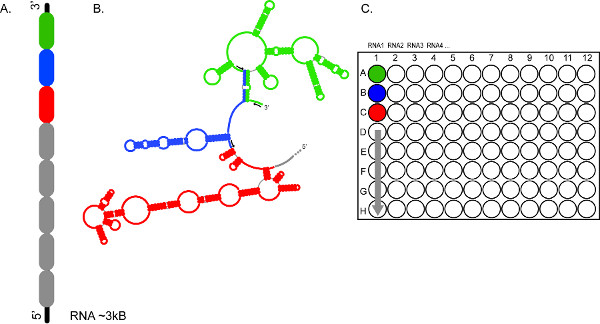
Abbildung 2. Die Hochdurchsatz-Charakter der CE-Form ermöglicht eine schnelle Analyse mehrerer RNAs, und / oder mehrere Segmente der gleichen RNA. (A) </stRong> Gibt an, wie ein RNA in 300-600 nt Abschnitte (Farbe in grün, blau und rot kodiert) (B) Sektionen der RNA unabhängig sondiert mit verschiedenen Sätzen von fluoreszierenden Primer (schwarze Pfeile) (C) Sets unterteilt werden kann Reaktionen werden vereinigt und in die Vertiefungen A1, B1, C1, etc., die jeweils geladen, wodurch eine vollständige Abdeckung des ~ 3 kb RNA1. Reaktionsprodukte von RNAs 2, 3, 4, usw. können in ähnlicher Weise für die Fraktionierung in aufeinanderfolgenden Läufen Elektrophorese vorbereitet werden. Klicken Sie hier, um eine größere Abbildung anzuzeigen.
Protocol
Representative Results
Discussion
Wir stellen Ihnen hier eine detaillierte Protokoll für High-Throughput-SHAPE, eine Technik, die sekundäre Struktur Bestimmung erlaubt single-nucleotide Auflösung für RNAs jeder Größe. Außerdem Kopplung experimentellen Daten mit SHAPE Sekundärstruktur Vorhersage-Algorithmen ermöglicht Generation von RNA 2D-Modelle mit einem höheren Grad an Genauigkeit, als es mit beiden Methoden allein. Die Kombination von fluoreszenzmarkierten Primern und automatisierte CE bietet erhebliche Vorteile gegenüber dem herkömmlich…
Divulgations
The authors have nothing to disclose.
Acknowledgements
S. Lusvarghi, J. Sztuba-Solinska, KJ Purzycka, JW Rausch und SFJ Le Grice werden durch die Interne Research Program des National Cancer Institute, National Institutes of Health, USA unterstützt.
Materials
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5′ labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied – including any 3’terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies |
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 ‘-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 ‘-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2′-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues – A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).

