使用高通量形状的RNA二级结构预测
Summary
的高通量选择性2'羟基的酰化反应分析的引物延伸(SHAPE)利用了一种新的化学探测技术,反转录,毛细管电泳确定结构的RNA二级结构预测软件从几百到几千个核苷酸,单核苷酸分辨率。
Abstract
了解RNA的功能涉及生物过程,需要全面了解的RNA结构。为此,该方法被称为“引物延伸”,或形状分析的高通量选择性的2'羟基的酰化,允许预测RNA二级结构的单核苷酸分辨率。这种方法利用化学探测剂,优先酰化的RNA在水溶液中的单链或柔性区域。化学修饰的位点被检测到的修饰的RNA的反转录,并在此反应中的产品是通过自动毛细管电泳(CE)分馏。由于修改后由SHAPE试剂在这些RNA的核苷酸逆转录酶暂停,由此产生的cDNA文库间接映射的上下文中的折叠的RNA的核糖核苷酸的单链。使用的电泳软件ShapeFinder,产生的自动化CE的处理和转换成菜单核苷酸反应表转换成伪能量约束在RNAStructure(V5.3)预测算法。二维形状探测在硅片 RNA二级结构预测相结合所获得的RNA结构被认为是比单独使用这两种方法得到的结构更准确。
Introduction
要了解催化和非编码RNA的功能,参与调控拼接,翻译,病毒的复制和癌症,RNA结构的详细知识是必需的1,2。不幸的是,准确预测RNA折叠提出了一个严峻的挑战。古典探测剂遭受许多缺点,如毒性,不完整覆盖核苷酸和/或每100-150个核苷酸实验吞吐量有限。肉眼二级结构预测算法同样是不利的,因为不准确,致使他们无法有效区分大力相似的结构。特别大的RNA也常耐火材料,如X-射线晶体学和核磁共振(NMR)谱,由于它们的构象灵活性和大量的这些技术所需的高纯度的样品的三维结构的测定方法。
ħIGH-吞吐量形状可以解决许多这些问题提供一个有效的,简单的方法来探测的大RNA的结构,单核苷酸分辨率。此外,该试剂用于形状是安全的,易于处理,在大多数其他化学探测试剂相比,所有四个核糖反应。这些试剂也可以穿透细胞膜,使得有可能探测的RNA在其体内上下文(次)3。最初4周实验室开发的,形状已经用于分析各种各样的RNA〜9 KB HIV-1 RNA基因组的完整的二级结构的测定,最显着的例子。其他显着的成果,使用形状感染类病毒6,人长非编码RNA 7,酵母核糖8,9和核糖开关以及确定蛋白质结合位点的病毒体相关的HIV-1 RNA的结构,包括澄清。瓦其他地方10-12法兰西岛大区的原件和高通量的变化形状协议已经公布,目前的工作提供了详细的说明,通过高通量的RNA二级结构的测定采用荧光寡核苷酸,贝克曼CEQ 8000遗传分析仪, SHAPEfinder RNAStructure(V5.3)软件。先前未公开的技术细节和故障排除建议也包括在内。
形状变化
的本质的外型及其变化是暴露的RNA在水溶液中电酸酐选择性酰化2'-羟基(2'-OH)的核糖基生产笨重的加成物在网站修改。这种化学反应作为一种手段,询问当地的RNA结构动力学,单链核苷酸更容易采取有利于电攻击这些试剂的构象,而碱基配对或建筑构造ained核苷酸少或不反应10。加合物的形成的位点被检测到通过反转录启动从荧光或放射性标记的引物杂交到特定站点上的修饰的RNA(“(+)”引物延伸反应)。当逆转录酶(RT)失败遍历酰化核糖核苷酸cDNA的产品生产,游泳池,其长度,配合网站修改。一控“( – )”引扩展利用RNA尚未暴露试剂的反应也表现( 即 “停止”)由于DNA合成RNA结构,非特异性的RNA链断裂, 等等 ,可提前终止通过化学修饰暂时停止生产加以区分。最后,两个二脱氧测序反应,启动从相同的引物被用来作为标记,电泳后的RNA一级序列相关联反应的核苷酸。
在原来的应用SHAPE,在相同的32个P-末端标记的引物被用于(+),( – ),和两个测序反应。这些反应的产品装入相邻的井,在5%至8%的聚丙烯酰胺平板凝胶,并通过变性聚丙烯酰胺凝胶电泳(PAGE, 图1)分馏。可以执行常规形状所产生的凝胶图像的定量分析,使用SAFA,半自动化的足迹分析软件13。
与此相反,高通量的形状采用荧光标记的引物和自动化的毛细管电泳。具体而言,对每个区域的RNA的调查中,一组四个的DNA引物,具有一个共同的序列,但不同的5'端荧光标记必须被合成或购买。这些不同的标记的寡核苷酸素两个形状的反应和两个测序反应,汇集和分离/检测自动毛细管电泳(CE)产品服务。到哪里去EAS 100-150个核苷酸的RNA的反应档案可从一组四个反应,使用原始的方法,高通量的形状允许从单一样本3的分辨率为300-600个核苷酸。最多8台反应同时进行分馏,而多达96个样品可以在连续12个CE运行( 图2)的过程中分馏制备。此外,的SHAPEfinder软件,开发新兴CEQ和其他遗传分析仪的数据处理和分析,更加自动化,需要用户干预的少得多,比SAFA 13或其他凝胶分析软件包。
最近出现了更先进的高通量方法如PARS(平行的RNA结构分析)14和片段序列(片段测序)15,它使用结构特异性的酶,而不是配合新一代测序技术获得烷基化试剂的资n关于RNA的结构。尽管这些技术的吸引力,核酸探测的许多固有的局限性仍然16。这些问题是可以规避在SHAPE的测序(形状序列)17的协议,进行常规形状类似的方式通过化学修饰的RNA的反转录前面下一代测序。虽然这些方法可能代表了未来的RNA结构测定,重要的是要记住,下一代测序是非常昂贵的,并且仍然无法许多实验室。
形状数据分析
在遗传分析仪产生的数据的形式的电泳,其特征在于,所述的样品的荧光强度(次)流经毛细管探测器的对一个索引的迁移时间作图。此图的形式重叠痕迹对应于四个荧光通道用来在不同检测不同的荧光基团,其中每个轨迹是由对应于个体的cDNA或测序反应产物的峰。电泳数据是从遗传分析仪为制表符分隔的文本文件导出和导入ShapeFinder转换和分析软件18。
ShapeFinder最初用于执行一系列的数据的数学变换,以确保准确地反映迁移时间和峰卷的身份和数量的反应产物分别。峰,然后对准和集成,并与主要的RNA序列的结果列在一起。减去控制值的(+)的值与每个RNA核苷酸,和归一化数据(如下文所述)得到的RNA相关的段A“反应性配置文件”。此配置文件导入到软件19,20 RNAstructure(V5.3),归一化反应的val转换UES成伪能量纳入RNA二级结构折叠算法的约束。配合化学探测和折叠的算法以这种方式,显着地提高了单独12,21的任一方法相比,结构预测的准确度。输出的RNAstructure(V5.3)包括最低能量的图像颜色编码的形状反应曲线(S),以及文字圆点括号表示法中的相同结构的RNA二级结构。随后,后者可被导出RNA二级结构(如瓦尔纳22 PseudoViewer 23)的图形显示的专用软件。
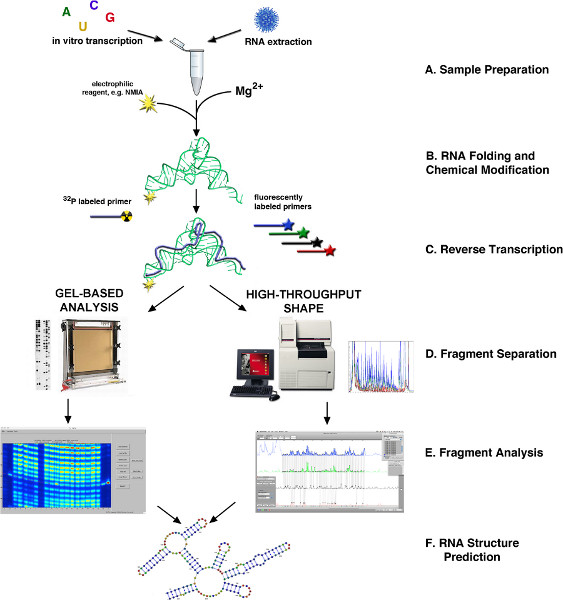
图1。 (A)+ RNA米流程图形状4,10 RNA结构测定通过。获得(B)根据讯号源,RNA折叠或以其他方式处理和修改形状试剂(C)反转录使用荧光或放射性标记的引物(D)cDNA产物是从生物样品或通过体外转录。分馏通过毛细管或平板凝胶电泳(E)片段分析(F)RNA结构预测点击这里查看大图。
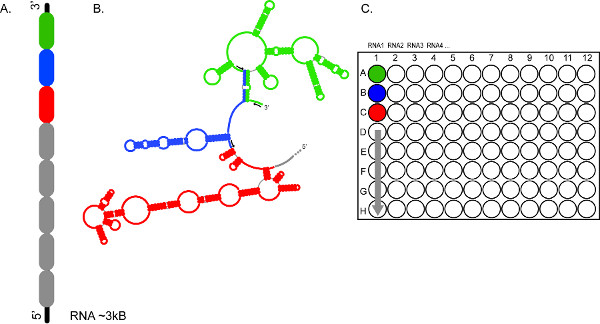
图2。 (A)基于CE的形状允许高吞吐量的字符的多个RNA的快速分析,和/或相同的RNA的多个段。 </st荣>表示的RNA可以被划分为300-600核苷酸部分(编码的颜色为绿色,蓝色和红色),(B)的被探测的RNA部分独立地使用不同组的荧光引物(黑色箭头)(C)设置反应池井A1,B1,C1 等 ,分别装入,提供完整的覆盖〜3 kb的基因组RNA1。的RNA 2,3,4,等反应产物可能会同样分馏连续电泳奔跑的准备。 点击这里查看大图。
Protocol
Representative Results
Discussion
我们在座的详细协议为高吞吐量形状,一种技术,使任何规模的RNA的单核苷酸分辨率为二级结构的测定。此外,耦合形状实验数据与二级结构预测算法有利于RNA二维模型生成具有更高的准确度可能比单独的任一方法。荧光标记的引物和自动CE的结合,提供了显着优于传统的基于凝胶的形状在一次实验中,有利于解决长的RNA序列,以及相当高的速度和通过多个实验。权宜之计此方法,并提供合适的?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
院内研究计划的美国国家癌症研究所,美国国立卫生研究院,美国,JW,KJ Purzycka的J. Sztuba Solinska S. Lusvarghi,劳施和SFJ格莱斯支持。
Materials
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5′ labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied – including any 3’terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies |
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 ‘-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 ‘-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2′-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues – A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).

