חיזוי מבנה המשני RNA באמצעות SHAPE תפוקה גבוהה
Summary
acylation הידרוקסיל '2 סלקטיבית תפוקה גבוהה נותח על ידי הארכה תחל (SHAPE) מנצל כימי רומן חיטוט טכנולוגיה, שעתוק לאחור, אלקטרופורזה נימים ותוכנות לחיזוי מבנה משנית כדי לקבוע את המבנים של RNAs מכמה מאה עד כמה אלף נוקלאוטידים ברזולוציה נוקלאוטיד יחידה.
Abstract
הבנת תפקידו של רנ"א המעורב בתהליכים ביולוגיים דורשת ידע מעמיק של מבנה RNA. לשם כך, את המתודולוגיה המכונה "acylation הידרוקסיל 2 סלקטיבית תפוקה גבוהה 'נותח על ידי הארכה תחל", או צורה, מאפשרת חיזוי של מבנה המשני RNA עם רזולוציה נוקלאוטיד יחידה. גישה זו מנצלת סוכני חיטוט כימיים שמעדיפים acylate אזורים תקועים או גמישים בודדות של רנ"א בתמיסה מימית. אתרים של שינוי כימי מזוהים על ידי שעתוק הפוך של RNA השונה, ואת המוצרים של התגובה הזו הם מופרדים על ידי אלקטרופורזה נימים אוטומטית (CE). מאז רברס טרנסקריפטאז הפסקות באותם נוקלאוטידים RNA שונה על ידי חומרים כימיים צורה, ספריית cDNA כתוצאה בעקיפין ממפה האלה ribonucleotides שהם תקועים אחת בהקשר של RNA המקופל. באמצעות תוכנת ShapeFinder, המיוצר על ידי את electropherograms האוטומטית לספירה מעובד והוסב לנושולחנות תגובתיות cleotide שהם עצמם הפכו לאילוצים פסאודו אנרגיה בשימוש באלגוריתם חיזוי RNAStructure (V5.3). את מבני RNA דו ממדים המתקבלים על ידי שילוב של SHAPE חיטוט עם בחיזוי מבנה המשני סיליקון ורנ"א כבר נמצא להיות הרבה יותר מדויק מאשר מבנים המתקבלים בשני שיטות לבד.
Introduction
כדי להבין את הפונקציות של RNAs קטליטיים וללא קידוד מעורב בויסות של שחבור, תרגום, שכפול נגיף וסרטן, ידע מפורט של מבנה RNA נדרש 1,2. לרוע המזל, חיזוי מדויק של רנ"א מתקפל מציג אתגר עצום. סוכני חיטוט הקלסיים סובלים מחסרונות רבים כגון רעילות, כיסוי חלקי נוקליאוטידים ו / או תפוקה מוגבלת ל100-150 נוקלאוטידים לכל ניסוי. אלגוריתמים לחיזוי מבנה משניים בכוחות עצמו הם דומה נחות, בשל אי דיוקים הנובעים מחוסר היכולת שלהם להבחין ביעילות בין מבנים אנרגטי דומים. RNAs גדול בפרט הם לעתים קרובות גם עקשן לשיטות של קביעת מבנה 3D כגון קריסטלוגרפיה רנטגן ותהודה מגנטית גרעינית (NMR) ספקטרוסקופיה, בשל הגמישות שלהם קונפורמציה וכמויות גדולות של דגימות טהורות ביותר הנדרשות לטכניקות אלה.
HSHAPE igh תפוקה פותר רבים מהבעיות הללו על ידי מתן גישה יעילה, פשוטה לחיטוט המבנים של RNAs גדול ברזולוציה נוקלאוטיד יחידה. יתר על כן, את חומרים כימיים המשמשים עבור צורה בטוח, קל לטפל ו, בניגוד לרוב כימי אחרות חיטוט ריאגנטים, מגיבים עם כל ארבע ribonucleotides. ריאגנטים אלה יכולים גם לחדור ממברנות תאים, מה שמאפשר לחקור RNAs בהם בהקשר vivo (ים) 3. פותח במקור בשבועות המעבדה 4, צורה נעשתה שימוש כדי לנתח מגוון רחב של RNAs, הדוגמא הבולטת ביותר היא קביעת המבנה המשני המלא של ~ 9 KB HIV-1 RNA הגנום 5. הישגים בולטים אחרים בשימוש בצורה כוללים הבהרה של המבנים של viroids זיהומיות 6, RNAs אדם הארוך ללא קידוד 7, שמרי ריבוזומים 8, וriboswitches 9, כמו גם לזהות אתרי קישור בחלבון virion הקשורים RNA HIV-1 3. מה עלי לעשותאיל הווריאציות המקוריות ותפוקה גבוהה של פרוטוקול SHAPE כבר פורסם במקומות אחרים 10-12, העבודה הנוכחית מספקת תיאור מפורט של קביעת מבנה המשנית RNA על ידי צורת תפוקה גבוהה באמצעות oligonucleotides ניאון, מנתח Beckman Coulter CEQ 8000 הגנטי, ו SHAPEfinder וRNAStructure תוכנה (V5.3). פרטים טכניים שלא פורסמו בעבר ועצות לפתרון בעיות כלולות גם.
וריאציות של צורה
המהות של צורה והווריאציות שלה היא חשיפה של רנ"א בתמיסה מימית לפטאליק electrophilic כי סלקטיבי acylate קבוצות (2'-OH) 2'-הידרוקסיל ריבוז, ייצור adducts המגושם באתרים של שינוי. תגובה כימית זו משמשת כאמצעי לחקירת דינמיקה מבנית מקומית RNA, נוקלאוטידים כיחידים תקועים נוטים יותר לאמץ תצורות תורמות להתקפת electrophilic על ידי חומרים כימיים האלה, ואילו בסיס לזווג או ארכיטקטוני constrנוקלאוטידים ained הם פחות או unreactive 10. אתרים של היווצרות adduct מזוהים על ידי שעתוק לאחור ייזום מfluorescently או radiolabeled פריימרים הכלאה לאתר מסוים ברנ"א השונה ("(+)" תגובת ההארכה תחל). כאשר רברס טרנסקריפטאז (RT) לא מצליח לחצות את ribonucleotides acylated, בריכת של מוצרים מיוצרת cDNA שאורכי קנה אחד עם אתרים של שינוי. שליטה, "(-)" פריימר סיומת תגובת ניצול RNA שלא נחשף למגיב מבוצעת גם כדי שהסיום המוקדם של סינתזת דנ"א ("תחנות", כלומר) בשל מבנה RNA, שבר ספציפי RNA גדיל, וכו ', במאי. להבדיל ממשתהה מיוצר על ידי שינוי כימי. לבסוף, שתי תגובות dideoxy-רצף ייזום מאותם פריימרים משמשות כסמנים כדי לקשר בין נוקלאוטידים תגובתי עם רצף הרנ"א הראשוני בעקבות אלקטרופורזה.
ביישום המקורי של ShapE, אותו 32-P-שכותרתו סוף פריימר מנוצל ל( +), – ושתי תגובות רצף (). מוצרים של תגובות אלה נטענים לתוך בארות סמוכות בג'ל לוח polyacrylamide 5-8%, ומופרדים על ידי denaturing ג'ל אלקטרופורזה polyacrylamide (דף; איור 1). ניתוח כמותי של דימויי ג'ל מיוצרים על ידי צורה קונבנציונלית יכול להתבצע באמצעות סאפא, תוכנת ניתוח footprinting חצי אוטומטית 13.
לעומת זאת, צורת תפוקה גבוהה מעסיקה פריימרים שכותרתו fluorescently ואלקטרופורזה נימים אוטומטיות. באופן ספציפי, לכל אזור של רנ"א תחת חקירה, סט של ארבעה פריימרים DNA בעלי רצף משותף אך שונה 5 'תוויות ניאון חייבת להיות מסונתזים או נרכשו. oligonucleotides שונה שכותרתו אלה משמשים לשתי תגובות SHAPE הממשלה ושתי תגובות רצף, את המוצרים של אשר אספו ומופרד / זוהו על ידי אלקטרופורזה נימים אוטומטית (CE). WherEAS ניתן להשיג את פרופיל תגובתיות של 100-150 NT של רנ"א מתוך סדרה של ארבע תגובות המשתמשות בגישה המקורית, צורת תפוקה גבוהה מאפשרת רזולוציה של 300-600 NT ממדגם 3 חד נקווה. עד 8 קבוצות של תגובות עשויים להיות מופרד בו זמנית, ואילו רבים כמו 96 דגימות יכולות מוכנות לחלוקה במהלך 12 ריצות לספירה רצופות (איור 2). יתר על כן, תוכנת SHAPEfinder, שפותחה כדי לעבד ולנתח נתונים העולים מתוך CEQ ומנתחים גנטיים אחרים, היא אוטומטית יותר ודורשת התערבות משתמש הרבה פחות מסאפא 13 או חבילות ג'ל ניתוח אחרות.
מתודולוגיות תפוקה גבוהה יותר מתקדמות שהתפתחו לאחרונה כגון PARS (ניתוח מקביל של מבנה RNA) 14 ו-Frag Seq (שבר-רצף) 15, המשתמש באנזימי מבנה ספציפיים ולא מגיבים לאלקילציה בשיתוף עם הדור הבא רצף טכניקות כדי להשיג information על מבנה RNA. למרות האטרקטיביות של טכניקות אלה, המגבלות רבות הגלומות לnuclease חיטוט עדיין יישארו 16. בעיות אלה ניתן לעקוף ברצף הצורה (SHAPE-Seq) 17 פרוטוקול, שבו הרצף של הדור הבא הוא קדמה שינוי כימי ושעתוק לאחור של RNAs באופן דומה לזה שבוצע בצורה קונבנציונלית. בעוד שיטות אלה עשויות לייצג את העתיד של קביעת מבנה RNA, חשוב לזכור שהרצף של הדור הבא הוא יקר מאוד, ונשאר זמין למעבדות רבות.
ניתוח נתוני צורה
נתונים שהופקו במנתח הגנטי מוצגים בצורה של electropherogram, שבו עוצמת הקרינה של הדוגמא (ו) זורמת דרך גלאי הנימים זממו נגד מדד של זמן הגירה. עלילה זו לובשת הצורה של עקבות חופפות המקבילה לערוץ הקרינה ארבעהים משמש כדי לזהות את fluorophores השונים, ובו כל עקבות מורכבת מפסגות מתאימות למוצרי cDNA או רצף בודדים. נתוני electropherogram מיוצאים מהמנתח הגנטי כקובץ טקסט מופרד באמצעות טאבים ויובאו לשינוי ShapeFinder וניתוח תוכנה 18.
ShapeFinder משמש בתחילה לבצע סדרה של טרנספורמציות מתמטיות על הנתונים כדי לוודא שפעמי הגירה והיקפי שיא משקפים במדויק את זהותם וכמויות של מוצרי התגובה, בהתאמה. פסגות הן אז מיושרות ומשולבים, ואת התוצאות נספרו יחד עם רצף הרנ"א הראשוני. "פרופיל תגובתיות" למקטע הרלוונטי של רנ"א מתקבל על ידי החסרת ערכי בקרה מ( +) ערכים הקשורים לכל רנ"א נוקלאוטיד, ונרמול הנתונים כפי שיתואר להלן. פרופיל זה מיובא לRNAstructure (V5.3) 19,20 תוכנה, אשר ממירה val תגובתיות המנורמלues תוך אילוצים פסאודו אנרגיה שמשולבים באלגוריתם קיפול המבנה המשני RNA. שילוב כימי חיטוט וקיפול אלגוריתמים בדרך זו משפר באופן משמעותי את הדיוק של חיזוי מבנה בהשוואה לכל אחת מהשיטות לבד 12,21. הפלט של RNAstructure (V5.3) כולל תמונות של האנרגיה הנמוכה ביותר RNA מבנים משניים בצבעים עם צורת פרופיל תגובתיות (ים), כמו גם את אותם מבנים בסימון הדוט סוגר טקסטואלי. זו האחרונה עשויה להיות מיוצאות לתוכנה המיועדת לתצוגה הגרפית של מבנה המשני RNA כגון רנה 22 וPseudoViewer 23.
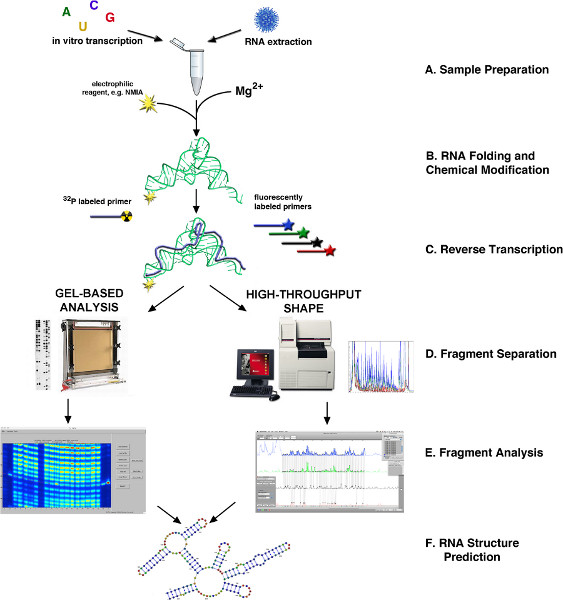
איור 1. תרשים זרימה של קביעת מבנה RNA באמצעות SHAPE 4,10. () RNA מ 'איי יתקבלו מדגימות ביולוגיות או על ידי שעתוק במבחנה. (ב) בהתאם למקור, RNA הוא מקופל או מעובד בדרך אחרת ושונים עם מגיב SHAPE. (ג) שעתוק הפוך באמצעות פריימרים שכותרתו fluorescently או רדיואקטיבית. (D) מוצרי cDNA הם מופרדים באמצעות שתי אלקטרופורזה מבוססת ג'ל נימים או לוח. (ה) ניתוח שבר. (F) ניבוי מבנה RNA. לחץ כאן לצפייה בדמות גדולה.
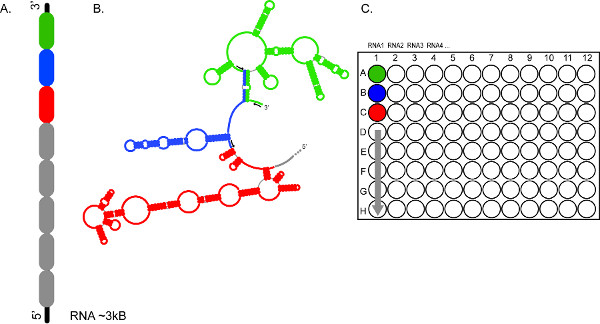
איור 2. אופי התפוקה גבוהה של צורה מבוססת CE מאפשר ניתוח מהיר של RNAs מרובים, ו / או מקטעים מרובים של אותם רנ"א. () </stרונג> מייצג כיצד ניתן לחלק RNA לסעיפי 300-600 NT (מקודד לפי צבע בירוק, כחול ואדום) (ב) חלקים של RNA הם חקרו באופן עצמאי באמצעות סטים שונים של פריימרים ניאון (חיצים שחורים) (ג) סטים תגובות הם אספו והעמיסו לתוך בארות A1, B1, C1, וכו ', בהתאמה, ומספקות כיסוי מלא ל~ 3 KB RNA1. תוצרי תגובה מRNAs 2, 3, 4, וכו 'יכול להיות מוכן באופן דומה לחלוקה בריצות electrophoretic רצופות. לחץ כאן לצפייה בדמות גדולה.
Protocol
Representative Results
Discussion
אנו מציגים כאן פרוטוקול מפורט עבור צורה של תפוקה גבוהה, טכניקה המאפשרת קביעת מבנה משנית לרזולוצית נוקלאוטיד יחידה לRNAs בכל גודל. יתר על כן, צימוד נתוני צורה ניסיונית עם אלגוריתמים לחיזוי מבנה משניים מאפשר יצירת המודלים 2D RNA עם רמה גבוהה יותר של דיוק מאשר אפשרי עם כל אח?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
ס Lusvarghi, ג'Sztuba-סולינסקי, KJ Purzycka, ג"ו ראוש וSFJ Le גרייס נתמך על ידי תכנית המחקר העירונית של המכון הלאומי לסרטן, המכונים הלאומי לבריאות, ארה"ב.
Materials
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5′ labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied – including any 3’terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies |
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 ‘-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 ‘-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2′-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues – A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).

