उच्च throughput के आकार का उपयोग करते हुए शाही सेना माध्यमिक संरचना भविष्यवाणी
Summary
प्राइमर एक्सटेंशन (आकार) द्वारा विश्लेषण उच्च throughput चयनात्मक 2 'हाइड्रॉक्सिल acylation एकल nucleotide प्रस्ताव पर कई हजार न्यूक्लियोटाइड करने के लिए कई सौ से RNAs के संरचनाओं निर्धारित करने के लिए एक प्रौद्योगिकी की जांच उपन्यास रासायनिक, रिवर्स प्रतिलेखन, केशिका वैद्युतकणसंचलन और माध्यमिक संरचना भविष्यवाणी सॉफ्टवेयर का इस्तेमाल करता है.
Abstract
जैविक प्रक्रियाओं में शामिल शाही सेना के समारोह को समझना शाही सेना संरचना का एक संपूर्ण ज्ञान की आवश्यकता है. यह अंत की ओर है, करार दिया पद्धति, या आकार "प्राइमर विस्तार से विश्लेषण उच्च throughput चयनात्मक 2 'हाइड्रॉक्सिल acylation", एकल nucleotide संकल्प के साथ शाही सेना माध्यमिक संरचना की भविष्यवाणी की अनुमति देता है. इस दृष्टिकोण को प्राथमिकता जलीय घोल में शाही सेना के एकल असहाय या लचीला क्षेत्रों acylate कि रासायनिक जांच कर रही एजेंटों का इस्तेमाल करता है. रासायनिक संशोधन की साइटें संशोधित शाही सेना की रिवर्स प्रतिलेखन ने पता लगाया है, और इस प्रतिक्रिया के उत्पादों स्वचालित केशिका वैद्युतकणसंचलन (सीई) द्वारा fractionated हैं. रिवर्स ट्रांसक्रिपटेस आकार अभिकर्मकों द्वारा संशोधित उन आरएनए न्यूक्लियोटाइड पर विराम देता है के बाद से, जिसके परिणामस्वरूप सीडीएनए पुस्तकालय परोक्ष रूप से ही जोड़ शाही सेना के संदर्भ में फंसे हैं कि उन ribonucleotides नक्शे. ShapeFinder सॉफ्टवेयर का उपयोग करना, स्वचालित CE द्वारा उत्पादित electropherograms संसाधित और परमाणु में तब्दील कर रहे हैंcleotide जेट तालिकाओं कि खुद को RNAStructure (v5.3) भविष्यवाणी एल्गोरिथ्म में इस्तेमाल छद्म ऊर्जा की कमी में परिवर्तित कर रहे हैं. सिलिको शाही सेना माध्यमिक संरचना भविष्यवाणी में साथ जांच कर रही आकार के संयोजन से प्राप्त दो आयामी शाही सेना संरचनाओं अकेले भी विधि का उपयोग कर प्राप्त संरचनाओं की तुलना में कहीं अधिक सटीक होना पाया गया है.
Introduction
Splicing है, अनुवाद, वायरस प्रतिकृति है और कैंसर के नियमन में शामिल उत्प्रेरक और गैर कोडन RNAs के कार्यों को समझने के लिए, शाही सेना संरचना का विस्तृत ज्ञान 1,2 आवश्यक है. दुर्भाग्य से, आरएनए तह की सटीक भविष्यवाणी एक दुर्जेय चुनौती प्रस्तुत करता है. शास्त्रीय जांच कर रही एजेंटों पीड़ित ऐसे विषाक्तता, अधूरा न्यूक्लियोटाइड कवरेज और / या प्रयोग के प्रति 100-150 न्यूक्लियोटाइड तक सीमित throughput के रूप में कई नुकसान से. बेबस माध्यमिक संरचना भविष्यवाणी एल्गोरिदम इसी तरह, हानिकर प्रभावी रूप से उर्जा की समान संरचना के बीच अंतर करने में अपनी असमर्थता से उत्पन्न अशुद्धियों के कारण कर रहे हैं. विशेष रूप से बड़े RNAs भी इस तरह के एक्स – रे क्रिस्टलोग्राफी और उनके गठनात्मक लचीलापन और इन तकनीकों के लिए आवश्यक अत्यधिक शुद्ध नमूनों की बड़ी मात्रा के कारण, स्पेक्ट्रोस्कोपी (एनएमआर) अनुनाद परमाणु चुंबकीय के रूप में 3 डी संरचना निर्धारण के तरीकों को अक्सर आग रोक रहे हैं.
एचigh थ्रूपुट आकार एकल nucleotide संकल्प में बड़ी RNAs के ढांचों की जांच के लिए एक प्रभावी, सरल दृष्टिकोण प्रदान करके इन समस्याओं के कई हल करती है. इसके अलावा, आकार के लिए इस्तेमाल किया अभिकर्मकों संभालने के लिए सुरक्षित, आसान कर रहे हैं और, अभिकर्मकों की जांच सबसे अन्य रसायन के विपरीत, सभी चार ribonucleotides के साथ प्रतिक्रिया. इन अभिकर्मकों भी यह संभव विवो संदर्भ (ओं) 3 में अपने में RNAs की जांच के लिए बना रही है, सेलुलर झिल्ली घुसना कर सकते हैं. मूलतः सप्ताह प्रयोगशाला 4 में विकसित, आकार RNAs के एक विस्तृत विविधता, ~ 9 केबी एचआईवी -1 आरएनए जीनोम 5 की पूरी माध्यमिक संरचना के निर्धारण किया जा रहा है सबसे उल्लेखनीय उदाहरण का विश्लेषण करने के लिए इस्तेमाल किया गया है. आकार का उपयोग करते हुए अन्य उल्लेखनीय उपलब्धियों संक्रामक viroids 6, मानव लंबे गैर कोडन RNAs 7, खमीर राइबोसोम 8, और riboswitches 9 के साथ ही पहचान के लिए विरिअन जुड़े एचआईवी -1 आरएनए 3 में प्रोटीन बाध्यकारी साइटों के ढांचे की व्याख्या शामिल हैं. कआकार प्रोटोकॉल के इले मूल और उच्च throughput विविधताओं कहीं 10-12 प्रकाशित किया गया है, वर्तमान कार्य फ्लोरोसेंट oligonucleotides, Beckman कल्टर CEQ 8000 जेनेटिक विश्लेषक का उपयोग उच्च throughput आकार से शाही सेना माध्यमिक संरचना निर्धारण का विस्तृत विवरण प्रदान करता है, और SHAPEfinder और RNAStructure (v5.3) सॉफ्टवेयर. पहले अप्रकाशित तकनीकी जानकारी और समस्या निवारण सलाह भी शामिल किए गए हैं.
आकार के बदलाव
आकार और अपनी विविधताओं का सार चुनिंदा संशोधन के स्थलों पर भारी adducts उत्पादन, 2'-हाइड्रॉक्सिल (2'-OH) राइबोज़ समूहों acylate कि इलेक्ट्रोफिलिक anhydrides को जलीय घोल में शाही सेना के संपर्क में है. आधार बनती या वास्तुकला कंस्ट्रक्शन जबकि इस रासायनिक प्रतिक्रिया, एकल असहाय न्यूक्लियोटाइड इन अभिकर्मकों द्वारा इलेक्ट्रोफिलिक हमले के लिए अनुकूल रचना को अपनाने की संभावना है, के रूप में स्थानीय आरएनए संरचनात्मक गतिशीलता पूछताछ के साधन के रूप में कार्य करता हैained न्यूक्लियोटाइड कम या 10 unreactive हैं. अभिवर्तन गठन की साइटें संशोधित आरएनए ("()" प्राइमर विस्तार प्रतिक्रिया) पर एक विशिष्ट साइट के लिए संकरित fluorescently या radiolabeled प्राइमरों से की शुरुआत रिवर्स प्रतिलेखन द्वारा पता चला रहे हैं. रिवर्स ट्रांसक्रिपटेस (आरटी) acylated ribonucleotides पार करने में विफल रहता है, सीडीएनए उत्पादों की एक पूल जिनकी लंबाई संशोधन की साइटों के साथ मेल खाना उत्पादन किया जाता है. एक नियंत्रण, "(-)" प्राइमर विस्तार अभिकर्मक को उजागर नहीं किया गया है कि शाही सेना के उपयोग की प्रतिक्रिया भी इसलिए किया जाता है कि संरचना, अविशिष्ट शाही सेना किनारा टूटना, आदि, मई आरएनए के कारण डीएनए संश्लेषण (यानी "बंद हो जाता है ') के समय से पहले समाप्त. रासायनिक संशोधन द्वारा उत्पादित pausing से प्रतिष्ठित किया. अंत में, एक ही प्राइमरों से की शुरुआत दो dideoxy अनुक्रमण प्रतिक्रियाओं आरएनए प्राथमिक अनुक्रम वैद्युतकणसंचलन निम्नलिखित के साथ प्रतिक्रियाशील न्यूक्लियोटाइड सहसंबंधी मार्कर के रूप में उपयोग किया जाता है.
Shap के मूल आवेदन में(-), और दो अनुक्रमण प्रतिक्रियाओं ई, वही 32 पी अंत लेबल प्राइमर (+) के लिए उपयोग किया जाता है. इन प्रतिक्रियाओं के उत्पाद एक 5-8% polyacrylamide स्लैब जेल में आसन्न कुओं में भरी हुई है, और जेल वैद्युतकणसंचलन (पृष्ठ, चित्रा 1) denaturing द्वारा fractionated रहे हैं. पारंपरिक आकार द्वारा उत्पादित जेल छवियों के मात्रात्मक विश्लेषण साफा, एक अर्द्ध स्वचालित footprinting विश्लेषण सॉफ्टवेयर 13 का उपयोग किया जा सकता है.
इसके विपरीत, उच्च throughput आकार fluorescently लेबल प्राइमरों और स्वचालित केशिका वैद्युतकणसंचलन कार्यरत हैं. विशेष रूप से, जांच के तहत शाही सेना, एक आम दृश्य है, लेकिन विभिन्न 5 'फ्लोरोसेंट लेबल संश्लेषित या खरीदा जाना चाहिए होने के चार डीएनए प्राइमर का एक सेट के प्रत्येक क्षेत्र के लिए. ये अलग लेबल oligonucleotides के प्रधानमंत्री दो आकार प्रतिक्रियाओं और दो अनुक्रमण प्रतिक्रियाओं, जमा और fractionated / स्वचालित केशिका वैद्युतकणसंचलन (सीई) से पता चला रहे हैं जो उत्पादों के लिए काम करते हैं. Wherईएएस शाही सेना के 100-150 NT के जेट प्रोफाइल मूल दृष्टिकोण का उपयोग कर चार प्रतिक्रियाओं का एक सेट से प्राप्त किया जा सकता है, उच्च throughput आकार एक भी जमा नमूना 3 300-600 NT के संकल्प की अनुमति देता है. के रूप में कई 96 के रूप में नमूने लगातार 12 सीई रन (चित्रा 2) के पाठ्यक्रम पर विभाजन के लिए तैयार कर सकते हैं, जबकि एक साथ fractionated हो सकता है ऊपर प्रतिक्रियाओं के 8 सेट करने के लिए. इसके अलावा, CEQ और अन्य आनुवंशिक analyzers से उभरते डेटा की प्रक्रिया और विश्लेषण करने के लिए विकसित SHAPEfinder सॉफ्टवेयर,, अधिक स्वचालित है और साफा 13 या अन्य जेल विश्लेषण संकुल की तुलना में बहुत कम उपयोगकर्ता हस्तक्षेप की आवश्यकता होती है.
अधिक उन्नत उच्च throughput के तरीके में हाल ही में इस तरह की प्राप्त करने के लिए तकनीक अनुक्रमण अगली पीढ़ी के साथ संयोजन के रूप में संरचना विशेष एंजाइमों के बजाय alkylation अभिकर्मकों का उपयोग करें जो PARS (आरएनए संरचना के समानांतर विश्लेषण) 14 और frag-Seq (टुकड़ा अनुक्रमण) 15, के रूप में उभरा है informatioशाही सेना संरचना के बारे में पता. इन तकनीकों के आकर्षण के बावजूद, जांच nuclease लिए निहित कई सीमाएं अभी भी 16 में रहते हैं. इन समस्याओं के लिए अगली पीढ़ी के अनुक्रमण पारंपरिक आकार में प्रदर्शन किया है कि एक समान तरीके से रासायनिक संशोधन और RNAs के रिवर्स प्रतिलेखन से पहले है जहां आकार अनुक्रमण (आकार Seq) 17 प्रोटोकॉल में circumvented किया जा सकता है. इन तरीकों में शाही सेना संरचना निर्धारण के भविष्य का प्रतिनिधित्व कर सकते हैं, यह अगली पीढ़ी के अनुक्रमण बहुत महंगा है, और कई प्रयोगशालाओं के लिए उपलब्ध नहीं रहता है कि यह याद रखना महत्वपूर्ण है.
आकार डेटा विश्लेषण
आनुवंशिक विश्लेषक में उत्पादित डेटा स्थानांतरण समय के एक सूचकांक के खिलाफ साजिश रची है केशिका डिटेक्टर के माध्यम से बह नमूना के प्रतिदीप्ति तीव्रता (ओं), जिसमें एक electropherogram के रूप में प्रस्तुत किया है. इस साजिश के चार प्रतिदीप्ति चैनल को इसी अतिव्यापी निशान के रूप लेता हैअलग fluorophores का पता लगाने के लिए किया जाता है, और प्रत्येक का पता लगाने व्यक्ति सीडीएनए या अनुक्रमण उत्पादों के लिए इसी चोटियों के शामिल है, जहां. Electropherogram डेटा एक टैब सीमांकित पाठ फ़ाइल के रूप में आनुवंशिक विश्लेषक से निर्यात और ShapeFinder परिवर्तन और विश्लेषण सॉफ्टवेयर 18 में आयात किया जाता है.
ShapeFinder शुरू में प्रवास बार और शिखर मात्रा में सही क्रमशः प्रतिक्रिया उत्पादों की पहचान और मात्रा को प्रतिबिंबित है कि यह सुनिश्चित करने के लिए डेटा पर गणितीय परिवर्तनों की एक श्रृंखला प्रदर्शन करने के लिए प्रयोग किया जाता है. चोटियों तो गठबंधन और एकीकृत, और परिणाम प्राथमिक आरएनए अनुक्रम के साथ एक साथ सारणीबद्ध रहे हैं. शाही सेना के उचित खंड के लिए एक "जेट प्रोफ़ाइल" (+) न्यूक्लियोटाइड प्रत्येक शाही सेना के साथ जुड़े मूल्यों से नियंत्रण मूल्यों घटाकर, और नीचे वर्णित के रूप में डेटा सामान्य से प्राप्त किया जाता है. इस प्रोफाइल सामान्यीकृत जेट वैल धर्मान्तरित (v5.3) सॉफ्टवेयर 19,20, RNAstructure में आयात किया जाता हैशाही सेना माध्यमिक संरचना तह एल्गोरिथ्म में शामिल कर रहे हैं कि छद्म ऊर्जा की कमी में ues. इस तरह से एल्गोरिदम जांच और तह रासायनिक संयोजन काफी अकेले 12,21 या तो विधि की तुलना संरचना भविष्यवाणी की सटीकता में सुधार. RNAstructure का उत्पादन (v5.3) सबसे कम ऊर्जा की छवियों शामिल शाही सेना माध्यमिक संरचनाओं शाब्दिक डॉट ब्रैकेट अंकन में आकार जेट प्रोफाइल (एस), और साथ ही एक ही संरचना के साथ रंग कोडित. बाद के बाद इस तरह वर्ना 22 और PseudoViewer 23 के रूप में शाही सेना माध्यमिक संरचना की चित्रमय प्रदर्शन करने के लिए समर्पित सॉफ्टवेयर को निर्यात किया जा सकता है.
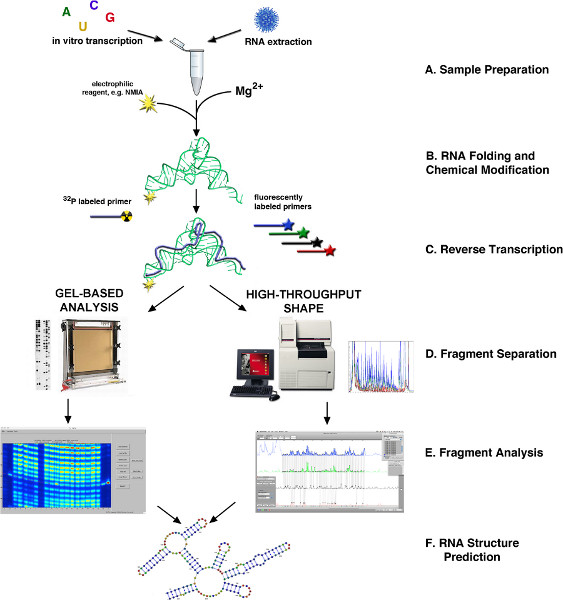
चित्रा 1. आकार 4,10 के माध्यम से शाही सेना संरचना निर्धारण के फ़्लोचार्ट. (ए) शाही सेना मीटरप्र जैविक नमूने से या इन विट्रो प्रतिलेखन में द्वारा प्राप्त किया. (बी) के स्रोत पर निर्भर करता है, शाही सेना मुड़ा हुआ या अन्यथा संसाधित और आकार अभिकर्मक के साथ संशोधित किया गया है. (सी) रिवर्स प्रतिलेखन fluorescently या radioactively लेबल प्राइमरों का उपयोग. (डी) सीडीएनए उत्पादों रहे हैं या तो केशिका या स्लैब जेल आधारित वैद्युतकणसंचलन माध्यम fractionated. (ई) टुकड़ा विश्लेषण. (एफ) आरएनए संरचना भविष्यवाणी. बड़ा आंकड़ा देखने के लिए यहां क्लिक करें.
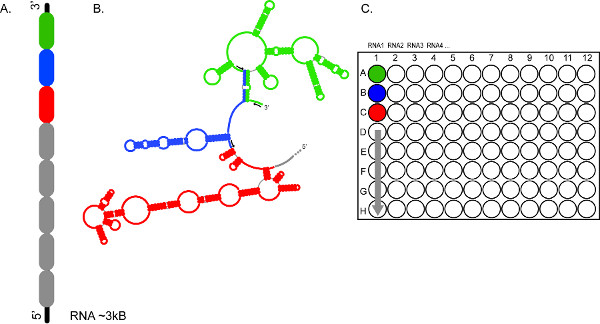
चित्रा 2. CE-आधारित आकार के उच्च throughput चरित्र तेजी से कई RNAs के विश्लेषण, और / या एक ही शाही सेना के कई क्षेत्रों की अनुमति देता है. (ए) </strong> एक शाही सेना 300-600 NT के वर्गों (रंग हरा, नीला और लाल रंग में कोडित) (बी) शाही सेना की धारा फ्लोरोसेंट प्राइमरों के विभिन्न सेट (काला तीर) का उपयोग कर स्वतंत्र रूप से जांच कर रहे हैं (सी) के समूह में विभाजित किया जा सकता है कि कैसे का प्रतिनिधित्व करता है प्रतिक्रियाओं ~ 3 केबी RNA1 लिए पूर्ण कवरेज प्रदान करने, क्रमशः कुओं ए 1, बी 1, सी 1, आदि में जमा और लोड कर रहे हैं. RNAs के 2, 3, 4, आदि से रिएक्शन उत्पादों इसी तरह लगातार electrophoretic रन में विभाजन के लिए तैयार किया जा सकता है. बड़ा आंकड़ा देखने के लिए यहां क्लिक करें.
Protocol
Representative Results
Discussion
हम यहाँ उच्च throughput आकार के लिए एक विस्तृत प्रोटोकॉल, किसी भी आकार के RNAs के लिए एकल nucleotide संकल्प माध्यमिक संरचना निर्धारण की अनुमति देता है कि एक तकनीक मौजूद है. इसके अलावा, माध्यमिक संरचना भविष्यवाणी एल्गो?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
एस Lusvarghi, जे Sztuba-Solinska, के.जे. Purzycka, जेडब्ल्यू रॉश और SFJ ले Grice राष्ट्रीय कैंसर संस्थान, राष्ट्रीय स्वास्थ्य संस्थान, संयुक्त राज्य अमेरिका के अंदर का रिसर्च प्रोग्राम द्वारा समर्थित हैं.
Materials
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5′ labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied – including any 3’terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies |
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 ‘-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 ‘-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2′-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues – A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).

