ハイスループットSHAPEを使用したRNA二次構造予測
Summary
プライマー伸長により解析し、高スループット選択2 'ヒドロキシルアシル化(SHAPE)数百から一塩基の解像度で数千ヌクレオチドRNAの構造を決定するための技術が、逆転写、キャピラリー電気泳動および二次構造予測ソフトウェアをプロービングする新規化学物質を利用しています。
Abstract
生物学的プロセスに関与してRNAの機能を理解することは、RNA構造の徹底的な知識が必要です。この終わりに向かって、またはSHAPE "プライマー伸長により解析のハイスループット選択2 'ヒドロキシルアシル化"と呼ばれる方法論は、一塩基分解能を持つRNAの二次構造を予測することができます。このアプローチは、優先的に水溶液中でのRNAの一本鎖またはフレキシブル領域をアシル化する化学プローブエージェントを利用しています。化学修飾部位が修飾されたRNAの逆転写によって検出され、この反応の生成物は、自動化されたキャピラリー電気泳動(CE)によって分画されている。逆転写酵素は、SHAPE試薬によって変更それらのRNAヌクレオチドで一時停止しているため、得られたcDNAライブラリーは、間接的に、単一の折られたRNAの文脈で足止めされているそれらのリボヌクレオチドをマッピング。 ShapeFinderソフトウェアを使用して、自動化されたCEによって生成された電気泳動図はNUに加工と変換される自身がRNAStructure(V5.3)予測アルゴリズムで使用される擬似エネルギー制約に変換されcleotide反応テーブル。 シリコ RNAの二次構造予測でプロービングSHAPEを組み合わせることにより得られる二次元RNA構造は単独の方法を用いて得られた構造体よりもはるかに正確であることが見出されている。
Introduction
スプライシング、翻訳、ウイルス複製および癌の調節に関与する触媒と非コーディングRNAの機能を理解するために、RNAの構造の詳細な知識が必要である1,2。残念なことに、RNA折りたたみの正確な予測は手ごわい課題を提示。クラシックプロービングエージェントは実験当たり100-150ヌクレオチドに限定され、このような毒性など多くの欠点から不完全なヌクレオチドカバレッジおよび/またはスループットに苦しむ。肉眼二次構造予測アルゴリズムは、効果的に精力的に類似した構造を区別することができないことに起因する不正確さに起因し、同様に不利である。特に大規模なRNAは、また、それらのコンフォメーションの柔軟性と、これらの技術に必要な高純度の試料を大量に起因するX線結晶構造解析と核磁気共鳴(NMR)分光法、などの3D構造決定の方法にしばしば難治性である。
HIGHスループットのSHAPEは、単一ヌクレオチド解像度で大RNAの構造をプロービングする効果的な、単純なアプローチを提供することによってこれらの問題の多くを解決する。また、SHAPEに使用される試薬は、取り扱いが安全で容易であり、試薬をプロービング他のほとんどの化学物質とは対照的に、すべての4つのリボヌクレオチドと反応する。これらの試薬 はまた、 インビボ可能コンテキスト (複数可)3 でその中にRNAを精査すること、細胞膜を貫通することができる。もともと週実験室で開発された4、SHAPEはRNAの多種多様〜9キロバイトHIV-1 RNAゲノムの5の完全な二次構造の決定が最も顕著な例を分析するために使用されている。 SHAPEを使用して他の注目すべき成果はビリオン関連HIV-1 RNAの3中のタンパク質結合部位を同定するだけでなく、感染ウイロイド6、人間の長い非コードRNA 7、酵母リボソーム8、リボスイッチ9の構造の解明を含む。 WHイルSHAPEプロトコルの元と高スループットの変化が他の場所で10-12公開されている、本研究は、蛍光オリゴヌクレオチドを用いてハイスループットSHAPEによるRNAの二次構造の決定の詳細な説明は、ベックマン·コールターCEQ 8000遺伝分析装置を提供し、 SHAPEfinderとRNAStructure(V5.3)ソフトウェア。以前に未発表の技術的な詳細とトラブルシューティングに関するアドバイスも含まれています。
SHAPEのバリエーション
SHAPEとそのバリエーションの本質は選択的に修正のサイトでかさばる付加物を生産し、2'-水酸基(2'-OH)リボースグループをアシル求電子無水物に水溶液中のRNAの露出です。この化学反応は、ベースが対になっている間、一本鎖ヌクレオチドは、これらの試薬による求電子攻撃を助長するコンホメーションを採用することが、より傾向があるように、地元のRNA構造力学を尋問の手段として、またはアーキテクチャコンストラクタainedヌクレオチドは10以下または非反応性である。付加体形成の部位が修飾されたRNA( "(+)"プライマー伸長反応)上の特定の部位にハイブリダイズし、蛍光または放射性標識されたプライマーから開始する逆転写によって検出される。逆転写酵素(RT)でアシル化リボヌクレオチドを通過しなかった場合には、cDNA産物のプールは、その長さ修飾部位と一致製造される。制御"( – )"試薬にさらされていないRNAを用いたプライマー伸長反応は、構造、非特異的なRNA鎖の破損等を RNAにそのDNA合成の早期終了( すなわち 、 "止まり")が原因ように制御されるかもしれない。化学修飾によって生成された一時停止と区別する。最後に、同じプライマーから開始する2ジデオキシ配列決定反応は電気泳動後のRNA一次配列と反応ヌクレオチドを相関させるためのマーカーとして使用される。
SHAPの元のアプリケーションで( – )、および2つ配列決定反応E、同一の32 P-末端標識プライマー(+)のために利用される。これらの反応の生成物は、5〜8%ポリアクリルアミドスラブゲルに隣接するウェルにロードし、ポリアクリルアミドゲル電気泳動(PAGE; 図1)変性により分画している。従来のSHAPEによって生成されたゲル画像の定量的分析は、SAFA、半自動フット解析ソフトウェア13を用いて行うことができる。
対照的に、高スループットSHAPEは、蛍光標識プライマーおよび自動化されたキャピラリー電気泳動を用いる。具体的には、調査中のRNAのそれぞれの地域のために、一般的な配列を有する4 DNAプライマーのセットが、異なる5 '蛍光標識は、合成または購入する必要があります。これらの異なる標識オリゴヌクレオチドは、プライム2 SHAPE反応と2配列決定反応、プールし、分留/自動キャピラリー電気泳動(CE)によって検出されたそのうちの製品に役立つ。 WHEREASのRNAの100〜150ヌクレオチドの反応性プロフィールは、独創的なアプローチを使用して、4つの一連の反応から得ることができる、高スループットSHAPEは、単一のプールされた試料300から600 3ヌクレオチドの分解能を可能にする。できるだけ多くの96としてサンプルが12連続したCEの実行( 図2)の過程で分別のために準備ができますが、同時に、分別することがあり、最大の反応8セットまで。また、SHAPEfinderソフトウェアは、プロセスに開発され、CEQや他の遺伝的分析装置から出てくるデータを分析し、より自動化されており、SAFA 13または他のゲル解析パッケージよりもはるかに少ないユーザーの介入を必要とする。
より高度な高スループットの方法論は、最近取得した次世代シーケンシング技術と一緒に構造特異的酵素ではなく、アルキル化試薬を使用PARS(RNA構造の並列解析)14とFragの-SEQ(断片シークエンシング)15、などの浮上しているINFORMATIORNA構造に関するnである。これらの技術の魅力にもかかわらず、プロービングヌクレアーゼに固有の多くの制限はまだ16のままです。これらの問題は、次世代のシーケンシングは、従来の形状で実施したのと同様の化学修飾およびRNAの逆転写によって先行されるSHAPEシークエンシング(配列SHAPE-)17プロトコルで回避することができる。これらのメソッドは、RNAの構造決定の未来を表すことが、次世代シーケンシングは非常に高価であり、多くの研究室が利用できないままであることを覚えておくことが重要です。
形状データ解析
遺伝分析装置に生成されたデータは、移動時間のインデックスに対してプロットされているキャピラリ検出器に流れる試料の蛍光強度(複数可)を含み、電気泳動の形で提示される。このプロットは、4つの蛍光チャネルに対応する重複のトレースの形をとるsが異なる蛍光を検出するために使用され、各トレースは、個々のcDNAの配列決定または製品に対応するピークで構成される。電気泳動データは、タブ区切りのテキストファイルとして遺伝アナライザからエクスポートしShapeFinder変換と解析ソフトウェア18にインポートされます。
ShapeFinderは、最初の移行時間とピークのボリュームを正確それぞれ、反応生成物のアイデンティティと数量を反映することを保証するためにデータを数学的な一連の変換を実行するために使用される。ピークは次いで、位置合わせて一体化し、その結果、一次RNA配列と一緒にまとめられている。 RNAの当該セグメントの "反応性プロフィールは、"(+)RNAヌクレオチドそれぞれに関連付けられた値からの制御値を減算し、後述するように、データを正規化することによって得られる。このプロファイルは、正規化された反応性valを変換しRNAstructure(V5.3)ソフトウェア19,20、にインポートされRNAの二次構造の折りたたみアルゴリズムに組み込まれている擬似エネルギー制約へのUE。このようにアルゴリズムをプロービングや製本化学物質を組み合わせることで大幅にどちらかの方法だけでは12,21に比べて構造予測の精度を向上させます。 RNAstructureの出力(V5.3)は、最低エネルギーの画像を含むRNA二次構造テキストドットブラケット表記でSHAPE反応性プロファイル(S)だけでなく、同じ構造を色分け。後者はその後、そのようなヴァルナ22およびPseudoViewer 23としてRNA二次構造のグラフィカル表示に専用のソフトウェアにエクスポートすることもできる。
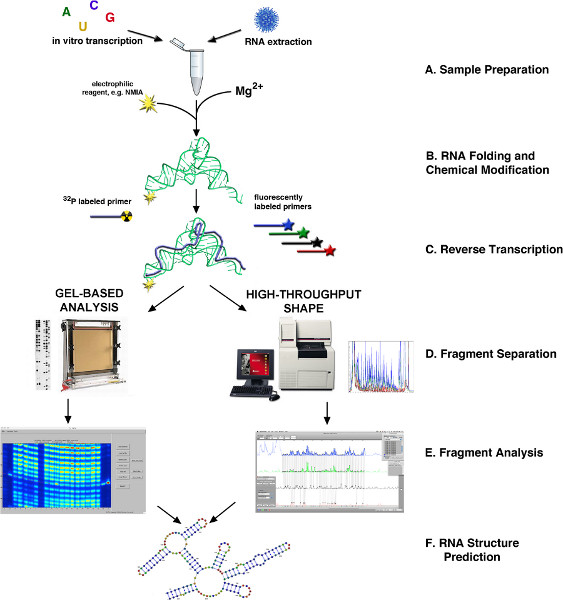
図1。 SHAPE 4,10経由RNAの構造決定のフローチャート(A)RNA MAYは、生物学的試料から、またはインビトロ転写によって得られる。(B)ソースに応じて、RNAは、折り畳まれたまたは他の方法で処理され、SHAPE試薬で修飾されている。(C)逆転写反応は、蛍光または放射性標識プライマーを用いた(D)cDNA産物であるどちらキャピラリーまたはスラブゲルベースの電気泳動経由画し。(E)フラグメント分析。(F)のRNA構造予測。 より大きい数字を表示するには、ここをクリックしてください。
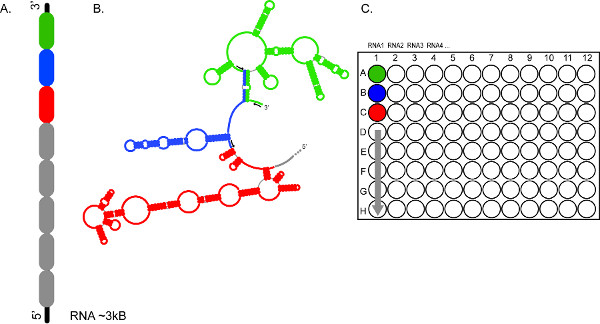
図2。 CEベースのSHAPEの高スループットの文字が急速複数RNAの解析、および/ または、同じRNAの複数のセグメントを許可します。() </st栄>は(緑、青と赤のカラーコード化)(B)RNAの切片を蛍光プライマーの異なるセット(黒矢印)(C)を用いて独立して、プローブされたRNAは、300〜600ヌクレオチドの区間に分割することができる方法のセットを表し反応は〜3キロバイトRNA1のための完全なカバレッジを提供し、それぞれのウェルA1、B1、C1、 等にプールされ、ロードされています。 RNAは2、3、4等からの反応生成物は、同様に連続した電気泳動の実行で分画に調製することができる。 より大きい数字を表示するには、ここをクリックしてください。
Protocol
Representative Results
Discussion
我々はここで高スループットSHAPEの詳細なプロトコルがあるどの規模のRNAのための一塩基の解像度に二次構造の決定を可能にする手法を提示する。また、二次構造の予測アルゴリズムを実験SHAPEデータを結合するだけではどちらの方法で可能であるよりも高精度にRNA二次元モデルの生成を容易にする。蛍光標識されたプライマーと自動CEの組み合わせは、単一の実験で長いRNA配列の解決を促進?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
S. Lusvarghi、J. Sztuba-Solinska、KJ Purzycka、JWラウシュとSFJル·グライスは、国立癌研究所、国立衛生研究所、米国の学内研究プログラムによってサポートされています。
Materials
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5′ labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied – including any 3’terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies |
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 ‘-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 ‘-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2′-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues – A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).

