Высокая пропускная способность Live Imaging микроколоний для измерения неоднородности в росте и экспрессии генов
Summary
Фенотипы роста дрожжей точно измеряются с помощью высокой параллельной покадровой визуализации обездвиженные клетки, растущие в микроколонии. Одновременно можно контролировать стрессоустойчивость, экспрессию белка и локализацию белка, создавая интегрированные наборы данных для изучения того, как экологические и генетические различия, а также неоднородность генной экспрессии среди изогенных клеток, модулируют рост.
Abstract
Точные измерения неоднородности между штаммом и внутри штамма в ставках роста микробов имеют важное значение для понимания генетических и экологических факторов, вводимых в стрессоустойчивость, патогенность и другие ключевые компоненты фитнеса. Эта рукопись описывает микроскоп на основе анализа, который отслеживает примерно 105 Saccharomyces cerevisiae микроколонии за эксперимент. После автоматической покадровой визуализации дрожжей, обездвиженные в многоукрашенной пластине, темпы роста микроколонии легко анализируются с помощью пользовательского программного обеспечения для анализа изображений. Для каждой микроколонии также можно контролировать экспрессию и локализацию флуоресцентных белков и выживание острого стресса. Этот анализ позволяет точно оценить средние темпы роста штаммов, а также всестороннее измерение неоднородности роста, экспрессии генов и стрессоустойчивости в клональных популяциях.
Introduction
Фенотипы роста вносят критический вклад в фитнес дрожжей. Естественный отбор может эффективно различать линии с темпами роста, отличающимися от обратного эффективного размера популяции, который может превышать 108 особей1. Кроме того, изменчивость темпов роста среди людей в популяции является эволюционно актуальным параметром, так как она может служить основой для стратегий выживания,таких как хеджирование ставок 2,3,4,5,6. Поэтому анализы, позволяющие проводить высокотоктяжелые измерения фенотипов роста и их распределения, имеют решающее значение для изучения микроорганизмов. Анализ роста микроколонии, описанный здесь, может генерировать индивидуальные измерения скорости роста на 105 микроколоний за эксперимент. Таким образом, этот анализ обеспечивает мощный протокол для изучения дрожжевой эволюционной генетики и геномики. Он поддается особенно хорошо для тестирования, как изменчивость в популяциях генетически идентичных одиночных клеток генерируется, поддерживается, и способствует популяции фитнес7,8,9,10.
Метод, описанный здесь (Рисунок 1) использует периодически захваченных, с низким увеличением яркое поле изображения клеток, растущих в жидких средствах массовой информации на 96- или 384-хорошо стеклянной нижней пластины для отслеживания роста в микроколонии. Клетки придерживаются лектина конканавалина А, который покрывает дно микроскопической пластины, и образуют двумерные колонии. Поскольку микроколонии растут в монослойном, область микроколонии сильно коррелирует с номером клетки7. Таким образом, точные оценки темпов роста микроколонии и времени задержки могут быть получены с помощью пользовательского программного обеспечения для анализа изображений, которое отслеживает скорость изменения площади каждой микроколонии. Кроме того, экспериментальная установка может контролировать изобилие и даже субклеточную локализацию флуоресцентно помеченных белков, выраженных в этих микроколониях. Вниз по течению обработки данных из этого микроколонии роста анализа может быть достигнуто путем пользовательского анализа или существующего программного обеспечения анализа изображений, таких как обработка изображений легко (PIE)11, алгоритм для надежного распознавания колонии области и высокой пропускной способности анализа роста с низким увеличением, яркие изображения, которая доступна через GitHub12.
Поскольку оценки темпов роста, полученные в результате анализа микроколонии роста, генерируются на основе большого числа одноколонных измерений, они чрезвычайно точны, при этом стандартные ошибки на несколько порядков меньше, чем сами оценки для эксперимента разумного размера. Таким образом, сила анализа для выявления различий в темпах роста между различными генотипами, обработками или условиями окружающей среды высока. Формат мультиэлементной пластины позволяет сравнивать множество различных комбинаций среды и генотипа в одном эксперименте. Если штаммы constitutively выражают различные флуоресцентные маркеры, они могут быть смешаны в том же хорошо и отличается последующим анализом изображения, который может увеличить мощность дальше, позволяя хорошо за колодец нормализации данных.
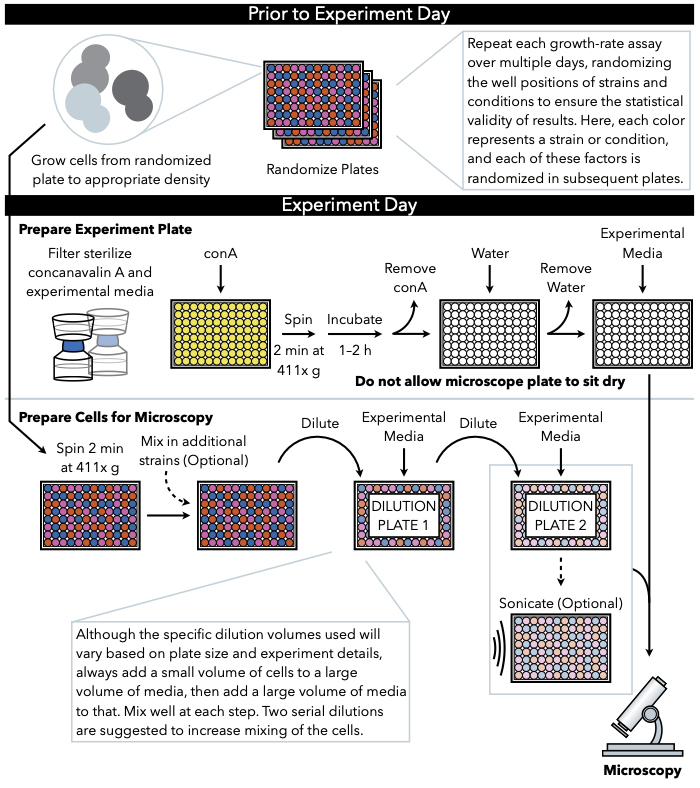
Рисунок 1: Схематическое представление протокола. Этот протокол следует за 2 главным образом шагами, которые подготовка экспериментальной плиты и подготовка клеток к изображению. Рандомизация пластин и рост клеток должны проводиться до и в течение дня эксперимента. Повторное смешивание клеток на каждом шагу во время разбавления необходимо в шагах до покрытия, и поэтому подготовка экспериментальной пластины сначала рекомендуется так, что она готова к покрытие сразу после завершения разбавления клеток. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Protocol
Representative Results
Discussion
Описанный здесь протокол представляет ею универсальный анализ, позволяющий одновременно контролировать рост клеток и экспрессию генов на уровне отдельных микроколоний. Сочетание этих двух условий дает уникальную биологическую информацию. Например, предыдущая работа использовала э?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Мы благодарим Наоми Зив, Сашу Леви и Шуанг Ли за их вклад в разработку этого протокола, Дэвида Грешема за совместное оборудование и Мариссу Нолл за помощь в производстве видео. Эта работа была поддержана Национальными институтами здравоохранения грант R35GM118170.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
References
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Génétique. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Génétique. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Génétique. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

