High-Throughput Live Imaging von Mikrokolonien zur Messung der Heterogenität in Wachstum und Genexpression
Summary
Hefewachstumsphänotypen werden präzise durch hochparallele Zeitraffer-Bildgebung von immobilisierten Zellen gemessen, die zu Mikrokolonien heranwachsen. Gleichzeitig können Stresstoleranz, Proteinexpression und Proteinlokalisierung überwacht werden, wodurch integrierte Datensätze generiert werden, um zu untersuchen, wie ökologische und genetische Unterschiede sowie die Genexpressionsheterogenität unter isogenen Zellen das Wachstum modulieren.
Abstract
Präzise Messungen der Heterogenität zwischen und innerhalb der Dehnung in mikrobiellen Wachstumsraten sind entscheidend für das Verständnis genetischer und ökologischer Inputs in Stresstoleranz, Pathogenität und anderen Schlüsselkomponenten der Fitness. Dieses Manuskript beschreibt einen mikroskopischen Assay, der pro Experiment etwa 105 Saccharomyces cerevisiae mikrocolonies nachverfolgt. Nach der automatisierten Zeitraffer-Bildgebung von Hefe, die in einer Multiwell-Platte immobilisiert ist, lassen sich die Wachstumsraten von Mikrokolonien mit einer benutzerdefinierten Bildanalysesoftware einfach analysieren. Für jede Mikrokolonie können auch die Expression und Lokalisation fluoreszierender Proteine und das Überleben akuter Belastungen überwacht werden. Dieser Test ermöglicht eine genaue Schätzung der durchschnittlichen Wachstumsraten der Stämme sowie eine umfassende Messung der Heterogenität in Wachstum, Genexpression und Stresstoleranz innerhalb klonaler Populationen.
Introduction
Wachstumsphänotypen tragen entscheidend zur Hefefitness bei. Die natürliche Selektion kann effizient zwischen Linien unterscheiden, wobei die Wachstumsraten durch die Umkehrung der effektiven Bevölkerungsgröße abweichen, die 108 Individuen1 überschreiten kann. Darüber hinaus ist die Variabilität der Wachstumsraten bei Individuen innerhalb einer Bevölkerung ein evolutionär relevanter Parameter, da er als Grundlage für Überlebensstrategien wie Wetten hedging2,3,4,5,6dienen kann. Daher sind Assays, die hochgenaue Messungen von Wachstumsphänotypen und deren Verteilungen ermöglichen, für die Untersuchung von Mikroorganismen von entscheidender Bedeutung. Der hier beschriebene Mikrokolonie-Wachstumstest kann individuelle Wachstumsratenmessungen für 105 Mikrokolonien pro Experiment generieren. Dieser Test bietet daher ein leistungsfähiges Protokoll, um Hefe evolutionäre Genetik und Genomik zu studieren. Es eignet sich besonders gut, um zu testen, wie Variabilität innerhalb von Populationen von genetisch identischen Einzelzellen erzeugt, gepflegt wird und zur Eignung der Bevölkerung beiträgt7,8,9,10.
Die hier beschriebene Methode (Abbildung 1) verwendet periodisch erfasste Hellfeldbilder von Zellen mit geringer Vergrößerung, die in flüssigen Medien auf einer 96- oder 384-Well-Glasbodenplatte wachsen, um das Wachstum in Mikrokolonien zu verfolgen. Die Zellen haften an dem Lektin Concanavalin A, das den Boden der Mikroskopplatte überzieht, und bilden zweidimensionale Kolonien. Da die Mikrokolonien in einer Monoschicht wachsen, ist das Mikrokoloniegebiet stark mit der Zellnummer7korreliert. Daher können genaue Schätzungen der Wachstumsrate und Verzögerungszeit von Mikrokolonien mit einer benutzerdefinierten Bildanalysesoftware generiert werden, die die Änderungsrate der Fläche jeder Mikrokolonie nachverfolgt. Darüber hinaus kann der Versuchsaufbau die Häufigkeiten und sogar die subzellulären Lokalisationen fluoreszierend markierter Proteine überwachen, die in diesen Mikrokolonien exprimiert werden. Die nachgeschaltete Verarbeitung von Daten aus diesem Mikrokolonie-Wachstumstest kann durch benutzerdefinierte Analyse oder durch vorhandene Bildanalysesoftware erreicht werden, wie z. B. Processing Images Easily (PIE)11, ein Algorithmus für robuste Kolonieflächenerkennung und Wachstumsanalyse mit hohem Durchsatz aus Hellfeldbildern mit geringer Vergrößerung, die über GitHub12verfügbar sind.
Da wachstumsfördernde Schätzungen, die aus dem Mikrokolonie-Wachstumstest abgeleitet werden, aus einer großen Anzahl von Einzelkolonienmessungen generiert werden, sind sie extrem genau, mit Standardfehlern, die um mehrere Größenordnungen kleiner sind als die Schätzungen selbst für ein angemessen großes Experiment. Daher ist die Kraft des Assays, Wachstumsratenunterschiede zwischen verschiedenen Genotypen, Behandlungen oder Umgebungsbedingungen zu erkennen, hoch. Das Multiwell-Plattenformat ermöglicht den Vergleich zahlreicher unterschiedlicher Umgebungs- und Genotypkombinationen in einem einzigen Experiment. Wenn Stämme konstitutiv unterschiedliche fluoreszierende Marker exprimieren, können sie in der gleichen Bohrung gemischt und durch nachfolgende Bildanalyse unterschieden werden, was die Leistung weiter erhöhen könnte, indem sie eine gut-durch-gut-datennormalisierung ermöglichen.
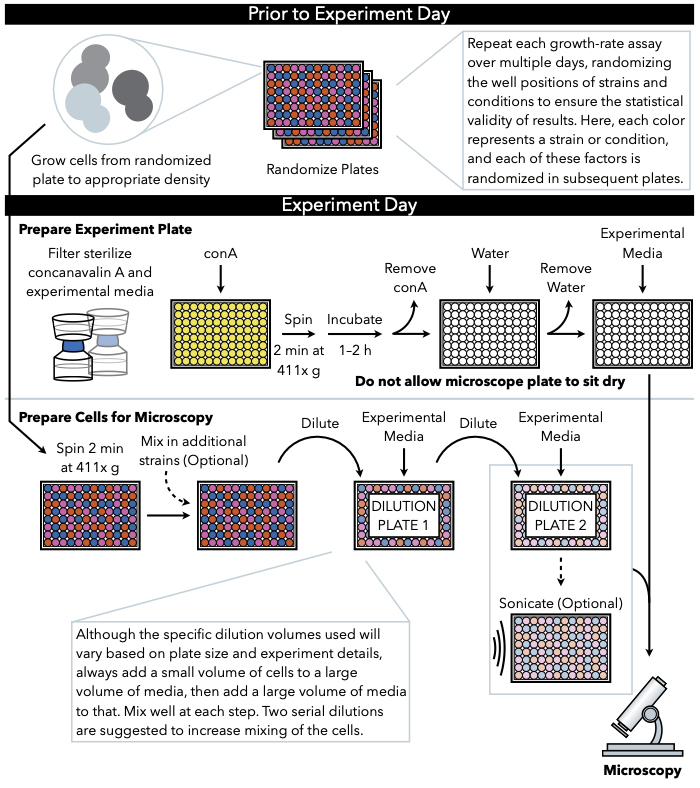
Abbildung 1:Schematische Darstellung des Protokolls. Dieses Protokoll folgt zwei Hauptschritten, die die Vorbereitung der Versuchsplatte und die Vorbereitung der Zellen auf das Bild sind. Die Randomisierung der Platten und das Wachstum der Zellen sollten vor und vor dem Experimenttag durchgeführt werden. Das wiederholte Mischen von Zellen bei jedem Schritt während der Verdünnung ist in den Schritten bis zur Beschichtung zwingend erforderlich, und daher wird die Vorbereitung der Versuchsplatte zuerst empfohlen, so dass sie sofort nach Abschluss der Zellverdünnung zur Beschichtung bereit ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protocol
Representative Results
Discussion
Das hier beschriebene Protokoll ist ein vielseitiger Test, der es ermöglicht, Zellwachstum und Genexpression gleichzeitig auf der Ebene einzelner Mikrokolonien zu überwachen. Die Kombination dieser beiden Modalitäten ergibt einzigartige biologische Erkenntnisse. Zum Beispiel haben frühere Arbeiten diesen Test verwendet, um eine negative Korrelation zwischen der Expression des TSL1-Gens und der Mikrokolonie-Wachstumsrate in isogenen Wildtypzellen zu zeigen, indem beide gleichzeitig7<su…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Wir danken Naomi Ziv, Sasha Levy und Shuang Li für ihre Beiträge zur Entwicklung dieses Protokolls, David Gresham für gemeinsame Ausrüstung und Marissa Knoll für die Hilfe bei der Videoproduktion. Diese Arbeit wurde durch den Zuschuss der National Institutes of Health R35GM118170 unterstützt.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
References
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Génétique. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Génétique. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Génétique. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

