تصوير حي عالي الإنتاجية للمستعمرات الدقيقة لقياس التغايرية في النمو والتعبير الجيني
Summary
يتم قياس الأنماط الظاهرية لنمو الخميرة بدقة من خلال التصوير المتوازي للغاية للفاصل الزمني للخلايا المشلولين التي تنمو إلى مستعمرات صغيرة. وفي الوقت نفسه، يمكن رصد تحمل الإجهاد، والتعبير عن البروتين، وتوطين البروتين، وتوليد مجموعات بيانات متكاملة لدراسة كيفية تعديل النمو بين الاختلافات البيئية والجينية، فضلا عن عدم التجانس بين الخلايا متساوية المنشأ.
Abstract
القياسات الدقيقة للتباين بين وداخل سلالة في معدلات النمو الميكروبية ضرورية لفهم المدخلات الوراثية والبيئية في تحمل الإجهاد، والإمراض، وغيرها من المكونات الرئيسية للياقة البدنية. تصف هذه المخطوطة المقايسة المستندة إلى المجهر التي تتعقب ما يقرب من 105 ساككروميس سيريفيسيا الألوان الدقيقة لكل تجربة. بعد التصوير الآلي الفاصل الزمني للخميرة معطلة في لوحة متعددة بويل ، يتم تحليل معدلات نمو الألوان الدقيقة بسهولة مع برامج تحليل الصور المخصصة. لكل ميكروكولون، والتعبير وتوطين البروتينات الفلورية والبقاء على قيد الحياة من الإجهاد الحاد يمكن أيضا رصدها. ويسمح هذا المقايسة بتقدير دقيق لمتوسط معدلات نمو السلالات، فضلا عن القياس الشامل لعدم التجانس في النمو، والتعبير الجيني، وتحمل الإجهاد داخل المجموعات السكانية اللاستنساخية.
Introduction
الأنماط الظاهرية للنمو تسهم بشكل حاسم في اللياقة البدنية الخميرة. الانتقاء الطبيعي يمكن أن يميز بكفاءة بين الأنساب مع معدلات النمو تختلف عن طريق عكس حجم السكان الفعال، والتي يمكن أن تتجاوز 108 أفراد1. وعلاوة على ذلك، فإن تقلب معدلات النمو بين الأفراد داخل السكان هو المعلمة ذات الصلة تطوريا، لأنها يمكن أن تكون بمثابة الأساس لاستراتيجيات البقاء على قيد الحياة مثل التحوط الرهان2،3،4،5،6. ولذلك، فإن المقايسات التي تسمح بإجراء قياسات دقيقة للغاية للأنماط الظاهرية للنمو وتوزيعاتها هي محورية لدراسة الكائنات الحية الدقيقة. يمكن أن يولد اختبار نمو الألوان الدقيقة الموصوف هنا قياسات معدل النمو الفردي ل ~105 ميكروكولونات لكل تجربة. ولذلك يوفر هذا المقايسة بروتوكولا قويا لدراسة علم الوراثة التطورية الخميرة وعلم الجينوم. فإنه يفسح المجال بشكل جيد بشكل خاص لاختبار كيف يتم إنشاء التباين داخل السكان من الخلايا المفردة متطابقة وراثيا، والحفاظ عليها، ويساهم في اللياقة البدنية للسكان7،8،9،10.
تستخدم الطريقة الموضحة هنا(الشكل 1)صورا ذات حقول ساطعة يتم التقاطها بشكل دوري ومنخفضة التكبير للخلايا التي تنمو في الوسائط السائلة على لوحة زجاجية قاعية من 96 أو 384 بئرا لتتبع النمو في المستعمرات الدقيقة. تلتزم الخلايا بالكونسانفالين الليكتين A ، الذي يغطي الجزء السفلي من لوحة المجهر ، وتشكل مستعمرات ثنائية الأبعاد. لأن المستعمرات الدقيقة تنمو في طبقة واحدة، ترتبط منطقة الكولون الدقيق ارتباطا وثيقا مع الخلية رقم7. لذلك، يمكن إنشاء تقديرات دقيقة لمعدل نمو الألوان الدقيقة ووقت التأخر مع برنامج مخصص لتحليل الصور يتتبع معدل التغيير في منطقة كل مجموعة صغيرة. وعلاوة على ذلك، يمكن للإعداد التجريبي رصد وفرة وحتى توطين دون الخلوية من البروتينات المسمى fluorescently أعرب في هذه المستعمرات الدقيقة. يمكن تحقيق معالجة المصب للبيانات من هذا المقايسة نمو الألوان الدقيقة عن طريق التحليل المخصص أو عن طريق برامج تحليل الصور الحالية ، مثل معالجة الصور بسهولة (PIE)11، خوارزمية للتعرف على منطقة المستعمرة القوية وتحليل النمو عالي الإنتاجية من التكبير المنخفض ، صور brightfield ، والتي تتوفر عبر GitHub12.
ولأن تقديرات معدل النمو المستمدة من مقايسة نمو المستعمرات الدقيقة يتم توليدها من عدد كبير من قياسات المستعمرة الواحدة، فإنها دقيقة للغاية، مع أخطاء قياسية أصغر بعدة أوامر من الحجم من التقديرات نفسها لتجربة معقولة الحجم. ولذلك، فإن قدرة المقايسة على اكتشاف الاختلافات في معدل النمو بين الأنماط الجينية المختلفة أو العلاجات أو الظروف البيئية عالية. يسمح تنسيق لوحة multiwell العديد من تركيبات البيئة والنمط الجيني المختلفة أن تقارن في تجربة واحدة. إذا كانت السلالات تعبر بشكل أساسي عن علامات فلورية مختلفة ، فقد تكون مختلطة في نفس البئر وتتميز بتحليل الصورة اللاحق ، مما قد يزيد من القوة من خلال السماح بتطبيع البيانات بشكل جيد.
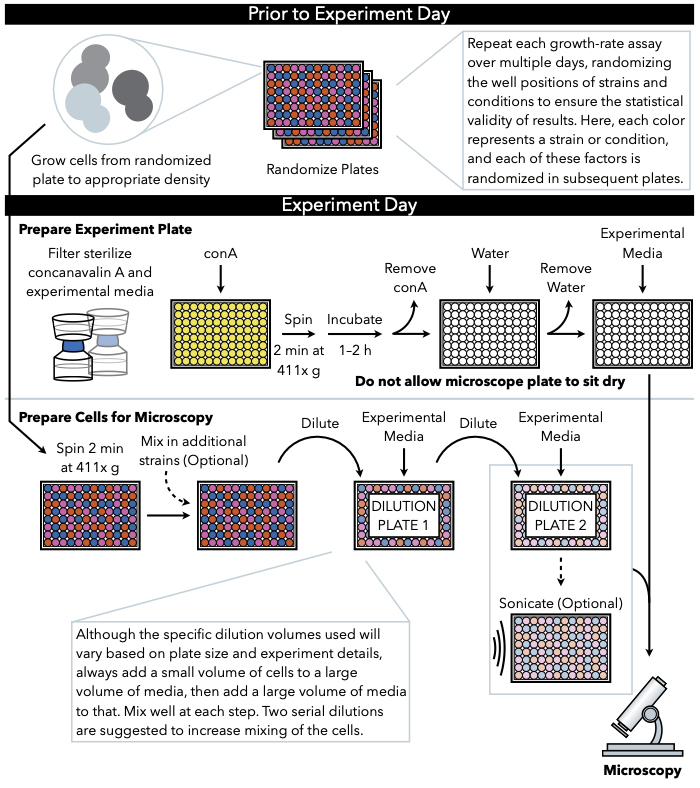
الشكل 1: تمثيل تخطيطي للبروتوكول. يتبع هذا البروتوكول خطوتين رئيسيتين ، وهما إعداد اللوحة التجريبية وإعداد الخلايا للصورة. وينبغي إجراء العشوائية لوحات ونمو الخلايا قبل وحتى يوم التجربة. الخلط المتكرر للخلايا في كل خطوة أثناء التخفيف أمر حتمي في الخطوات حتى الطلاء ، وبالتالي ينصح بإعداد اللوحة التجريبية أولا بحيث تكون جاهزة للطلاء فور الانتهاء من تمييع الخلية. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
Representative Results
Discussion
البروتوكول الموصوف هنا هو اختبار متعدد الاستخدامات يسمح بمراقبة نمو الخلايا والتعبير الجيني في وقت واحد على مستوى الألوان الدقيقة الفردية. والجمع بين هذين الشكلين يسفر عن رؤى بيولوجية فريدة من نوعها. على سبيل المثال، استخدم العمل السابق هذا المقايسة لإظهار ارتباط سلبي بين التعبير عن جين…
Divulgations
The authors have nothing to disclose.
Acknowledgements
نشكر نعومي زيف وساشا ليفي وشوانغ لي على مساهماتهم في تطوير هذا البروتوكول، وديفيد غريشام على المعدات المشتركة، وماريسا نول للمساعدة في إنتاج الفيديو. وقد تم دعم هذا العمل من قبل المعاهد الوطنية للصحة منحة R35GM118170.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
References
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Génétique. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Génétique. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Génétique. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

