RIBO-seq in Bacteria: коллекция образцов и протокол подготовки библиотеки для секвенирования NGS
Summary
Здесь мы опишем этапы отбора проб и подготовки к RIBO-seq у бактерий. Секвенирование библиотек, подготовленных в соответствии с этими руководящими принципами, приводит к получению достаточных данных для всестороннего биоинформатического анализа. Протокол, который мы представляем, прост, использует стандартное лабораторное оборудование и занимает семь дней от лизиса до получения библиотек.
Abstract
Метод профилирования рибосом (RIBO-seq) в настоящее время является наиболее эффективным инструментом для изучения процесса синтеза белка in vivo. Преимуществом этого метода, по сравнению с другими подходами, является его способность контролировать трансляцию путем точного отображения положения и количества рибосом на транскрипте мРНК.
В этой статье мы опишем последовательные этапы сбора проб и подготовки к методу RIBO-seq у бактерий, выделив детали, имеющие отношение к планированию и выполнению эксперимента.
Поскольку RIBO-seq опирается на интактные рибосомы и связанные с ними мРНК, ключевым шагом является быстрое ингибирование трансляции и адекватный распад клеток. Таким образом, мы предлагаем фильтрацию и мгновенное замораживание жидкого азота для сбора клеток с дополнительной предварительной обработкой хлорамфениколом для остановки трансляции бактерий. Для распада мы предлагаем шлифовать замороженные клетки раствором и пестиком в присутствии оксида алюминия, чтобы механически разрушить клеточную стенку. В этом протоколе не требуется сахарозная подушка или ультрацентрифугирование градиента сахарозы для очистки моносом. Вместо этого проводится разделение мРНК с использованием электрофореза полиакриламидного геля (PAGE) с последующим иссечением рибосомного следа (полоса 28-30 нт) и дает удовлетворительные результаты. Это значительно упрощает метод, а также сокращает время и требования к оборудованию для процедуры. Для подготовки библиотеки мы рекомендуем использовать коммерчески доступный небольшой набор РНК для секвенирования Illumina от New England Biolabs, следуя рекомендациям производителя с некоторой степенью оптимизации.
Полученные библиотеки кДНК представляют соответствующее количество и качество, необходимые для секвенирования следующего поколения (NGS). Секвенирование библиотек, подготовленных в соответствии с описанным протоколом, приводит к 2-10 млн однозначно нанесенных на карту считывания на образец, что обеспечивает достаточные данные для всестороннего биоинформатического анализа. Протокол, который мы представляем, является быстрым и относительно простым и может быть выполнен с помощью стандартного лабораторного оборудования.
Introduction
Методика профилирования рибосом (RIBO-seq) была разработана в лаборатории Джонатана Вайсмана в Калифорнийском университете в Сан-Франциско1. По сравнению с другими методами, используемыми для изучения экспрессии генов на трансляционном уровне, RIBO-seq фокусируется на связывании каждой рибосомы с мРНК и предоставляет информацию о ее местоположении и относительном количестве рибосом на транскрипте. Он позволяет контролировать процесс синтеза белка in vivo и может обеспечить одно разрешение и точность кодона, что позволяет измерять плотность рибосом как на отдельной мРНК, так и на всем транскриптоме в клетке. В основе методики RIBO-seq лежит тот факт, что во время трансляции рибосома связывает молекулу мРНК и тем самым защищает погребенный фрагмент транскрипта от переваривания рибонуклеазы. При добавлении рибонуклеазы незащищенная мРНК переваривается, и фрагменты, заключенные рибосомами – обычно длиной ~ 28-30 нт – остаются нетронутыми. Эти фрагменты, называемые рибосомными следами (RF), затем могут быть выделены, секвенированы и нанесены на транскрипт, из которого они произошли, что приводит к обнаружению точного положения рибосом. Фактически, способность рибосомы защищать фрагменты мРНК используется с 1960-х годов для изучения рибосомных сайтов связывания и инициации трансляции (TIS)2,3,4. Однако с развитием технологии глубокого секвенирования RIBO-seq стал золотым стандартом для мониторинга трансляции5, который благодаря взаимодействию с рибосомами может предоставить общегеномную информацию о синтезе белка6. Профилирование рибосом заполнило технологический пробел, который существовал между количественной оценкой транскриптома и протеома6.
Для проведения рибосомного профилирования необходимо получить лизат клеток организма, выросших в исследуемых условиях. Нарушение этих условий во время сбора и лизиса клеток может обеспечить ненадежные данные. Чтобы предотвратить это, обычно используются ингибиторы трансляции, быстрый сбор и мгновенное замораживание в жидком азоте. Клетки могут быть лизированы криогенным измельчением в механическом гомогенизаторе, таком как смеситель мельницы7,8 или бисерный взбиватель9,и путем тритурации через пипетку10 или с помощью иглы11. Буфер лизиса может быть добавлен непосредственно перед или вскоре после измельчения клеток. В нашем протоколе мы используем жидкий азот для предварительного охлаждения раствора и пестиков, а также оксид алюминия в качестве более мягкого подхода к разрушению бактериальной клеточной стенки, что предотвращает сдвиг РНК, часто встречающийся при применении таких методов, как сонификация. После измельчения мы добавляем ледяной буфер лизиса в охлажденный содержимое раствора. Выбор подходящего буфера лизиса важен для получения наилучшего разрешения рибосомных следов. Поскольку ионная сила влияет как на размер ВЧ, так и на точность кадра считывания, в настоящее время рекомендуется использовать лизисные буферы с низкой ионной силой и буферной емкостью, даже если окажется, что буферный состав не влияет на рибосомную занятость на мРНК11,12. Важными компонентами лизисного буфера являются ионы магния, наличие которых предотвращает диссоциацию рибосомных субъединиц и подавляет спонтанные конформационные изменения в бактериальных рибосомах11,13. Ионы кальция также играют значительную роль и необходимы для активности микрококковой нуклеазы (МНазы), используемой в методе профилирования бактериальных рибосом14. Добавление гуанозина 5′-[β,γ-имидо]трифосфата (GMP-PNP), негидролизуемого аналога ГТФ, вместе с хлорамфениколом ингибирует трансляцию во время лизиса15.
При получении лизата его осветляют центрифугированием и делят на две части, каждая для RIBO-seq и высокопроизводительного полного секвенирования мРНК (RNA-seq), поскольку они выполняются одновременно(фиг.1). RNA-seq обеспечивает точку отсчета, которая позволяет сравнивать данные как RIBO-seq, так и RNA-seq во время анализа данных. Исследуемый транслейлом определяется нормализацией рибосомных следов до обилия мРНК16. Данные из RNA-seq также могут помочь идентифицировать артефакты клонирования или секвенирования17.
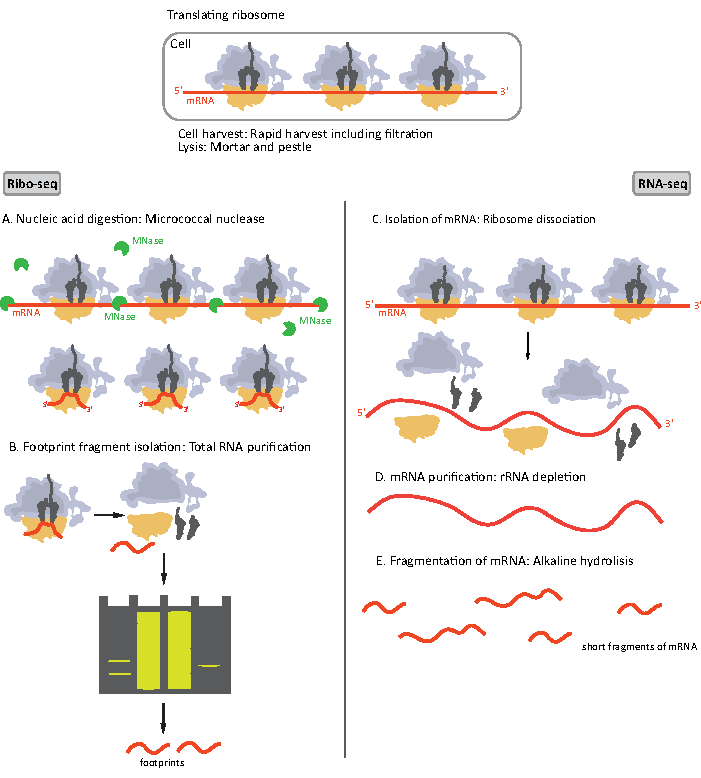
Рисунок 1. Схемы пробоподготовки мРНК для RIBO-seq и RNA-seq. Для подготовки библиотеки RIBO-seq РНК переваривается МНазой (A), с последующим выбором размера РЧ ~28-30 nt длины (B); для РНК-seq РНК выделяют (С), истощают рРНК (D), а полученную мРНК случайным образом фрагментируют на фрагменты различной длины (Е). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Начальные этапы процедуры пробоподготовки для RIBO-seq и RNA-seq отличаются незначительно(рисунок 1). Для рибосомного профилирования лизат должен быть переварен специфической эндонуклеазой, чтобы раздуть молекулы мРНК, не защищенные рибосомами. В стандартных протоколах полученные моносомы восстанавливают ультрацентрифугированием сахарозной подушки или ультрацентрифугированием градиента сахарозы8,14. В этой статье мы показываем, что этот шаг не является необходимым для выделения РЧ, необходимого для RIBO-seq у бактерий, аналогично для эукариотических клеток18,и что выбор размера фрагментов мРНК соответствующей длины из полиакриламидного геля является достаточным.
Для РНК-seq мРНК получается путем истощения рРНК из общей РНК – молекулы рРНК гибридизируются с биотинилированными олигонуклеотидными зондами, которые связываются с магнитными шариками, покрытыми стрептавидином. Комплексы рРНК-олигонуклеотид-шарики затем удаляются из образца магнитом, в результате чего рРНК истощается образцом19,20. Очищенные молекулы мРНК затем случайным образом фрагментируются щелочным гидролизом. Полученные фрагменты мРНК, а также рибосомные следы преобразуются в библиотеки кДНК и подготавливаются к глубокому секвенированию(рисунок 2). Это включает в себя восстановление концов, необходимое после щелочного гидролиза (для мРНК) и ферментативного пищеварения (для RF): дефосфорилирование 3′ концов с последующим фосфорилированием 5′ концов. Следующими шагами являются лигирование адаптеров и обратная транскрипция для создания вставок кДНК, обрамленных последовательностями, необходимыми для секвенирования следующего поколения (NGS) с использованием платформы Illumina. Последняя фаза подготовки библиотеки представляет собой реакцию ПЦР, в которой конструкции усиливаются и маркируются штрих-кодами для конкретных образцов, чтобы обеспечить мультиплексирование и секвенирование различных образцов на одном канале. Перед секвенированием качество и количество библиотек оцениваются с помощью высокочувствительного электрофореза ДНК на кристалле. Затем библиотеки cDNA с соответствующими параметрами могут быть объединены в пул и секвенированы. Секвенирование может выполняться на различных платформах Illumina, таких как MiSeq, NextSeq или HighSeq, в зависимости от количества библиотек, требуемой длины чтения и глубины секвенирования. После секвенирования проводится биоинформатический анализ.

Рисунок 2. Подготовка библиотеки. Подготовка библиотеки включает в себя ремонт концов, перевязку адаптеров, обратную транскрипцию и усиление штрих-кодированием. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Профилирование рибосом является универсальным методом, который может быть легко модифицирован и скорректирован в соответствии с научным вопросом. Первоначально он использовался в дрожжах1,но вскоре после этого он был применен к бактериальным клеткам21, а также к эукариотическим модельным организмам, включая мышь10,рыбку данио22,плодовуюмуху 23 и Arabidopsis thaliana24. Он также использовался для изучения различных типов рибосом: цитоплазматических, митохондриальных25,26 и хлоропластов27,28. У эукариот RIBO-seq обычно адаптируется и уточняется для исследования конкретных аспектов трансляции, включая инициацию10,11,29,30,31,32,удлинение1,10,11,31,33,рибосомное сваливание33 и изменение конформации33. Большинство модификаций предполагают использование различных ингибиторов трансляции. Однако на бактериях аналогичные исследования было трудно провести из-за нехватки ингибиторов с требуемым механизмом действия34. Наиболее часто используемым ингибитором трансляции у бактерий является хлорамфеникол (CAM), который связывается с центром пептидилтрансферазы (PTC) и предотвращает правильное позиционирование аминоацил-тРНК в А-сайте. В результате CAM предотвращает образование пептидной связи, что приводит к остановке удлиняющихся рибосом35. Другими примерами ингибиторов трансляции у бактерий являются тетрациклин (TET)36,ретапамулин (RET)34 и Onc11237, которые использовались для исследования сайтов инициации трансляции. ТЕТ, который предотвращает доставку тРНК в рибосому путем прямого перекрытия с антикодоном стволовым контуром тРНК в А-месте, первоначально применялся для проверки результатов, полученных от лечения CAM, поскольку они оба являются антибиотиками, ингибируя удлинение трансляции38. Было обнаружено, что TET обнаруживает первичный TIS, однако не смог выявить внутренний TIS36. RET связывается в PTC бактериальной рибосомы и предотвращает образование первой пептидной связи путем вмешательства в элонгатор аминоацил-тРНК в месте А. Применение RET приводит к остановке рибосом как на первичном, так и на внутреннем TISs34. Onc112, богатый пролином антимикробный пептид, связывается в выходном туннеле и блокирует связывание аминоацил-тРНК в рибосомном участке А. В результате Onc112 предотвращает вход комплексов инициации в фазуудлинения 37.
Основной информацией, которую обеспечивает профилирование рибосом, является плотность рибосом и их положение на мРНК. Он был успешно применен для исследования дифференциальной экспрессии генов на уровне трансляции в различных условиях роста1,6,измерения поступающей эффективности1,38,39 и обнаружения событий регуляции трансляции, таких как рибосомная пауза10. RIBO-seq также позволяет раскрыть трансляцию аннотированных ncRNA, псевдогенов и неаннотированных малых открытых кадров чтения (ORF), что приводит к идентификации новых и/или очень коротких генов, кодирующих белки10,12,22,30,37. В таких случаях RIBO-seq может точно настроить и улучшить аннотацию генома. Обладая высокой чувствительностью к идентификации переведенных ОРФ и его количественной природой, профилирование рибосом может также служить прокси для определения протеома или в оказании помощи протеомным исследованиям31,34,39. Путем картирования TIS, профилирование рибосом выявляет N-терминально расширенные и усеченные изоформы известных белков10,32. RIBO-seq также был адаптирован для изучения ко-трансляционного сворачивания белков14,21,24. Этот метод позволяет измерять скорости удлинения1,10,39 или скорость декодирования отдельных кодонов6 и помогает в разработке количественных моделей трансляции17. Метод профилирования рибосом также способен обеспечить механистическое понимание паузы рибосомы у бактерий7,15,17,сдвига кадра40,стоп-кодона через21,дефектов терминации/рециркуляции41,42 и рибосомных конформационных изменений33 у эукариот. RIBO-seq также был адаптирован для изучения влияния специфических трансдействующих факторов на трансляцию, таких как миРНК6 и РНК-связывающие белки у эукариот16,43. Однако важно признать, что экспериментальная конструкция и полученная разрешение RIBO-seq определяют объем информации, которая может быть извлечена из полученных данных секвенирования12.
Protocol
Representative Results
Discussion
Ключевой технической проблемой профилирования рибосом является необходимость быстрого ингибирования трансляции, чтобы захватить снимок рибосом на мРНК в определенном физиологическом состоянии, представляющих интерес. Для этого обычно используются ингибиторы трансляции, быстрый с?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
ALS хотел бы отметить финансовую поддержку EMBO Installation Grants IG 3914 и POIR. 04.04.00-00-3E9C/17-00 осуществляется в рамках программы First TEAM Фонда польской науки, совместно финансируемой Европейским Союзом в рамках Европейского фонда регионального развития.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
References
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

