Étude du trafic de protéines membranaires dans les cellules photoréceptrices de la drosophile à l’aide de protéines marquées eGFP
Summary
Ici, des méthodes non invasives sont décrites pour la localisation des protéines de la membrane photoréceptrice et l’évaluation de la dégénérescence rétinienne dans l’œil composé de la drosophile à l’aide de la fluorescence eGFP.
Abstract
Le trafic de protéines membranaires régule l’incorporation et l’élimination des récepteurs et des canaux ioniques dans la membrane plasmique. Ce processus est fondamentalement important pour la fonction cellulaire et l’intégrité cellulaire des neurones. Les cellules photoréceptrices de la drosophile sont devenues un modèle pour l’étude du trafic de protéines membranaires. Outre la rhodopsine, qui, lors de l’illumination, s’intériorise à partir de la membrane photoréceptrice et se dégrade, le canal ionique de type récepteur transitoire (TRPL) chez la drosophile présente une translocation dépendante de la lumière entre la membrane photoréceptrice rhabdomerale (où elle est située dans l’obscurité) et le corps cellulaire photorécepteur (vers lequel elle est transportée lors de l’illumination). Ce transport intracellulaire de TRPL peut être étudié de manière simple et non invasive en exprimant le TRPL marqué eGFP dans les cellules photoréceptrices. La fluorescence eGFP peut alors être observée soit dans le pseudopoche profond, soit par microscopie à immersion dans l’eau. Ces méthodes permettent la détection de la fluorescence dans l’œil intact et sont donc utiles pour les tests à haut débit et les criblages génétiques de mutants drosophiles défectueux dans la translocation TRPL. Ici, la préparation des mouches, les techniques microscopiques, ainsi que les méthodes de quantification utilisées pour étudier cette translocation déclenchée par la lumière de TRPL sont expliquées en détail. Ces méthodes peuvent également être appliquées pour des études de trafic sur d’autres protéines photoréceptrices de la drosophile , par exemple la rhodopsine. De plus, en utilisant des protéines rhabdomeral marquées eGFP, ces méthodes peuvent être utilisées pour évaluer la dégénérescence des cellules photoréceptrices.
Introduction
En délivrant et en éliminant les protéines vers et depuis la membrane plasmique, le trafic de protéines membranaires dans les neurones contrôle l’équipement de la membrane plasmique avec des récepteurs ainsi que des canaux ioniques et, par conséquent, régule la fonction neuronale. Une mauvaise régulation ou des défauts dans le trafic des protéines ont généralement des effets néfastes sur les cellules et entraînent une dégénérescence neuronale. Chez l’homme, cela peut provoquer des maladies neurodégénératives telles que la maladie d’Alzheimer et la maladie de Parkinson ou la rétinite pigmentaire1. Les photorécepteurs dans l’œil composé de Drosophila melanogaster sont devenus un système modèle in vivo pour l’étude du trafic de protéines membranaires2. Cela n’est pas seulement dû à la polyvalence génétique de la drosophile qui permet des criblages génétiques efficaces, mais aussi parce que tous les composants essentiels de la membrane photoréceptrice absorbant la lumière sont caractérisés en détail et que des techniques microscopiques efficaces sont disponibles qui peuvent être appliquées à l’œil de la mouche. Ces techniques sont au centre de cet article.
Dans les cellules photoréceptrices de la drosophile, la membrane plasmique apicale forme une pile dense de microvillosités le long d’un côté de la cellule, appelée rhabdomere. Les rhabdomeres des cellules photoréceptrices R1-6 sont disposées selon un motif trapézoïdal caractéristique tandis que les cellules photoréceptrices R7 et R8 forment un seul rhabdomere au centre de ce trapèze3. Le trafic de protéines membranaires est nécessaire pour un renouvellement régulé des protéines de la membrane rhabdomerale telles que la rhodopsine et les canaux ioniques TRP (potentiel récepteur transitoire) et TRPL (TRP-like) activés par la lumière afin d’assurer la quantité appropriée de ces protéines de phototransduction dans le rhabdomere. Les protéines de la membrane photoréceptrice sont synthétisées dans le réticulum endoplasmique et transportées via l’appareil de Golgi vers le rhabdomere. Après l’activation de la rhodopsine par la lumière, une molécule de rhodopsine peut soit être inactivée par absorption d’un deuxième photon, soit être retirée du rhabdomere par endocytose médiée par la clathrine. La rhodopsine endocytosée se dégrade dans le lysosome ou est recyclée en rhabdomere 4,5. Le canal ionique TRPL est également internalisé après activation de la cascade de phototransduction et subit une translocation dépendante de la lumière entre le rhabdomere (où il est situé lorsque les mouches sont maintenues dans l’obscurité) et un compartiment de stockage enrichi en ER dans le corps cellulaire (vers lequel il est transporté en quelques heures après l’illumination)6,7,8,9,10 . Contrairement à la rhodopsine endocytosée, seules de petites quantités de TRPL sont dégradées par la voie endolysosomale, et la majorité est stockée intracellulairement à la place et recyclée dans le rhabdomere lors de l’adaptation sombre6. TRPL peut ainsi être utilisé pour analyser le trafic déclenché par la lumière des protéines de la membrane plasmique. Les cellules photoréceptrices de la drosophile sont également utilisées pour étudier la dégénérescence neuronale. La dégénérescence des cellules photoréceptrices est souvent déterminée en évaluant la structure des rhabdomeres, qui se désintègrent à la suite de processus dégénératifs5.
Afin d’étudier la localisation subcellulaire de la TRPL et de la rhodopsine dans les cellules photoréceptrices ou la dégénérescence des cellules photoréceptrices, deux méthodes de microscopie à fluorescence qui diffèrent en termes de vitesse d’analyse et de résolution ont été appliquées ici. Une méthode très rapide et non invasive qui peut être utilisée pour les criblages génétiques mais avec une résolution spatiale limitée est la détection de fluorescence dans le pseudopieux profond (DPP). Le DPP est un phénomène optique d’yeux composés d’arthropodes dont l’origine géométrique a été expliquée en détail par Franceschini et Kirschfeld en 197111. En bref, sur plusieurs plans optiques sous la superposition de la rétine, des images de rhabdomeres d’ommatidies adjacentes peuvent être observées. Sur un plan focal passant par le centre de la courbure de l’œil, ces projections superposées forment une image qui ressemble à la disposition trapézoïdale des rhabdomeres dans un seul ommatidium seulement des ordres de grandeur plus grands. Ce phénomène peut également être observé indépendamment de l’expression exogène des protéines de fluorescence (par exemple TRPL::eGFP8), ce qui rend néanmoins le DPP plus facile à détecter (Figure 1A-A”)12. Une deuxième méthode non invasive est la microscopie par immersion dans l’eau qui repose sur l’imagerie de protéines marquées par fluorescence après avoir neutralisé optiquement l’appareil dioptrique des yeux avec de l’eau (Figure 1B-C”)12. En utilisant la méthode d’immersion dans l’eau, la quantité relative de TRPL::eGFP dans les rhabdomeres ou le corps cellulaire peut être évaluée quantitativement pour les cellules photoréceptrices individuelles. En outre, les protéines marquées par fluorescence non translocalisantes peuvent être utilisées pour évaluer l’intégrité rhabdomerale et pour déterminer l’évolution temporelle d’une dégénérescence potentielle de manière quantitative, comme décrit ici.
Bien que les enregistrements du DPP soient de loin les plus faciles et les plus rapides de ces méthodes, la résolution spatiale des données qu’ils génèrent est limitée. En outre, il existe de nombreuses raisons pour lesquelles un DPP peut être absent, qui ne sont pas nécessairement discernables par l’imagerie DPP elle-même. Étant donné que le DPP représente une somme de plusieurs ommatidies, des informations sur les cellules individuelles sont perdues. Ainsi, l’imagerie DPP à basse résolution joue un rôle important dans le dépistage d’un grand nombre de mouches, mais devrait généralement être suivie d’enregistrements à plus haute résolution par microscopie à immersion dans l’eau. Les micrographies à immersion dans l’eau permettent des interprétations sur les cellules individuelles, les défauts de développement, la morphologie des yeux, la mauvaise localisation des protéines ou la dégénérescence rétinienne ainsi que la quantification de ces effets. Le présent Protocole décrit ces deux techniques en détail.
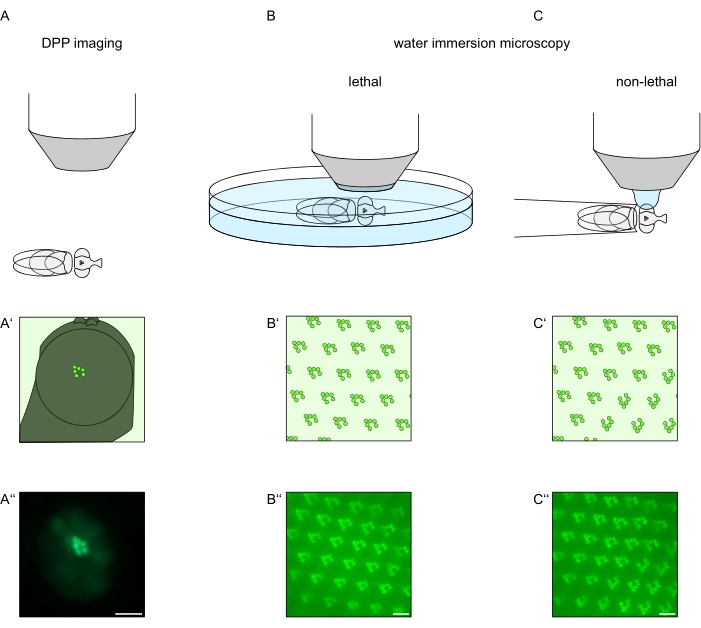
Figure 1 : Aperçu des variations microscopiques de l’œil de drosophile présentées dans ce protocole. Représentations schématiques et micrographies exemplaires de l’imagerie fluorescente par pseudopieux profond (DPP) (A-A”), de la microscopie par immersion dans l’eau létale (B-B”) des rhabdomeres fluorescents et de la microscopie à goutte d’eau non létale (C-C”) des rhabdomeres fluorescents. Barre d’échelle (A”): 100 μm. Barres d’échelle (B”–C”): 10 μm. La figure a été modifiée par rapport à la référence13. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocol
Representative Results
Discussion
L’applicabilité des protéines de fluorescence et la simplicité du dépistage par imagerie DPP et microscopie rétinienne par immersion dans l’eau se sont avérées efficaces par de nombreux groupes12. Des stratégies similaires à celles présentées ici ont été utilisées dans plusieurs tests génétiques pour détecter des défauts dans les niveaux d’expression de la rhodopsine, l’homéostasie, l’organisation rétinienne ou l’intégrité cellulaire à l’aide de Rh1::eGFP …
Divulgations
The authors have nothing to disclose.
Acknowledgements
Nous tenons à remercier nos étudiants chercheurs au fil des ans. En particulier, Nina Meyer, Sibylle Mayer, Juliane Kaim et Laura Jaggy, dont les données ont été utilisées dans ce protocole comme résultats représentatifs. La recherche de notre groupe présentée ici a été financée par des subventions de la Deutsche Forschungsgemeinschaft (Hu 839/2-4, Hu 839/7-1) à Armin Huber.
Materials
| 15 mL centrifuge tube | Greiner Bio-One | 188271 | |
| CO2 anaesthesia fly pad | Flystuff | 59-172 | |
| Cold light lamp (KL 1500 LCD) | Zeiss | ||
| Fiji/ImageJ | NIH | ||
| Fluorescence microscope with UV lamp, camera, filter set and software (AxioImager.Z1m, Axiocam 530 mono, 38 HE, ZEN2 blue edition) | Zeiss | ||
| Fluorescent tube (Lumilux T8, L 30W/840, 4000 K, G13) [1750 Lux, Ee470nm = 298 µW cm-2, Ee590nm = 215 µW cm-2] and [760 Lux, Ee470nm = 173 µW cm-2, Ee590nm = 147 µW cm-2] |
Osram | 4050300518039 | |
| Laboratory pipette (20-200 µL) | Eppendorf | ||
| Object slide | Roth | 0656.1 | |
| Petri dish (94 mm) | Greiner Bio-One | 633102 | |
| Pipette tips (200 µL) | Labsolute | 7695844 | |
| Plasticine (Blu-Tack) | Bostik | 30811745 | |
| Stereo microscope (SMZ445) | Nikon | ||
| Stereo microscope with UV lamp, camera, filer set and software (MZ16F, MC170 HD, GFP3, LAS 4.12) | Leica |
References
- Wang, X., Huang, T., Bu, G., Xu, H. Dysregulation of protein trafficking in neurodegeneration. Molecular Neurodegeneration. 9. 9, 31 (2014).
- Schopf, K., Huber, A. Membrane protein trafficking in Drosophila photoreceptor cells. European Journal of Cell Biology. 96 (5), 391-401 (2017).
- Hardie, R. C. The photoreceptor array of the dipteran retina. Trends in Neurosciences. 9, 419-423 (1986).
- Wang, T., Montell, C. Phototransduction and retinal degeneration in Drosophila. Pflügers Archiv – European Journal of Physiology. 454 (5), 821-847 (2007).
- Xiong, B., Bellen, H. J. Rhodopsin homeostasis and retinal degeneration: lessons from the fly. Trends in Neurosciences. 36 (11), 652-660 (2013).
- Bähner, M., et al. Light-regulated subcellular translocation of Drosophila TRPL channels induces long-term adaptation and modifies the light-induced current. Neuron. 34 (1), 83-93 (2002).
- Cronin, M. A., Lieu, M. -. H., Tsunoda, S. Two stages of light-dependent TRPL-channel translocation in Drosophila photoreceptors. Journal of Cell Science. 119, 2935-2944 (2006).
- Meyer, N., Joel-Almagor, T., Frechter, S., Minke, B., Huber, A. Subcellular translocation of the eGFP-tagged TRPL channel in Drosophila photoreceptors requires activation of the phototransduction cascade. Journal of Cell Science. 119, 2592-2603 (2006).
- Oberegelsbacher, C., Schneidler, C., Voolstra, O., Cerny, A., Huber, A. The Drosophila TRPL ion channel shares a Rab-dependent translocation pathway with rhodopsin. European Journal of Cell Biology. 90 (8), 620-630 (2011).
- Wagner, K., Smylla, T. K., Lampe, M., Krieg, J., Huber, A. Phospholipase D and retromer promote recycling of TRPL ion channel via the endoplasmic reticulum. Traffic. , (2021).
- Franceschini, N., Kirschfeld, K. Les phénoménes de pseudopupille dans l’oeil compose de Drosophila. Kybernetik. 9 (5), 159-182 (1971).
- Pichaud, F., Desplan, C. A new visualization approach for identifying mutations that affect differentiation and organization of the Drosophila ommatidia. Development. 128 (6), 815-826 (2001).
- Smylla, T. K., Wagner, K., Huber, A. Application of fluorescent proteins for functional dissection of the Drosophila visual system. International Journal of Molecular Sciences. 22 (16), (2021).
- Meyer, N. Mechanisms of the light-dependent translocation of the ion channel TRPL in the photoreceptors of Drosophila melanogaster: Dissertation for obtaining the academic degree Dr. rer. nat. University of Karlsruhe (TH). , (2005).
- Meyer, N., Oberegelsbacher, C., Dürr, T. D., Schäfer, A., Huber, A. An eGFP-based genetic screen for defects in light-triggered subcelluar translocation of the Drosophila photoreceptor channel TRPL. Fly. 2 (1), 36-46 (2008).
- Zheng, L., Carthew, R. W. Lola regulates cell fate by antagonizing Notch induction in the Drosophila eye. Mechanisms of Development. 125 (1-2), 18-29 (2008).
- Hibbard, K. L., O’Tousa, J. E. A role for the cytoplasmic DEAD box helicase Dbp21E2 in rhodopsin maturation and photoreceptor viability. Journal of Neurogenetics. 26 (2), 177-188 (2012).
- Huang, Y., Xie, J., Wang, T. A Fluorescence-based genetic screen to study retinal degeneration in Drosophila. PloS One. 10 (12), 0144925 (2015).
- Zhao, H., Wang, J., Wang, T. The V-ATPase V1 subunit A1 is required for rhodopsin anterograde trafficking in Drosophila. Molecular Biology of the Cell. 29 (13), 1640-1651 (2018).
- Xiong, L., et al. ER complex proteins are required for rhodopsin biosynthesis and photoreceptor survival in Drosophila and mice. Cell Death and Differentiation. 27 (2), 646-661 (2020).
- Zhao, H., Wang, T. PE homeostasis rebalanced through mitochondria-ER lipid exchange prevents retinal degeneration in Drosophila. PLoS Genetics. 16 (10), 1009070 (2020).
- Cerny, A. C., et al. The GTP- and phospholipid-binding protein TTD14 regulates trafficking of the TRPL ion channel in Drosophila photoreceptor cells. PLoS Genetics. 11 (10), 1005578 (2015).
- Richter, D. Structural and functional analyses of TRP ion channels in the photoreceptor cells of Drosophila melanogaster: Dissertation for obtaining the academic degree Dr. rer, nat. University of Karlsruhe (TH). , (2007).
- Gambis, A., Dourlen, P., Steller, H., Mollereau, B. Two-color in vivo imaging of photoreceptor apoptosis and development in Drosophila. Biologie du développement. 351 (1), 128-134 (2011).
- Hardie, R. C., Liu, C. -. H., Randall, A. S., Sengupta, S. In vivo tracking of phosphoinositides in Drosophila photoreceptors. Journal of Cell Science. 128 (23), 4328-4340 (2015).
- Chakrabarti, P., et al. A dPIP5K dependent pool of phosphatidylinositol 4,5 bisphosphate (PIP2) is required for G-protein coupled signal transduction in Drosophila photoreceptors. PLoS Genetics. 11 (1), 1004948 (2015).

