Ein "Dual-Addition"-Calciumfluoreszenz-Assay für das Hochdurchsatz-Screening von rekombinanten G-Protein-gekoppelten Rezeptoren
Summary
In dieser Arbeit wird ein intrazellulärer Hochdurchsatz-Calciumfluoreszenz-Assay für 384-Well-Platten beschrieben, um kleine Molekülbibliotheken auf rekombinanten G-Protein-gekoppelten Rezeptoren (GPCRs) zu screenen. Das Ziel, der Kinin-Rezeptor aus der Rinderpestzecke Rhipicephalus microplus, wird in CHO-K1-Zellen exprimiert. Dieser Assay identifiziert Agonisten und Antagonisten unter Verwendung der gleichen Zellen in einem “Dual-Addition”-Assay.
Abstract
G-Protein-gekoppelte Rezeptoren (GPCRs) stellen die größte Superfamilie von Rezeptoren dar und sind das Ziel zahlreicher Humanmedikamente. Das Hochdurchsatz-Screening (HTS) von zufälligen niedermolekularen Bibliotheken gegen GPCRs wird von der pharmazeutischen Industrie für die zielgerichtete Wirkstoffforschung eingesetzt. In dieser Studie wurde ein HTS eingesetzt, um neuartige niedermolekulare Liganden von wirbellosen-spezifischen Neuropeptid-GPCRs als Sonden für physiologische Untersuchungen von Vektoren tödlicher human- und veterinärmedizinischer Krankheitserreger zu identifizieren.
Der wirbellose Kinin-Rezeptor wurde als Ziel gewählt, weil er viele wichtige physiologische Prozesse bei Wirbellosen reguliert, einschließlich Diurese, Fütterung und Verdauung. Darüber hinaus ist die Pharmakologie vieler wirbelloser GPCRs schlecht oder gar nicht charakterisiert; Daher fügt die differentielle Pharmakologie dieser Rezeptorgruppen in Bezug auf die verwandten GPCRs in anderen Metazoen, insbesondere beim Menschen, Erkenntnisse über die Struktur-Wirkungs-Beziehungen von GPCRs als Superfamilie hinzu. Ein HTS-Assay wurde für Zellen in 384-Well-Platten entwickelt, um Liganden des Kinin-Rezeptors aus der Rinderpestzecke oder der südlichen Rinderzecke Rhipicephalus microplus zu entdecken. Der Zecken-Kinin-Rezeptor wurde in CHO-K1-Zellen stabil exprimiert.
Der Kinin-Rezeptor löst, wenn er durch endogene Kinin-Neuropeptide oder andere niedermolekulare Agonisten aktiviert wird, die Freisetzung von Ca2+ aus den Kalziumspeichern in das Zytoplasma aus. Dieser Calciumfluoreszenz-Assay in Kombination mit einem “Dual-Addition”-Ansatz kann funktionelle Agonisten- und Antagonisten-“Hit”-Moleküle in derselben Assayplatte nachweisen. Jeder Assay wurde unter Verwendung von Wirkstoffplatten durchgeführt, die eine Anordnung von 320 zufälligen kleinen Molekülen enthielten. Es wurde ein zuverlässiger Z’-Faktor von 0,7 erhalten, und drei Agonisten- und zwei Antagonisten-Hit-Moleküle wurden identifiziert, wenn das HTS eine Endkonzentration von 2 μM aufwies. Der hier beschriebene Calciumfluoreszenz-Assay kann angepasst werden, um andere GPCRs zu screenen, die die Ca2+ -Signalkaskade aktivieren.
Introduction
G-Protein-gekoppelte Rezeptoren (GPCRs), die von der Hefe bis zum Menschen vorhanden sind, stellen die größte Superfamilie von Rezeptoren in vielen Organismen dar1. Sie spielen eine entscheidende Rolle bei der Regulierung fast aller biologischen Prozesse bei Tieren. Es gibt 50-200 GPCRs im Genom von Arthropoden, was bedeutet, dass sie die größte Membranrezeptor-Superfamilie2 darstellen. Sie werden in sechs Hauptklassen, A-F, eingeteilt, basierend auf ihrer Sequenzähnlichkeit und ihren Funktionen3. GPCRs transduzieren verschiedene extrazelluläre Signale, wie z.B. die von Hormonen, Neuropeptiden, biogenen Aminen, Glutamat, Protonen, Lipoglykoproteinen und Photonen4. GPCRs koppeln an heterotrimere G-Proteine (Gα, Gβ und Gγ), um nachgeschaltete Signale zu übertragen. GPCRs, die an Gαs oder Gαi/o-Proteine gekoppelt sind, erhöhen bzw. senken die intrazellulären 3′, 5′-zyklischen Adenosinmonophosphat (cAMP)-Spiegel durch Aktivierung oder Hemmung der Adenylylcyclase. GPCRs, die an Gαq/11 gekoppelt sind, induzieren die Freisetzung von Kalzium aus den Kalziumspeichern des endoplasmatischen Retikulums, indem sie den Phospholipase C (PLC)-Inositol-1,4,5-triphosphat (IP3) -Weg aktivieren. GPCRs, die an Gα12/13 gekoppelt sind, aktivieren die RhoGTPase-Nukleotidaustauschfaktoren 5,6. GPCRs sind das Ziel von mehr als 50% der Humanarzneimittel und eines Akarizids, Amitraz4. Da GPCRs so unterschiedliche Signale übertragen, sind sie vielversprechende Ziele für die Entwicklung neuartiger Pestizide, die wirbellose physiologische Funktionen stören.
Das Ziel von HTS ist es, Hit-Moleküle zu identifizieren, die Rezeptorfunktionen modulieren können. HTS umfasst Assay-Entwicklung, Miniaturisierung und Automatisierung7. Arthropoden-Neuropeptid-GPCRs sind an den meisten physiologischen Funktionen wie Entwicklung, Häutung und Ekdysis, Ausscheidung, Energiemobilisierung und Fortpflanzungbeteiligt 4. Die meisten Neuropeptid-GPCRs von Arthropoden und Metazoen signalisieren durch die Calcium-Signalkaskade 2,6,8,9,10, wie z.B. in den myoinhibitorischen Peptid- und SIFamid-Rezeptoren der schwarzbeinigen Zecke Ixodes scapularis; Ihre Liganden sind in den Motilitätstests des Hinterdarms antagonistisch, wobei SIF eine Kontraktion auslöst und MIP sie hemmt11,12. Ein NPY-ähnlicher Rezeptor der Gelbfiebermücke, Aedes aegypti, reguliert die weibliche Wirtssuche13. Im Vergleich zu anderen alternativen Calcium-Mobilisierungsassays, wie dem Aequorin-Calcium-Biolumineszenz-Assay14, ist der Calcium-Fluoreszenz-Assay einfach durchzuführen, erfordert keine Transfektion anderer rekombinanter Calcium-Detektionsproteine und ist kostengünstig. Der Calcium-Fluoreszenz-Assay erzeugt ein verlängertes Signal im Vergleich zu dem schnellen kinetischen Signal, das im Aequorin-Calcium-Biolumineszenz-Assay14,15 erhalten wurde.
Im vorliegenden Beispiel wurde der Kinin-Rezeptor aus der Rinderpestzecke Rhipicephalus microplus rekombinant in der CHO-K1-Zelllinie exprimiert und für den Calciumfluoreszenz-Assay verwendet. Es gibt nur ein Kinin-Rezeptor-Gen in R. microplus; Der Rezeptor signalisiert über einen Gq-Protein-abhängigen Signalweg und löst den Ausfluss von Ca2+ aus Calciumspeichern in den intrazellulären Raum16 aus. Dieser Prozess kann durch ein Fluorophor nachgewiesen und quantifiziert werden, das bei der Bindung von Calciumionen ein Fluoreszenzsignal auslöst (Abbildung 1).
Der Kinin-Rezeptor ist ein wirbellosen-spezifischer GPCR, der zu den Klasse-A-Rhodopsin-ähnlichen Rezeptoren gehört. Kinin ist ein uraltes Signal-Neuropeptid, das in Mollusca, Crustacea, Insecta und Acari 4,17,18 vorkommt. Coleopteren (Käfer) fehlt das Kinin-Signalsystem; In der Mücke Aedes aegypti gibt es nur einen Kinin-Rezeptor, der drei Aedeskinine bindet, während Drosophila melanogaster einen Kinin-Rezeptor mit Drosokinin als einzigartigem Ligandenhat 19,20,21. Bei Wirbeltieren gibt es keine homologen Kinine oder Kininrezeptoren. Obwohl die genaue Funktion von Kinin bei Zecken unbekannt ist, zeigen die Kininrezeptor-RNAi-stillgelegten Weibchen von R. microplus eine signifikant reduzierte Fortpflanzungsfähigkeit22. Kinine sind pleotrope Peptide in Insekten. Bei Drosophila melanogaster sind sie sowohl am zentralen als auch am peripheren Nervensystembeteiligt 23, an der Präekdysis24, an der Nahrungsaufnahme25, am Stoffwechsel 26 und an den Schlafaktivitätsmustern 26,27 sowie an der Fortbewegung der Larven 28. Kinine regulieren die Kontraktion des Hinterdarms, die Diurese und die Fütterung der Mücke A. aegypti 29,30,31. Die Kininpeptide weisen ein konserviertes C-terminales Pentapeptid Phe-X1-X2-Trp-Gly-NH2 auf, das die minimal erforderliche Sequenz für die biologische Aktivität32 darstellt. Die Arthropodenspezifität, die geringe Größe des endogenen Liganden, die sie für kleinmolekulare Interferenzen empfänglich macht, und die pleiotropen Funktionen bei Insekten machen den Kininrezeptor zu einem vielversprechenden Ziel für die Schädlingsbekämpfung4.
Der “Dual-Addition”-Assay (Abbildung 2) ermöglicht die Identifizierung von Agonisten oder Antagonisten im selben HTS-Assay15. Es basiert auf einem “Dual-Addition”-Assay, der in der pharmazeutischen Industrie häufig für die Wirkstoffforschung verwendet wird33. Kurz gesagt, die erste Zugabe von Medikamenten in die Zellplatte ermöglicht die Identifizierung potenzieller Agonisten in der chemischen Bibliothek, wenn ein höheres Fluoreszenzsignal im Vergleich zur Anwendung der Lösungsmittelkontrolle detektiert wird. Nach 5 Minuten Inkubation mit diesen kleinen Molekülen wird ein bekannter Agonist (Kinin-Peptid) auf alle Vertiefungen aufgetragen. Diejenigen Vertiefungen, die zufällig einen Antagonisten von der Wirkstoffplatte erhielten, zeigen ein niedrigeres Fluoreszenzsignal bei der Agonistenzugabe im Vergleich zu den Kontrolltöpfen, die das Lösungsmittel bei der ersten Zugabe erhielten. Dieser Assay ermöglicht dann die Identifizierung potenzieller Agonisten und Antagonisten mit den gleichen Zellen. In einem Standard-HTS-Projekt würden diese Hit-Moleküle durch Dosis-Wirkungs-Assays und durch zusätzliche biologische Aktivitätsassays, die hier nicht gezeigt werden, weiter validiert.
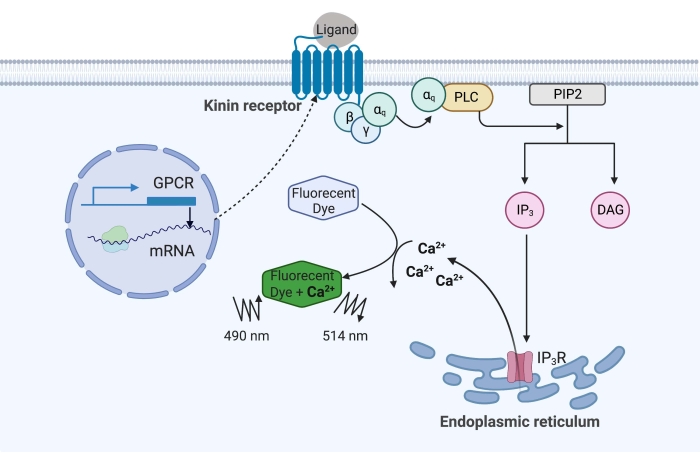
Abbildung 1: Illustration des Calciumfluoreszenz-Assay-Mechanismus. Das Gq-Protein löst den intrazellulären Kalzium-Signalweg aus. Der Kinin-Rezeptor (G-Protein-gekoppelter Rezeptor) wurde in CHO-K1-Zellen rekombinant exprimiert. Wenn der Agonistenligand an den Rezeptor bindet, aktiviert das mit dem Kininrezeptor assoziierte Gq-Protein PLC, das die Umwandlung eines PIP-2-Moleküls in IP3 und DAG katalysiert. IP 3 bindet dann an die IP3R auf der Oberfläche des endoplasmatischen Retikulums, was zur Freisetzung von Ca 2 + in das Zytoplasma führt, wo Ca2 + -Ionen an die Fluorophore binden und ein Fluoreszenzsignal auslösen. Das Fluoreszenzsignal kann durch Anregung bei 490 nm erhalten und bei 514 nm detektiert werden. Abkürzungen: GPCR = G-Protein-gekoppelter Rezeptor; PLC = phospholipase C; PIP2 = Phosphatidylinositol-4,5-bisphosphat; IP3 = Inositoltrisphosphat; DAG = Diacylglycerin; IP 3 R =IP-3-Empfänger. Erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
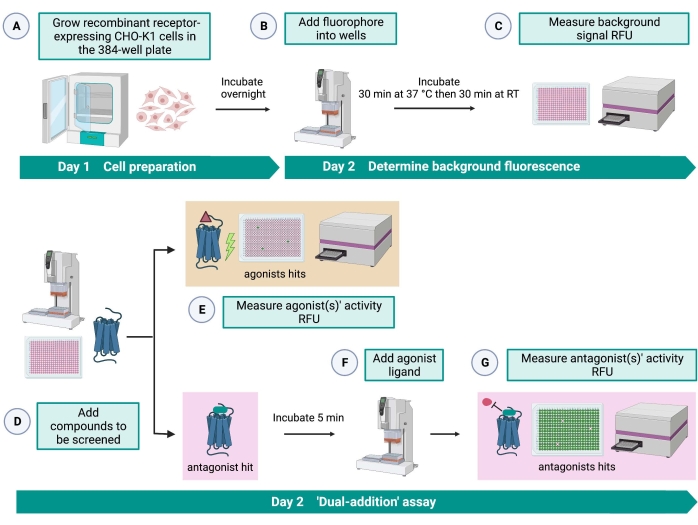
Abbildung 2: Der Arbeitsablauf für das Hochdurchsatz-Screening von kleinen Molekülen auf einem G-Protein-gekoppelten Rezeptor, der in CHO-K1-Zellen exprimiert wird. (A) Rekombinante CHO-K1-Zellen, die den Kinin-Rezeptor stabil exprimieren, wurden unter Verwendung eines Liquid-Handling-Systems (25 μL/Well) in die 384-Well-Platte (10.000 Zellen/Well) gegeben und in einem befeuchteten CO2 -Inkubator für 12-16 h inkubiert. ) Der Assay-Puffer, der den Fluoreszenzfarbstoff (25 μl/Well) enthielt, wurde unter Verwendung eines Liquid-Handling-Systems in die Zellplatte gegeben. Die Platte wurde für 30 min bei 37 °C für 30 min inkubiert und bei RT für weitere 30 min äquilibriert. (C) Das Hintergrundfluoreszenzsignal der Zellen in jeder Vertiefung wurde mit einem Plattenleser gemessen. (D) Arzneimittellösungen aus einer Bibliotheksplatte mit 384 Vertiefungen und ein Leerlösungsmittel (alle mit 0,5 μl/Well) wurden unter Verwendung eines Liquid-Handling-Systems in die zelluläre Assayplatte gegeben. (E) Die zellulären Calciumfluoreszenzreaktionen wurden unmittelbar nach der Zugabe der Arzneimittellösungen mit dem Plattenleser gemessen; Verbindung(en), die überdurchschnittliche Fluoreszenzsignale hervorruft, wurden als Agonisten-Treffer ausgewählt. Antagonisten-Treffer, die die GPCR blockieren (Symbol unten), wurden nach der Zugabe des Peptidagonisten während Schritt G aufgedeckt. (F) In der gleichen Testplatte wurde nach 5 min Inkubation der Zellen mit Screening-Verbindungen ein endogenes Agonistenpeptid Rhimi-K-1 (QFSPWGamid) des Zeckenkininrezeptors zu jeder Vertiefung (1 μM) gegeben. (G) Zelluläre Fluoreszenzreaktionen nach der Agonisten-Peptidaddition wurden vom Plattenleser sofort gemessen. Verbindung(en), die das Fluoreszenzsignal hemmen, wurden als Antagonisten-Treffer ausgewählt. Abkürzungen: GPCR = G-Protein-gekoppelter Rezeptor; RT = Raumtemperatur; RFU = relative Fluoreszenzeinheiten. Erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protocol
Representative Results
Discussion
Das Ziel von HTS ist es, Treffermoleküle durch das Screening einer großen Anzahl kleiner Moleküle zu identifizieren. Daher stellen die Ergebnisse dieses Beispiels nur einen kleinen Teil eines konventionellen HTS-Experiments dar. Darüber hinaus müssen die identifizierten Treffermoleküle in nachgeschalteten Assays validiert werden, wie z. B. einem dosisabhängigen Assay an derselben rekombinanten Zelllinie und an einer CHO-K1-Zelllinie, die nur den leeren Vektor trägt, was gleichzeitig durchgeführt werden kann, um …
Divulgations
The authors have nothing to disclose.
Acknowledgements
Diese Arbeit wurde durch den USDA-NIFA-AFRI Animal Health and Well-Being Award (Preisnummer 2022-67015-36336, PVP [Projektleiter]) und aus wettbewerbsfähigen Mitteln des Texas A&M AgriLife Research Insect Vector Diseases Grant Program (FY’22-23) an P.V.P. unterstützt. Die A.W.E.S.O.M.E.-Fakultätsgruppe des College of Agriculture and Life Sciences, TAMU, wird für die Hilfe bei der Bearbeitung des Manuskripts gedankt. Die Zusatztabelle S2 enthält Daten aus einer internen, zufälligen, kleinmolekularen Bibliothek, die aus dem Labor von Dr. James Sacchettini an der Texas A&M University und Texas A&M AgriLife Research stammt.
Materials
| 0.25% trypsin-EDTA | Gibco Invitrogen | 15050-065 | with phenol red |
| 0.4% trypan blue | MilliporeSigma | T8154 | liquid, sterile |
| 1.5 mL microcentrifuge tubes | Thermo Fisher | AM12400 | RNase-free Microfuge Tubes |
| 5 mL serological pipette | Corning | 29443-045 | Corning Costar Stripette individually wrapped |
| 10 mL serological pipette | Corning | 29443-047 | Corning Costar Stripette individually wrapped |
| 15 mL conical tubes | Falcon | 352196 | sterile |
| 20 µL filter tips | USA Scientifc Inc. | P1121 | sterile, barrier |
| 25 mL serological pipette | Corning | 29443-049 | Corning Costar Stripette individually wrapped |
| 50 mL conical tubes | Corning | 430828 | graduated, sterile |
| 150 mL auto-friendly reservior | Integra Bioscience | 6317 | sterile, individually wrapped for cell seeding in day 1 |
| 150 mL auto-friendly reservior | Integra Bioscience | 6318 | sterile, stacked, for loading dye in day 2 |
| 384/ 12.5 µL low retention tips | Integra Bioscience | 6405 | long, sterile filter |
| 384/ 12.5 µL tips | Integra Bioscience | 6404 | long, sterile filter |
| 384-well plate | Greiner | 781091 | CELLSTAR, clear polystyrene, µClear, Black/Flat |
| Aluminum plate seals | Axygen Scientific | PCR-AS-200 | polyester-based |
| Aluminum foil wrap | Walmart | ||
| Biosafty cabinet II | NuAire | NU-540-300 | |
| Cell counter | Nexcelom | AutoT4 | |
| cell counting slides | Nexcelom | SD-100 | 20 µL chamber |
| CO2 humidified incubator | Thermo Fisher | Forma Series II | |
| Desk Lamp | SunvaleeyTEK | RS1000B | |
| Dimethyl sulfoxide | MilliporeSigma | 276855 | anhydous, >99.9% |
| Drug plate | Corning | 3680 | |
| Dulbecco's phosphate-buffered saline | Corning | 21-031-CV | DPBS, 1x without calcium amd magnesium |
| Ethanol | Koptec | 2000 | |
| F-12K Nutrient Mixture | Corning | 45000-354 | (Kaighn's Mod.) with L-glutamine |
| Fetal bovine serum | Equitech-Bio | SFBU30 | |
| Fluorescent calcium assay kit | ENZO Lifescience | ENZ-51017 | 10×96 tests |
| G418 sulfate | Gibco Invitrogen | 10131-027 | Geneticin selective antibiotic 50 mg/mL |
| Hank's buffer | MilliporeSigma | 55037C | HBSS modified, with calcium, with magnesium, without phenol read |
| HEPES buffer | Gibco Invitrogen | 15630-080 | 1 Molar |
| HTS data storage plateform | CDD vault | https://www.collaborativedrug.com/ | |
| Liquid handling system | Integra Bioscience | Viaflo | 384/12.5 µL |
| Plate centrifuge | Thermo Fisher | Sorvall ST8 | |
| Plate reader | BMG technology | Clariostar | |
| Poly-D-lysine | MilliporeSigma | P6407 | |
| Rhimi-K-1 agonist peptide | Genscript | custom order | QFSPWGamide |
| T-75 flask | Falcon | 353136 |
References
- Hanlon, C. D., Andrew, D. J. Outside-in signaling – A brief review of GPCR signaling with a focus on the Drosophila GPCR family. Journal of Cell Science. 128 (19), 3533-3542 (2015).
- Liu, N., Li, T., Wang, Y., Liu, S. G-protein coupled receptors (GPCRs) in insects-A potential target for new insecticide development. Molecules. 26 (10), 2993 (2021).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nature Reviews Molecular Cell Biology. 3, 639-650 (2002).
- Pietrantonio, P. V., Xiong, C., Nachman, R. J., Shen, Y. G protein-coupled receptors in arthropod vectors: Omics and pharmacological approaches to elucidate ligand-receptor interactions and novel organismal functions. Current Opinion in Insect Science. 29, 12-20 (2018).
- Hilger, D., Masureel, M., Kobilka, B. K. Structure and dynamics of GPCR signaling complexes. Nature Structural & Molecular Biology. 25 (1), 4-12 (2018).
- Liu, N., Wang, Y., Li, T., Feng, X. G-protein coupled receptors (GPCRs): Signaling pathways, characterization, and functions in insect physiology and toxicology. International Journal of Molecular Sciences. 22 (10), 5260 (2021).
- Hansen, K. B., Bräuner-Osborne, H., Leifert, W. FLIPR® assays of intracellular calcium in GPCR drug discovery. G Protein-Coupled Receptors in Drug Discovery. , (2009).
- Bauknecht, P., Jekely, G. Large-scale combinatorial deorphanization of Platynereis neuropeptide GPCRs. Cell Reports. 12 (4), 684-693 (2015).
- Frooninckx, L., et al. Neuropeptide GPCRs in C. elegans. Frontiers in Endocrinology. 3, 167 (2012).
- Caers, J., et al. More than two decades of research on insect neuropeptide GPCRs: An overview. Frontiers in Endocrinology. 3, 151 (2012).
- Šimo, L., Koči, J., Park, Y. Receptors for the neuropeptides, myoinhibitory peptide and SIFamide, in control of the salivary glands of the blacklegged tick Ixodes scapularis. Insect Biochemistry and Molecular Biology. 43 (4), 376-387 (2013).
- Šimo, L., Park, Y. Neuropeptidergic control of the hindgut in the black-legged tick Ixodes scapularis. International Journal for Parasitology. 44 (11), 819-826 (2014).
- Liesch, J., Bellani, L. L., Vosshall, L. B. Functional and genetic characterization of neuropeptide Y-like receptors in Aedes aegypti. PLoS Neglected Tropical Diseases. 7 (10), 2486 (2013).
- Lu, H. L., Kersch, C. N., Taneja-Bageshwar, S., Pietrantonio, P. V. A calcium bioluminescence assay for functional analysis of mosquito (Aedes aegypti) and tick (Rhipicephalus microplus) G protein-coupled receptors. Journal of Visualized Experiments. (50), e2732 (2011).
- Xiong, C., Baker, D., Pietrantonio, P. V. The cattle fever tick, Rhipicephalus microplus, as a model for forward pharmacology to elucidate kinin GPCR function in the Acari. Frontiers in Physiology. 10, 1008 (2019).
- Holmes, S. P., Barhoumi, R., Nachman, R. J., Pietrantonio, P. V. Functional analysis of a G protein-coupled receptor from the Southern cattle tick Boophilus microplus (Acari: Ixodidae) identifies it as the first arthropod myokinin receptor. Insect Molecular Biology. 12 (1), 27-38 (2003).
- Cox, K. J., et al. Cloning, characterization, and expression of a G-protein-coupled receptor from Lymnaea stagnalis and identification of a leucokinin-like peptide, PSFHSWSamide, as its endogenous ligand. Journal of Neuroscience. 17 (4), 1197-1205 (1997).
- Dircksen, H., Kastin, A. J. Chapter 32 – Crustacean bioactive peptides. Handbook of Biologically Active Peptides (Second Edition). , 209-221 (2013).
- Halberg, K. A., Terhzaz, S., Cabrero, P., Davies, S. A., Dow, J. A. Tracing the evolutionary origins of insect renal function. Nature Communications. 6, 6800 (2015).
- Pietrantonio, P. V., Jagge, C., Taneja-Bageshwar, S., Nachman, R. J., Barhoumi, R. The mosquito Aedes aegypti (L.) leucokinin receptor is a multiligand receptor for the three Aedes kinins. Insect Molecular Biology. 14 (1), 55-67 (2005).
- Radford, J. C., Davies, S. A., Dow, J. A. Systematic G-protein-coupled receptor analysis in Drosophila melanogaster identifies a leucokinin receptor with novel roles. Journal of Biological Chemistry. 277 (41), 38810-38817 (2002).
- Brock, C. M., et al. The leucokinin-like peptide receptor from the cattle fever tick, Rhipicephalus microplus, is localized in the midgut periphery and receptor silencing with validated double-stranded RNAs causes a reproductive fitness cost. International Journal for Parasitology. 49 (3-4), 287-299 (2019).
- Nässel, D. R. Leucokinin and associated neuropeptides regulate multiple aspects of physiology and behavior in Drosophila. International Journal of Molecular Sciences. 22 (4), 1940 (2021).
- Kim, Y. -. J., et al. Central peptidergic ensembles associated with organization of an innate behavior. Proceedings of the National Academy of Sciences of the United States of America. 103 (38), 14211-14216 (2006).
- Al-Anzi, B., et al. The leucokinin pathway and its neurons regulate meal size in Drosophila. Current Biology. 20 (11), 969-978 (2010).
- Yurgel, M. E., et al. A single pair of leucokinin neurons are modulated by feeding state and regulate sleep-metabolism interactions. PLoS Biology. 17 (2), 2006409 (2019).
- Nässel, D. R., Zandawala, M. Recent advances in neuropeptide signaling in Drosophila, from genes to physiology and behavior. Progress in Neurobiology. 179, 101607 (2019).
- Okusawa, S., Kohsaka, H., Nose, A. Serotonin and downstream leucokinin neurons modulate larval turning behavior in Drosophila. Journal of Neuroscience. 34 (7), 2544-2558 (2014).
- Kersch, C. N., Pietrantonio, P. V. Mosquito Aedes aegypti (L.) leucokinin receptor is critical for in vivo fluid excretion post blood feeding. FEBS letters. 585 (22), 3507-3512 (2011).
- Kwon, H., et al. Leucokinin mimetic elicits aversive behavior in mosquito Aedes aegypti (L.) and inhibits the sugar taste neuron. Proceedings of the National Academy of Sciences of the United States of America. 113 (25), 6880-6885 (2016).
- Xiong, C., Baker, D., Pietrantonio, P. V. A random small molecule library screen identifies novel antagonists of the kinin receptor from the cattle fever tick, Rhipicephalus microplus (Acari: Ixodidae). Pest Management Science. 77 (5), 2238-2251 (2021).
- Torfs, P., et al. The kinin peptide family in invertebrates. Annals of the New York Academy of Sciences. 897 (1), 361-373 (1999).
- Ma, Q., Ye, L., Liu, H., Shi, Y., Zhou, N. An overview of Ca2+ mobilization assays in GPCR drug discovery. Expert Opinion on Drug Discovery. 12 (5), 511-523 (2017).
- Zhang, J. -. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Zhang, R., Xie, X. Tools for GPCR drug discovery. Acta Pharmacologica Sinica. 33 (3), 372-384 (2012).
- Offermanns, S., Simon, M. I. Gα15 and Gα16 couple a wide variety of receptors to phospholipase C. Journal of Biological Chemistry. 270 (25), 15175-15180 (1995).
- Murgia, M. V., et al. High-content phenotypic screening identifies novel chemistries that disrupt mosquito activity and development. Pesticide Biochemistry and Physiology. 182, 105037 (2022).
- Lismont, E., et al. Can BRET-based biosensors be used to characterize G-protein mediated signaling pathways of an insect GPCR, the Schistocerca gregaria CRF-related diuretic hormone receptor. Insect Biochemistry and Molecular Biology. 122, 103392 (2020).

