Un protocollo completo per Segmentazione manuale delle Strutture mediale del lobo temporale
Summary
The present work provides a comprehensive set of guidelines for manually tracing the medial temporal lobe (MTL) structures. This protocol can be applied to research involving structural and/or combined structural-functional magnetic resonance imaging (MRI) investigations of the MTL, in both healthy and clinical groups.
Abstract
Il presente documento descrive un protocollo completo per il tracciamento manuale del set di regioni cerebrali comprendente il lobo temporale mediale (MTL): amigdala, ippocampo, e le regioni parahippocampal associati (peririnale, entorinale, e parahippocampal corretta). A differenza di molti altri protocolli di tracciamento disponibili, di solito concentrandosi su alcune aree MTL (ad esempio, amigdala e / o ippocampo), la prospettiva di integrazione adottato dai presenti orientamenti tracciamento permette per una chiara localizzazione di tutte le sottoregioni MTL. Integrando le informazioni da una varietà di fonti, inclusi i protocolli di tracciabilità esistenti rivolti separatamente varie strutture MTL, rapporti istologici, e atlanti cerebrali, e con il complemento di materiali visivi illustrativo, l'attuale protocollo fornisce una guida precisa, intuitiva e conveniente per la comprensione del MTL anatomia. La necessità di tali linee guida il tracciamentoè sottolineata anche illustrando le possibili differenze tra i protocolli automatici e manuali di segmentazione. Questa conoscenza può essere applicato verso la ricerca che coinvolge non solo le indagini di risonanza magnetica strutturale, ma anche colocalization e il segnale fMRI estrazione strutturale-funzionale da ROI anatomicamente definiti, in gruppi sani e clinici simili.
Introduction
Il lobo temporale mediale (MTL), una zona putativo del più alto livello di integrazione delle informazioni sensoriali 1, è stato oggetto frequente di analisi mirate. Ad esempio, l'ippocampo e le aree parahippocampal associati sono stati ampiamente studiati nella ricerca di memoria 2-5. Inoltre, il ruolo dell'amigdala è stato spesso sottolineato in ricerca esaminando trasformazione emozione e interazioni emozione-cognizione 6-11. Recentemente, diverse regioni MTL hanno anche ricevuto l'attenzione nel settore emergente delle neuroscienze personalità, che collega la struttura e la funzione di queste e di altre regioni del cervello a variazione individuale in tratti di personalità 12. Valutare l'anatomia e la funzione delle strutture MTL può essere importante nel facilitare la diagnosi delle malattie degenerative in cui specifiche anomalie strutturali e funzionali possono verificarsi in diverse strutture MTL. Ad esempio, nella malattia di Alzheimer (AD), una significativatrofeo della corteccia entorinale e nell'ippocampo si osserva 13,14, e atrofia dell'ippocampo può prevedere il passaggio da deterioramento cognitivo lieve a 15 dC. Algoritmi automatici di segmentazione sono di recente diventati popolari per la segmentazione delle strutture corticali e sottocorticali, ma come con qualsiasi strumento, questi programmi inevitabilmente verificarsi errori in alcuni casi. In questi casi un ricercatore dovrebbe essere equipaggiata sia con le conoscenze e le linee guida per riconoscere i confini anatomici delle strutture MTL. La tendenza in letteratura esistente è stato quello di indirizzare le singole sottoregioni MTL 16-21, con molti protocolli che tendono a concentrarsi su ippocampo 16-19.
Diversamente dalla maggior parte degli orientamenti pubblicati disponibili per MTL tracing, l'attuale protocollo fornisce un insieme completo di linee guida che consentono chiara la localizzazione di tutte le sottoregioni MTL. Sono descritte le linee guida l'analisi per le seguenti strutture MTL: l'amigdala (AMY), l'ippocampo (HC), la corteccia peririnale (PRC), la corteccia entorinale (ERC), e la corteccia parahippocampal (PHC). L'AMY e HC sono tracciate in primo luogo, e sono poi seguiti dal giro paraippocampale (PHG) strutture. Si noti che il HC generico termine è usato qui per riferirsi alla formazione HC, che comprende l'HC corretto, il subiculum, e il segmento posteriore uncus 22-24. Si noti inoltre che il PHG può essere diviso in due segmenti, la porzione anteriore e la porzione posteriore. All'interno della parte anteriore del PHG, può essere ulteriormente suddivisa in PHG anteriore laterale e mediale, le cui aree corticali corrispondere alla RPC e del CER, rispettivamente. Il PHC, la zona corticale della porzione posteriore del PHG, corrisponde alla corteccia parahippocampal corretta. Per motivi di semplicità, useremo i termini RPC e CER per riferirsi a anteriore PHG laterale e mediale, e PHC per riferirsi al PHG posteriori. Il segmentation per ogni struttura inizia con una localizzazione di massima delle frontiere anteriori e posteriori, insieme ad altri punti di riferimento importanti, che viene poi seguita dal tracciato effettivamente compiuto slice-by-slice sul piano coronale, in un anterior-posterior/rostro-caudal direzione. In tutti i casi, le sezioni sagittali ed assiali sono strettamente monitorati per aiutare la localizzazione dei confini e punti di riferimento anatomici.
La necessità di tali orientamenti tracciamento è anche illustrata nelle figure che mostrano possibili differenze tra l'uscita di protocolli automatiche e manuali di segmentazione. Il vantaggio di un protocollo che descrive tutte le strutture MTL nel formato di visualizzazione corrente è che le variazioni di anatomia (ad esempio, il solco collaterale profondità [CS]) che potrebbe influenzare definizioni del bordo possono essere descritti nel contesto con l'anatomia circostante (ad esempio , la RPC e CER mediale e bordi laterali variano in posizione a seconda della profondità del CS 25 </sup>). Questo potrebbe non essere chiaro o comprensibile a un tracciante inesperto o un tracciante esperto che ripercorre solo singole strutture o separati, e la nostra conoscenza, un orientamento tale visivamente globale non esiste.
Il presente protocollo è una presentazione esplicita di linee guida utilizzate per MTL tracing in una precedente inchiesta che identifica contributi differenziali da sottoregioni MTL l'effetto memoria migliorando di emozione 26, adattato alle più alte immagini di risoluzione del cervello consentite dalle recenti sviluppi in risonanza magnetica strutturale (RM) . Il tracciato è illustrato su scansioni ottenute da un volontario sano (femmina, 24 anni), usando uno scanner 3T MR. Immagini anatomiche sono state acquisite come 3D MPRAGE (TR = 1.800 msec, TE = 2.26 msec; FOV = 256 x 256 mm, dimensione voxel = 1 x 0,5 x 0,5 mm) con un angolo di acquisizione parallela ad AC-PC. Se i dati immagine viene acquisita con un angolo di acquisizione differente, come l'orientamento obliquo, i dati devono essere regridded ad un parallelo o perpendicolarmente alla AC-PC, in modo tale che le descrizioni anatomiche limite tradurre in modo appropriato. Le immagini sono state poi tradotte in formato Nifti e l'ingresso in un software di segmentazione 27 per il tracciamento manuale. Dati di scansione utilizzati nel protocollo attuale è stato raccolto come parte di uno studio che è stato approvato dal Comitato Istituzionale, e il volontario fornito consenso scritto.
Attingendo informazioni dai vari protocolli di tracciabilità separate per queste strutture 18-22,28-31, nonché da analisi e atlanti anatomici 23,32,33, il presente protocollo presenta una serie completa di linee guida che indirizzano incongruenze nella letteratura esistente. Completato da materiali visivi di accompagnamento, questo lavoro si prevede di promuovere la chiara comprensione delle strutture MTL, e suscitare l'interesse della ricerca futura adozione segmentazione manuale, sia come metodo primario di MTL tracciato o come supplementaMetodo ry di segmentazione automatica. Fornendo una guida precisa, intuitiva e conveniente per comprendere l'anatomia MTL, questo protocollo aiuterà i ricercatori a identificare la posizione di tutti sottoregioni MTL, rispetto ai loro strutture vicine, anche se solo alcune strutture MTL sono specificamente mirati per le analisi. Questo non solo per aumentare la precisione di localizzazione, ma aiuterà anche rivelatori a prendere decisioni informate in caso di variazione morfologica, che è altamente probabile nel MTL. Queste linee guida possono essere applicati alla ricerca che implica indagini MRI strutturali e / o funzionali del MTL, comprese le analisi volumetriche e cervello rilevamento delle anomalie, nonché procedure di localizzazione delle funzionale, anatomica e analisi tractographic, in gruppi sani. Il presente protocollo potrebbe essere utilizzato anche per informare la segmentazione delle strutture MTL per i pazienti (ad esempio, pazienti con atrofia), se i principali punti di riferimento anatomici sono relativamente conservati. Tracing soggetto clinicadati s 'può richiedere tempo e impegno supplementare, a seconda della gravità della atrofia e / o cambiamenti anatomici.
E 'importante considerare la distinzione tra circonvoluzioni e cortecce al momento di definire il ROI. Anatomicamente, giro qui si riferisce sia materia bianca e materia grigia, mentre la corteccia si riferisce alla materia grigia solo. A seconda dell'uso previsto del ROI, segmentazioni potrebbero includere sostanza bianca o escluderlo.
Si consiglia il tracciato da eseguire in sequenza, sottostruttura da sottostruttura, un emisfero per volta. Alcuni pacchetti software 34 permettono di tracciare i confini delineati su una fetta per essere incollato su fette successive, una funzione che accelera il processo. E 'sempre consigliabile fare riferimento all'emisfero opporsi, se necessario, al fine di verificare la coerenza tra le due parti (ad esempio, per individuare punti di repere anatomici). In alternativa, tracciamento parallelo delle stesse strutture all'interno del due emisferos può anche essere eseguita. Indipendentemente dal fatto che il tracciato è sequenziale o parallelo, una volta che il processo è completo, i rivelatori devono ricontrollare il risultato finale e apportare le modifiche necessarie, facendo riferimento entrambi gli emisferi e viste multiple aereo. Seconda dell'esperienza del tracciante e la risoluzione dei dati di imaging, segmentazione manuale del MTL per dati soggetti sani può richiedere 8-10 ore o più, nel caso di un tracciante novizio, per 3-4 ore, in caso di un esperto.
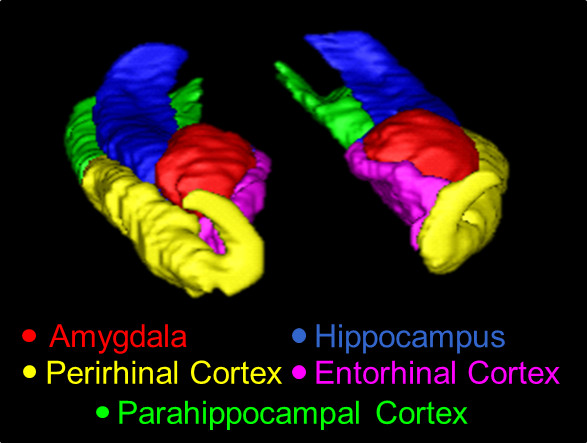
Panoramica Figura 1. Un 3D del MTL, tracciata utilizzando il presente protocollo. Strutture mostrato qui il AMY (rosso), l'HC (blu), la Repubblica popolare cinese (giallo), il CER (rosa), ed il PHC (verde) .
Protocol
Representative Results
Discussion
Tradizionalmente, la segmentazione manuale è stato considerato il gold standard da molti ricercatori. Tuttavia, una precisa definizione delle singole strutture è stata complicata dalla morfologia altamente variabile delle strutture MTL, e dai contrasti solito deboli MRI di queste strutture contro le aree di tessuto e non-neuronali neurali circostanti. Storicamente, ci sono stati descrizioni contrastanti in letteratura per alcune strutture MTL. In alcuni casi di segmentazione RPC, ad esempio, il solco collaterale è st…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
This research was supported by funds to FD. MM was supported by an IGERT Fellowship under National Science Foundation Grant No. 0903622. The authors wish to thank the Dolcos Lab members for assistance with data collection and preparation.
Materials
| ITK-SNAP | ITK-SNAP Team at University of Pennsylvania and University of Utah | ITK-SNAP v2.2 | |
| FSL | Functional Magnetic Resonance Imaging of the Brain (FMRIB) Analysis Group | FSL v4.1 | |
| 3T Siemens Trio MR Scanner | Siemens | 3T Trio |
Riferimenti
- Amaral, D. G. Introduction: what is where in the medial temporal lobe. Hippocampus. 9, 1-6 (1999).
- Squire, L. R., Zola-Morgan, S. The medial temporal lobe memory system. Science. 253 (5026), 1380-1386 (1991).
- Eichenbaum, H., Otto, T., Cohen, N. J. The hippocampus: what does it do. Behavioral & Neural Biology. 57 (1), 2-36 (1992).
- Henke, K., Buck, A., Weber, B., Wieser, H. G. Human hippocampus establishes associations in memory. Hippocampus. 7 (3), 249-256 (1997).
- Tulving, E., Markowitsch, H. J. Episodic and declarative memory: role of the hippocampus. Hippocampus. 8 (3), 198-204 (1998).
- Dolcos, F., Iordan, A. D., Dolcos, S. Neural correlates of emotion–cognition interactions: a review of evidence from brain imaging investigations. Journal of Cognitive Psychology. 23 (6), 669-694 (2011).
- Davidson, R. J., Irwin, W. The functional neuroanatomy of emotion and affective style. Trends in Cognitive Sciences. 3 (1), 11-21 (1999).
- Lindquist, K. A., Wager, T. D., Kober, H., Bliss-Moreau, E., Barrett, L. F. The brain basis of emotion: a meta-analytic review. The Behavioral and Brain Sciences. 35 (3), 121-143 (2012).
- Phan, K. L., Wager, T., Taylor, S. F., Liberzon, I. Functional neuroanatomy of emotion: a meta-analysis of emotion activation studies in PET and fMRI. Neuroimage. 16 (2), 331-348 (2002).
- Wager, T. D., Phan, K. L., Liberzon, I., Taylor, S. F. Valence, gender, and lateralization of functional brain anatomy in emotion: a meta-analysis of findings from neuroimaging. Neuroimage. 19, 513-531 (2003).
- Zald, D. H. The human amygdala and the emotional evaluation of sensory stimuli. Brain Research Reviews. 41, 88-123 (2003).
- DeYoung, C. G., Hirsh, J. B., Shane, M. S., Papademetris, X., Rajeevan, N., Gray, J. R. Testing predictions from personality neuroscience: brain structure and the big five. Psychological Science. 21 (6), 820-828 (2010).
- Visser, P. J., Verhey, F. R., Hofman, P. A., Scheltens, P., Jolles, J. Medial temporal lobe atrophy predicts Alzheimer’s disease in patients with minor cognitive impairment. Journal of Neurology, Neurosurgery, & Psychiatry. 72 (4), 491-497 (2002).
- Ezekiel, F., et al. Comparisons between global and focal brain atrophy rates in normal aging and Alzheimer disease. Alzheimer Disease & Associated Disorders. 18 (4), 196-201 (2004).
- de Leon, M. J., et al. Imaging and CSF studies in the preclinical diagnosis of Alzheimer’s disease. Annals of the New York Academy of Sciences. 1097, 114-145 (2007).
- Boccardi, M., et al. Survey of protocols for the manual segmentation of the hippocampus: preparatory steps towards a joint EADC-ADNI harmonized protocol. Journal of Alzheimer’s Disease. 26, 61-75 (2011).
- Konrad, C., Ukas, T., Nebel, C., Arolt, V., Toga, A. W., Narr, K. L. Defining the human hippocampus in cerebral magnetic resonance images-an overview of current segmentation protocols. Neuroimage. 47 (4), 1185-1195 (2009).
- Hasboun, D., et al. MR determination of hippocampal volume: comparison of three methods. American Journal of Neuroradiology. 17 (6), 1091-1098 (1996).
- Pantel, J., et al. A new method for the in vivo volumetric measurement of the human hippocampus with high neuroanatomical accuracy. Hippocampus. 10, 752-758 (2000).
- Entis, J. J., Doerga, P., Barrett, L. F., Dickerson, B. C. A reliable protocol for the manual segmentation of the human amygdala and its subregions using ultra-high resolution MRI. Neuroimage. 60 (2), 1226-1235 (2012).
- Goncharova, I. I., Dickerson, B. C., Stoub, T. R., deToledo-Morrell, L. MRI of human entorhinal cortex: a reliable protocol for volumetric measurement. Neurobiology of Aging. 22, 737-745 (2001).
- Watson, C., et al. Anatomic basis of amygdaloid and hippocampal volume measurement by magnetic resonance imaging. Neurology. 42 (9), 1743-1750 (1992).
- Duvernoy, H. The human hippocampus: functional anatomy, vascularization, and serial sections with MRI. Third Edition. , (2005).
- Amaral, D. G., Witter, M. P. The three-dimensional organization of the hippocampal formation: a review of anatomical data. Neuroscienze. 31 (3), 571-591 (1989).
- Insausti, R., et al. MR volumetric analysis of the human entorhinal, perirhinal, and temporopolar cortices. American Journal of Neuroradiology. 19 (4), 659-671 (1998).
- Dolcos, F., LaBar, K. S., Cabeza, R. Interaction between the amygdala and the medial temporal lobe memory system predicts better memory for emotional events. Neuron. 42 (5), 855-863 (2004).
- Yushkevich, P. A., et al. User-guided 3D active contour segmentation of anatomical structures: significantly improved efficiency and reliability. Neuroimage. 31 (3), 1116-1128 (2006).
- Bonilha, L., Kobayashi, E., Cendes, F., Li, M. L. Protocol for volumetric segmentation of medial temporal structures using high-resolution 3-D magnetic resonance imaging. Human Brain Mapping. 22 (2), 145-154 (2004).
- Bronen, R. A., Cheung, G. Relationship of hippocampus and amygdala to coronal MRI landmarks. Magnetic Resonance Imaging. 9 (3), 449-457 (1991).
- Pruessner, J. C., et al. Volumetry of temporopolar, perirhinal, entorhinal and parahippocampal cortex from high-resolution MR images: considering the variability of the collateral sulcus. Cerebral Cortex. 12 (12), 1342-1353 (2002).
- Pruessner, J. C., et al. Volumetry of hippocampus and amygdala with high-resolution MRI and three-dimensional analysis software: minimizing the discrepancies between laboratories. Cerebral Cortex. 10 (4), 433-442 (2000).
- Duvernoy, H. . The human brain: surface, three-dimensional sectional anatomy with MRI, and blood supply. Second Edition. , (1999).
- Amaral, D. G., Lavenex, P., et al. . in The hippocampus book. Hippocampal neuroanatomy. , (2006).
- Blaizot, X., et al. The human parahippocampal region: I. temporal pole cytoarchitectonic and MRI correlation. Cerebral Cortex. 20 (9), 2198-2212 (2010).
- Ding, S. -. L., Van Hoesen, G. W. Borders, extent, and topography of human perirhinal cortex as revealed using multiple modern neuroanatomical and pathological markers. Human Brain Mapping. 31 (9), 1359-1379 (2010).
- Ding, S. -. L., Van Hoesen, G. W., Cassell, M. D., Poremba, A. Parcellation of human temporal polar cortex: a combined analysis of multiple cytoarchitectonic, chemoarchitectonic, and pathological markers. The Journal of Comparative Neurology. 514 (6), 595-623 (2009).
- Frankó, E., Insausti, A. M., Artacho-Pérula, E., Insausti, R., Chavoix, C. Identification of the human medial temporal lobe regions on magnetic resonance images. Human Brain Mapping. 35 (1), 248-256 (2014).
- Lehmann, M., et al. Atrophy patterns in Alzheimer’s disease and semantic dementia: a comparison of FreeSurfer and manual volumetric measurements. Neuroimage. 49 (3), 2264-2274 (2010).
- Winterburn, J. L., et al. A novel in vivo atlas of human hippocampal subfields using high-resolution 3T magnetic resonance imaging. Neuroimage. 74, 254-265 (2013).
- Malykhin, N. V., Bouchard, T. P., Ogilvie, C. J., Coupland, N. J., Seres, P., Camicioli, R. Three-dimensional volumetric analysis and reconstruction of amygdala and hippocampal head, body and tail. Psychiatry research. Neuroimaging. 155 (2), 155-165 (2007).
- Patenaude, B., Smith, S. M., Kennedy, D. N., Jenkinson, M. A Bayesian model of shape and appearance for subcortical brain segmentation. Neuroimage. 56 (3), 907-922 (2011).
- Smith, S. M., et al. Advances in functional and structural MR image analysis and implementation as FSL. Neuroimage. 23, (2004).
- Woolrich, M. W., et al. Bayesian analysis of neuroimaging data in FSL. Neuroimage. 45, (2009).
- Smith, S. M. Fast robust automated brain extraction. Human Brain Mapping. 17 (3), 143-155 (2002).
- Morey, R. A., et al. A comparison of automated segmentation and manual tracing for quantifying hippocampal and amygdala volumes. Neuroimage. 45 (3), 855-866 (2009).
- Baldassano, C., Beck, D. M., Fei-Fei, L. Differential connectivity within the parahippocampal place area. Neuroimage. 75, 228-237 (2013).
- Shrout, P. E., Fleiss, J. L. Intraclass correlations: uses in assessing rater reliability. Psychological Bulletin. 86 (2), 420-428 (1979).
- Bland, J. M., Altman, D. G. A note on the use of the intraclass correlation coefficient in the evaluation of agreement between two methods of measurement. Computers in Biology and Medicine. 20, 337-340 (1990).
- Yushkevich, P. A., et al. A high-resolution computational atlas of the human hippocampus from postmortem magnetic resonance imaging at 9.4 T. Neuroimage. 44 (2), 385-398 (2009).

