상용 현미경에서 라이브 세포막에 분자 확산 법률에 빠른 형광 이미징
Summary
Spatial distribution and temporal dynamics of plasma membrane proteins and lipids is a hot topic in biology. Here this issue is addressed by a spatio-temporal image fluctuation analysis that provides conceptually the same physical quantities of single particle tracking, but it uses small molecular labels and standard microscopy setups.
Abstract
그것은 공간적 분포 및 지질과 단백질과 같은 막 성분의 움직임이 많은 세포 기능의 조절에 중요한 요인이라는 것을 분명하게 증가했다. 그러나, 빠른 역학과 관련된 작은 구조로, 매우 높은 시공간 해상도는 분자의 실제 동작을 잡을 필요합니다. 여기에서 우리는 높은 시공간 해상도의 살아있는 세포에 형광 표지 된 플라즈마 막 단백질과 지질의 역학을 공부 실험 프로토콜을 제시한다. 특히,이 방법은 각각의 분자를 추적 할 필요가 없지만, 그 막의 소정 영역에있는 모든 분자를 사용하는 인구의 동작을 계산한다. 시작점은 막에 소정 영역의 고속 촬상이다. 이후, 완전한 시공간적 자기 상관 함수는 예를 들어, 각각 2, 3, N 번 반복 시간 지연을 증가에서 획득 된 영상의 상관 관계를 계산한다. 이는 폭을 입증 할 수있다확산으로 인한 입자 운동의 함수로서 시간 지연을 증가에서 공간적 자기 상관 함수 증가 피크. 따라서, 자기 상관 함수의 일련의 피팅하는 것은 실제의 단백질이 여기에 평균 겉보기 변위 대 확산의 형태로 표시, 영상 (iMSD)에서 제곱 평균 변위를 추출 할 수있다. 이 나노 미터 정밀도로 단일 분자의 평균 역학의 양적보기를 얻을 수 있습니다. 표지 된 트랜스페린 수용체 (TFR) 및 ATTO488의 GFP 표지 된 변형을 사용하여 1 – 팔미 토일 -2 – 하이드 록시 SN -glycero-3-phosphoethanolamine (PPE)가 켜져 단백질 및 지질 확산 시공간 규제를 준수 할 수있다 마이크로 – 투 – 밀리 초의 시간 범위에서 μm의 막 크기의 영역.
Introduction
가수 Nicolson 의해 일본어 "유체 모자이크"모델부터 출발 세포 원형질막의 영상이 지속적으로 세포 골격과 지질 1,2 도메인의 새로운 역할을 포함하기 위해 지난 수십 년 동안 업데이트되었습니다.
첫번째 관찰은 막 단백질의 상당 부분이 3-5 움직입니다 (FRAP) 발표를 photobleaching에 후 형광 복구에 의해 얻어졌다. 이러한 선구적인 연구는 매우 유익하지만, FRAP의 설정의 상대적으로 가난한 공간 (마이크론)의 해상도와 시간 (초)으로 고통. 또한, 앙상블 평균 측정되는, FRAP는 단일 분자의 행동에 관한 정보를주는 없다.
이러한 맥락에서, 특히 가능성 (시 확산 처리 한 분자의 연구를 허용) 매우 밝은 태그와 단일 분자 라벨을 매우 성공적이었다. 특히, 밀어등 마이크로 척도, 쿠스미 크게 막 생리 6 액틴 기반 막 골격의 역할 인식에 기여 지질과 단백질 역학의 알 수없는 기능에 알. 얻은 액세스 단일 입자 추적 (SPT) 방식의 시간 해상도 , 7. 이러한 결과는 지질 단백질 및 액틴이 확산 기반 골격에 의해 조절되는 '피켓 울타리'모델을, 소위 생성. 그러나 여러 실험적인 문제 SPT에 의해 제공되는 정보의 양에 큰 권한을 부여하기 위해 해결되어야한다. 특히, 라벨 절차는 일반적으로 시스템에 표시된 종의 생산, 정제 및 도입 등 여러 단계로 구성되어있다. 또한, 라벨은 큰, 양자점 또는 금속 나노 입자와 같이 자주 서브 – 밀리 세컨드 타임 스케일 많은 경우에 회피 될 수없는 라벨 표적 분자의 가교에 도달해야한다. 마지막으로, 많은 궤적통계 학적 기준에 맞게 기록 될 필요와 병용 라벨의 저밀도는 추적을 허용하도록 요구된다.
SPT에 비해 이러한 단점을 많이 극복 형광 상관 분광학 (FCS)는, 분자 역학을 연구하는 매우 유망한 접근 방식을 나타냅니다. 사실, FCS는 일시적으로 형질 세포에 형광 단백질 태그 분자의 역학을 연구 할 수 있도록, 희미하고 조밀 한 레이블 또한 잘 작동합니다. 또한, 제한된 시간에 높은 통계에 도달한다. 마지막으로, 라벨의 "높은"밀도에도 불구하고 FCS는 단일 분자 정보를 제공합니다. 덕분에 모든 속성, FCS는 매우 간단 접근 방식을 나타냅니다 광범위 모델 막에서 라이브 세포 8-10에서 모두 지질과 단백질 역학을 연구하기 위해 적용되었습니다. 많은 다른 접근법은 분자 확산의 상세를 공개하는 FCS의 능력을 증가시키기 위해 제안되었다. 예를 들면, SH이었다자신의 다른 크기의 관찰 영역에 FCS를 수행하여 하나의 분자 운동 (11, 12)의 "FCS 확산 법"계몽 숨겨진 기능을 정의 할 수 있습니다. 크기가 변화되는 외에, 초점 영역도 13 중복 된 고속 카메라 (21, 22)와 선 (14-20) 또는 복합 따라 공간에서 움직였다. 이 '시공간'의 상관 관계에 접근하여, 여러 가지 막 구성 요소의 관련 생물학적 매개 변수를 정량적 모델 막 실제 생물들, 막 공간 조직에 따라서 항복 통찰력을 모두 설명했다.
그러나, 모든 FRAP 및 FCS 애플리케이션에서 설명한 바와 초점 영역의 크기를 극복 할 수없는 공간 해상도의 한계를 나타낸다. 몇몇 초 – 해상도 이미징 방법은 최근이 제한을 우회하기 위해 개발되어왔다. 일부는 같은 확률 광 재건 현미경 (STORM)로, 현지화 정밀도를 기반으로 <SUP> 23,24, photoactivation 현지화 현미경 (PALM) (25), 형광 PALM (FPALM) (26), 및 단일 입자 추적 PALM (sptPALM) 27 : 각 샷에 필요한 광자의 비교적 큰 양 그러나의 시간 해상도를 제한 적어도 몇 밀리 초에 이러한 방법, 따라서 생체 내에서의 적용을 방해.
대조적으로, 슈퍼 해상도 이미징을위한 유망한 대안은 공간적 자극 방출 고갈 방법 (STED 또는 가역 포화 광학 형광 전환 (RESOLFT)) (28, 29)과 형광 방출을 조절함으로써 열렸다. 이러한 접근 방식은 빠른 스캐닝 현미경 및 탐지 시스템을 사용하는 가능성 관찰 볼륨 성형 아니라 아래 회절 한계를 결합한다. 형광 변동 분석과 함께, STED 현미경 직접 지질 및 P의 나노 시공간 역학을 조사 할 수라이브 세포막 (30, 31)에서 roteins.
STED 기반 현미경 같은 물리량이 생균에 형광 표지 된 막 단백질 및 / 또는 지질의 역학 연구에 적합한 개질 시공간적 화상 상관 분광법 (STICS 32,33)의 방법에 의해 얻을 수있다 및 상용 현미경으로. 여기에 제시된 실험 프로토콜은 몇 가지 단계로 구성되어있다. 첫 번째는 막에 관심 영역의 고속 이미징을 필요로한다. 그 후, 화상의 생성 스택 평균 시공간 상관 함수를 계산하는데 사용된다. 상관 함수의 시리즈를 끼워 맞춤으로써, 분자 '확산 법칙은'명백한 확산 계수 (D 앱)의 형태로 영상에서 직접 획득 될 수있다 – – 평균 변위 플롯 대. 이 플롯은 매우 분자에 의해 탐구 환경에 따라 직접 실제 확산 모드를 인식 허용관심 지질 / 단백질.
이전 34와 같이 더 상세하게, 상기 취득 된 이미지 시리즈의 시공간적 자동 상관 함수가 매우 수집 된 이미지 시리즈에서 이동 분자 역학에 의존한다 (같은 추론이 라인 획득에 적용될 수 있음을주의하세요 공간에서 하나의 차원은) 생각입니다. 특히, 상관 함수를로 정의한다 :
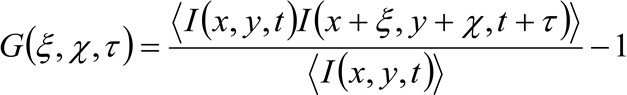 (1)
(1)
여기서 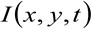 위치 (x)에서 측정 한 형광 강도를 나타내고, Y 및 시간 t에서,
위치 (x)에서 측정 한 형광 강도를 나타내고, Y 및 시간 t에서, ![]() 및
및 ![]() 나타내고 X의 거리각각 y 방향,
나타내고 X의 거리각각 y 방향, ![]() 타임 래그를 나타내고,
타임 래그를 나타내고, ![]() 평균을 나타냅니다. 이 함수는 다음과 같이 표현 될 수있다 :
평균을 나타냅니다. 이 함수는 다음과 같이 표현 될 수있다 :
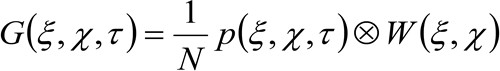 (2)
(2)
'N'은 관찰 영역에서 분자의 평균 수를 나타내고, ![]() 공간에서의 컨볼 루션 연산을 나타내고,
공간에서의 컨볼 루션 연산을 나타내고, 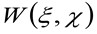 쓸모있는 허리의 자기 상관을 나타냅니다. 후자 인해 광 기록 / 셋업 단일 광자 에미 터가 공간 내에 확산되는 방법의 측정으로 해석 될 수있다 (소위 점 분포 함수, PSF, 유전자잘) 가우스 함수에 의해 근사 집회. 마지막으로,
쓸모있는 허리의 자기 상관을 나타냅니다. 후자 인해 광 기록 / 셋업 단일 광자 에미 터가 공간 내에 확산되는 방법의 측정으로 해석 될 수있다 (소위 점 분포 함수, PSF, 유전자잘) 가우스 함수에 의해 근사 집회. 마지막으로, 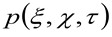 거리 입자를 찾을 확률을 나타낸다
거리 입자를 찾을 확률을 나타낸다 ![]() 및
및 ![]() 시간 지연 후에
시간 지연 후에 ![]() . 우리는 입자가 모든 방향으로 임의로 이동 순 플럭스가 존재하지되는 확산 역학을 고려하면,이 기능들은 일반적 차이가 움직이는 입자의 평균 제곱 변위 (MSD)로 식별 될 수 가우시안 함수로 근사 . 따라서, 상관 함수의 허리는 (로도 지칭
. 우리는 입자가 모든 방향으로 임의로 이동 순 플럭스가 존재하지되는 확산 역학을 고려하면,이 기능들은 일반적 차이가 움직이는 입자의 평균 제곱 변위 (MSD)로 식별 될 수 가우시안 함수로 근사 . 따라서, 상관 함수의 허리는 (로도 지칭 ![]() ), 입자 근골격계의 합 수단 허리로 정의 될 수 있고, 가우시안 피트에 의해 측정 될 수있다각 시간 지연에 대한 상관 함수의 팅. 측정 전 MSD는 움직이는 분자의 겉보기 확산을 계산하는데 사용될 수있다
), 입자 근골격계의 합 수단 허리로 정의 될 수 있고, 가우시안 피트에 의해 측정 될 수있다각 시간 지연에 대한 상관 함수의 팅. 측정 전 MSD는 움직이는 분자의 겉보기 확산을 계산하는데 사용될 수있다 ![]() 및 평균 변위
및 평균 변위 ![]() 로 :
로 :
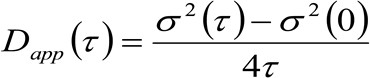 (3)
(3)
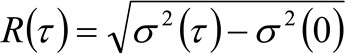 (4)
(4)
다음 섹션을 통해 독자를 안내 할 수 실험 구성에 대한 몇 가지 고려 사항. 선택적 상업용 TIR 형광을 이용하여, 우리는 전체 내부 반사 (TIR) 조명을 사용하는 살아있는 세포의 기저막에 형광체를 여기하기 위해 (TIRF) 현미경 (상세는 재료 부분에서 찾아 볼 수있다). 또한, 순서 번째 수집전자 형광 우리 고배율 대물 사용하며 (칩 16 μM의 화소의 물리적 크기) EMCCD 카메라 (100X NA 1.47, 높은 개구는 TIRF 조명 필요함). 100nm의 픽셀 크기에 도달하기 위해 우리는 1.6 부가 배율 렌즈를 적용. 후술하는 바와 같이, 1 밀리 초 이하의 시간 해상도는 100 나노 미터 이하로 적절히 빠른 막 지질의 역학을 설명하는 데 필요한 것. 이 시간 해상도에 도달하기 위해 우리는 카메라 (512 X 512)의 온 – 칩보다 작은이자 (ROI)의 영역을 선택해야한다. 이러한 방식으로, 카메라는 시간 해상도를 증가 선의 감소 된 수를 판독 할 것이다. 그러나,이 판독 정권 프레임 시간은 카메라 판독 칩에 노출 전하를 이동하는 데 필요한 512 X 512 픽셀 EMCCD 대 밀리 위해 통상 시간에 의해 제한 될 것이다. 이 한계를 이길하려면 새로운 기술은 와트, ROI 라인 만 대신 전체 프레임을 이동 할 수 있습니다(우리 EMCCD에서 자른 센서 모드라고 함)에 노출 칩 사이즈의 실질적인 감소 효과 제 i. 이 구성에 적용하려면, ROI 외부의 칩은 광로에 장착 슬릿 커플 적용되어야한다. 시간 해상도 다운 10-4 초로 설정이 덕분에 달성 될 수있다. '토론'섹션에 설명 된대로이 방법이 여러 가지 실험 설정과 결합 될 수 있다는 점 유의하시기 바랍니다.
방법은 데모 GFP- ATTO488 표지 모두 1 – 팔미 토일 -2 – 하이드 록시 -3 -glycero SN-phosphoethanolamine (ATTO488-PPE) 및 트랜스페린 수용체 GFP 표지 된 변형을 (사용함으로써, 생균에 제공 될 TFR). 이전에보고 된 바와 같이 30,35 ATTO488-PPE의 경우이 접근법은 성공적 주로 자유 확산을 나타내는 평균 변위의 함수로서 거의 일정한 D 애플리케이션을 복구 할 수있다. 대조적으로, TFR-GFP는 감소 D를 도시 <su부분적으로 한정된 확산 6 건의 평균 변위의 함수로서 B> 애플리케이션. 또한, 후자의 경우에는 로컬 확산 상수 및 막 평면에 많은 미크론 이상의 평균 협착 영역을 정량화 할 수있다.
Protocol
Representative Results
Discussion
단일 입자 추적 (SPT)은 분자 역학을 연구하는 가장 일반적인 전략 중 하나를 의미하며, 입자의 궤적을 측정의 큰 장점을 갖는다. 차례로 이것은 복잡한 시스템에도 몇몇 표지 입자의 거동을 프로빙한다. 그러나, 일반적 SPT 프로브와 매우 밝은 라벨 저밀도 필요 이러한 이점을 달성한다. 생산 시스템으로, 라벨 및 삽입의 복잡한 절차가 필요하다이 경우 : 특히, 높은 시간 해상도 (마이크로 초 범위) …
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
This work is supported in part by NIH-P41 P41-RRO3155 and NIH P50-GM076516 (grant to EG), and Fondazione Monte dei Paschi di Siena (grant to FB).
Materials
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| iXon Ultra 897 | Andor | DU-897U-CS0 | |
| Solis | Andor | ||
| CHO-K1 | ATCC | CCL-61 | |
| ATTO 488 labeled PPE | ATTO-TEC GmbH | AD 488-151 | |
| DOPE | Avanti Polar Lipids, Inc. | 850725 | |
| DOTAP | Avanti Polar Lipids, Inc. | 890890 | |
| 100X Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | |
| DMEM/F-12 | Gibco | 21331 | |
| FBS | Gibco | 10082147 | |
| HEPES | Gibco | 15630-106 | |
| PBS | Gibco | 10010-023 | |
| SimFCS 3.0 | Globals Software | the software can be downloaded here: http://www.lfd.uci.edu/globals/ | |
| DMI6000 with TIRF modulus | Leica | ||
| LAS AF | Leica | ||
| Lipofectamine 2000 | Lipofectamine | 11668019 | |
| Matlab | MathWork | ||
| Imagej | NIH | ||
| C-terminal GFP tagged Tranferrin Receptor | OriGene | RG200980 | |
| Agar | Sigma Aldrich | A5306 | |
| Chloroform | Sigma Aldrich | 528730 | |
| Latex beads, fluorescent yellow-green, 30 nm | Sigma Aldrich | L5155 | |
| SONICA Ultrasonic Cleaners | SOLTEC | ETH S3 | |
| Petri Dishes | Willco | GWSt-3522 | |
| Bio-Format importer for Matlab | http://www.openmicroscopy.org/site/support/bio-formats5/users/matlab/ | ||
| ICS-MatLab Tools | https://www.cellmigration.org/resource/imaging/software/ICSMATLAB_28-02-06.zip | ||
| Simulation by Matlab Tutorial | https://www.cellmigration.org/resource/imaging/icsmatlab/ICSTutorial.html | ||
| Simulation by SimFCS Tutorial | https://www.cellmigration.org/resource/imaging/ppt-pdf/RICS%20Simulations.ppt |
Riferimenti
- Engelman, D. M. Membranes are more mosaic than fluid. Nature. 438 (7068), 578-580 (2005).
- Vereb, G., et al. yet structured: The cell membrane three decades after the Singer-Nicolson model. Proc. Natl. Acad. Sci. U. S. A. 100 (14), 8053-8058 (1073).
- Ishihara, A., Hou, Y., Jacobson, K. The Thy-1 antigen exhibits rapid lateral diffusion in the plasma membrane of rodent lymphoid cells and fibroblasts. 84 (5), 1290-1293 (1987).
- Axelrod, D., et al. Lateral motion of fluorescently labeled acetylcholine receptors in membranes of developing muscle fibers. Proc. Natl. Acad. Sci. U. S. A. 73 (12), 4594-4598 (1976).
- Jacobson, K., Derzko, Z., Wu, E. S., Hou, Y., Poste, G. Measurement of the lateral mobility of cell surface components in single, living cells by fluorescence recovery after photobleaching. J. Supramol. Struct. 5 (4), 10-1002 (1976).
- Kusumi, A., et al. Paradigm shift of the plasma membrane concept from the two-dimensional continuum fluid to the partitioned fluid: high-speed single-molecule tracking of membrane molecules. Annu. Rev. Biophys. Biomol. Struct. 34, 351-378 (2005).
- Kusumi, A., Ike, H., Nakada, C., Murase, K., Fujiwara, T. Single-molecule tracking of membrane molecules: plasma membrane compartmentalization and dynamic assembly of raft-philic signaling molecules. Semin. Immunol. 17 (1), 3-21 (2005).
- Schwille, P., Korlach, J., Webb, W. W. Fluorescence correlation spectroscopy with single-molecule sensitivity on cell and model membranes. Cytometry. 36, 176-182 (1999).
- Gielen, E., et al. Diffusion of sphingomyelin and myelin oligodendrocyte glycoprotein in the membrane of OLN-93 oligodendroglial cells studied by fluorescence correlation spectroscopy. C. R. Biol. 328 (12), 1057-1064 (2005).
- Weiss, M., Hashimoto, H., Nilsson, T. Anomalous protein diffusion in living cells as seen by fluorescence correlation spectroscopy. Biophys. J. 84, 4043-4052 (2003).
- Wawrezinieck, L., Rigneault, H., Marguet, D., Lenne, P. F. Fluorescence correlation spectroscopy diffusion laws to probe the submicron cell membrane organization. Biophys. J. 89 (6), 4029-4042 (2005).
- Lenne, P. F., et al. Dynamic molecular confinement in the plasma membrane by microdomains and the cytoskeleton meshwork. EMBO J. 25 (14), 3245-3256 (2006).
- Ries, J., Schwille, P. Studying slow membrane dynamics with continuous wave scanning fluorescence correlation spectroscopy. Biophys. J. 91 (5), 1915-1924 (2006).
- Ruan, Q., Cheng, M. A., Levi, M., Gratton, E., Mantulin, W. W. Spatial-temporal studies of membrane dynamics: scanning fluorescence correlation spectroscopy (SFCS). Biophys. J. 87 (2), 1260-1267 (2004).
- Berland, K. M., So, P. T., Chen, Y., Mantulin, W. W., Gratton, E. Scanning two-photon fluctuation correlation spectroscopy: particle counting measurements for detection of molecular aggregation. Biophys. J. 71, 410-420 (1996).
- Heinemann, F., Betaneli, V., Thomas, F. A., Schwille, P. Quantifying lipid diffusion by fluorescence correlation spectroscopy: a critical treatise. Langmuir. 28 (37), 13395-13404 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Capturing directed molecular motion in the nuclear pore complex of live cells. Proc. Natl. Acad. Sci. U. S. A. 109 (25), 9863-9868 (2012).
- Sanchez, S. A., Tricerri, M. A., Gratton, E. Laurdan generalized polarization fluctuations measures membrane packing micro-heterogeneity in vivo. Proc. Natl. Acad. Sci. U. S. A. 109 (19), 7314-7319 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Fluorescence correlation spectroscopy of intact nuclear pore complexes. Biophys. J. 101 (4), 27-29 (2012).
- Di Rienzo, C., et al. Unveiling LOX-1 receptor interplay with nanotopography: mechanotransduction and atherosclerosis onset. Sci. Rep. 3, 10-1038 (2013).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophys. J. 95 (11), 5385-5398 (2008).
- Kannan, B., et al. Electron multiplying charge-coupled device camera based fluorescence correlation spectroscopy. Anal. Chem. 78 (10), 3444-3451 (2006).
- Jones, S. A., Shim, S. H., He, J., Fast Zhuang, X. three-dimensional super-resolution imaging of live cells. Nat. Methods. 8 (6), 499-508 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy. 3 (10), 793-795 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91 (11), 4258-4272 (2006).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Klar, T. A., Hell, S. W. Subdiffraction resolution in far-field fluorescence microscopy. Opt. Lett. 24 (14), 954-956 (1999).
- Eggeling, C., et al. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457 (7233), 1159-1162 (2009).
- Hedde, P. N., et al. Stimulated emission depletion-based raster image correlation spectroscopy reveals biomolecular dynamics in live cells. Nat. Commun. 4, .
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88 (5), 3601-3614 (2005).
- Brown, C. M., et al. Probing the integrin-actin linkage using high-resolution protein velocity mapping. J. Cell Sci. 119, 5204-5214 (2006).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proc. Natl. Acad. Sci. U. S. A. 110 (30), 12307-12312 (2013).
- Mueller, V., et al. STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys. J. 101 (7), 1651-1660 (2011).
- Kleusch, C., Hersch, N., Hoffmann, B., Merkel, R., Csiszar, A. Fluorescent lipids: functional parts of fusogenic liposomes and tools for cell membrane labeling and visualization. Molecules. 17 (1), 1055-1073 (2012).
- Ries, J., Chiantia, S., Schwille, P. Accurate determination of membrane dynamics with line-scan FCS. Biophys. J. 96 (5), 1999-2008 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49 (3), 141-164 (2007).
- Digman, M. A., et al. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89 (2), 1317-1327 (2005).
- Ritchie, K., et al. Detection of non-Brownian diffusion in the cell membrane in single molecule tracking. Biophys. J. 88 (3), 2266-2277 (2005).
- Voie, A. H., Burns, D. H., Spelman, F. A. Orthogonal-plane fluorescence optical sectioning: three-dimensional imaging of macroscopic biological specimens. J. Microsc. 170, 229-236 (1993).
- Huisken, J., Swoger, J., Del Bene, ., Wittbrodt, F., J, E. H., Stelzer, Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Wohland, T., Shi, X., Sankaran, J., Stelzer, E. H. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments. Opt. Express. 18 (10), 10627-10641 (2010).

