Generatie Fluorescent Protein Fusies<em> Candida</em> Species
Summary
PCR-gemedieerde genetische modificatie kan worden gebruikt voor fluorescent proteïne fusies in Candida soorten die visualisatie en kwantificering van gistcellen en eiwitten vergemakkelijkt genereren. Hierin presenteren we een strategie voor het construeren van een fluorescerend eiwit fusie (Eno1-FP) Candida parapsilosis.
Abstract
Candida species, gangbare kolonisators van de darmen en urogenitalis, de oorzaak van de meeste invasieve schimmelinfecties bij mensen. Aldus worden moleculaire en genetische instrumenten om de studie van de pathogenese mechanismen te vergemakkelijken. PCR-gemedieerde gen modificatie een eenvoudige en snelle manier van epitoop-gemerkte eiwitten te genereren om detectie te vergemakkelijken. In het bijzonder, fluorescerend eiwit (FP) fusies zijn krachtige instrumenten die visualisatie en kwantificering van zowel gistcellen en eiwitten mogelijk met behulp van fluorescentie microscopie en immunoblotting, respectievelijk. Plasmiden die FP coderende sequenties, samen met nutritionele merker genen dat de transformatie van Candida species te vergemakkelijken, zijn gegenereerd ten behoeve van FP constructie en expressie in Candida. Hierin presenteren we een strategie voor het construeren van een fusie-FP in een Candida species. Plasmiden die de nourseothricin resistantnce transformatie markergen (NAT1) samen met sequenties voor zowel groen, geel of kersen FP (GFP, YFP, mCherry) worden gebruikt tezamen met primers die genspecifieke sequenties in een polymerasekettingreactie (PCR) worden uitgevoerd, om FP cassette genereren . Deze genspecifieke cassette in staat om te integreren in het 3'-uiteinde van het corresponderende gen-locus via homologe recombinatie. Succesvolle in-frame fusie van het KP sequentie in het gen locus van belang genetisch gecontroleerd, gevolgd door analyse van fusie-eiwit expressie door microscopie en / of immuno-detectiemethoden. Bovendien, in het geval van sterk tot expressie gebrachte eiwitten, succesvolle fusies kunnen worden gescreend op de eerste plaats door fluorescentie beeldvorming technieken.
Introduction
Candida species zijn commensaal schimmels die de darm- en genito-urinaire stukken alle mensen koloniseren. Onder omstandigheden van immunodeficiëntie, zoals die optreden bij vroeggeboorte of immunosuppressieve effecten van behandelingen voor kanker, kan Candida species opportunistische pathogenen worden. Van de Candida species, Candida albicans is de meest voorkomende schimmel kolonisator en doet de meerderheid van invasieve schimmelinfecties. Andere Candida soorten zoals C. glabrata, C. parapsilosis, C. tropicalis en C. kruseii ook leiden tot ernstige infecties bij immuungecompromitteerde patiënten, met enkele vertonen intrinsieke weerstand tegen veelgebruikte anti-schimmel antibiotica zoals fluconazol en amfotericine B. Vandaar infecties met sommige van deze soorten worden vaker waargenomen, vooral bij patiënten die profylactisch behandeld met anti-schimmelmiddelen. Zelfs met de juiste en tijdige eennti-schimmel behandeling, invasieve Candida infecties blijven geassocieerd met significante morbiditeit en mortaliteit 1. Gezien het belang van Candida soorten menselijke gezondheid, is er een behoefte aan direct beschikbare moleculaire hulpmiddelen die de studie en onderzoek naar hun pathogenese mechanismen mogelijk.
Een belangrijk instrument waarmee onderzoekers te visualiseren en te kwantificeren microbiële cellen en de eiwitten die ze uiten is FP fusietechnologie. Polymerase kettingreactie (PCR) gemedieerde genetische modificatie, zoals beschreven in dit document, kan de constructie van fusies tussen FP sequenties en Candida eiwit coderende sequentie van belang in de genomische locus. Stabiele integratie van het construct vergemakkelijkt de analyse van eiwitexpressie en eiwit lokalisatie dynamiek. Plasmiden die FP sequenties geoptimaliseerd voor expressie in Candida albicans en die kunnen worden gebruikt in de PCR-gemedieerde gene modificatie strategie eerder geconstrueerd 2, 3, 4, 5. Plasmiden FP transformatie "cassettes": een FP-sequentie gekoppeld aan een nutritionele merker gen dat de transformatie van C. albicans en C. parapsilosis 2, 3, 4, 5, 6, 7 vergemakkelijkt. Momenteel verkrijgbare plasmiden bevatten verschillende selecteerbare merkergenen voeding (URA3, HIS1, ARG4) voor transformatie van auxotrofe stammen en dominante resistentie merker (NAT1), die transformatie van klinische stammen ontbreekt auxotrophies vergemakkelijkt. Bovendien plasmiden opties voor maximaal vier verschillende FP sequenties (groen [GFP], yellow [YFP], cyaan [GVB] en kersen [mCherry]) en ofwel een ADH1 terminatiesequentie voor de bouw van carboxy-terminus eiwitfusies of een promotersequentie voor de bouw van amino-terminus eiwitfusies. Primers zijn ontworpen met homologie met het plasmide-DNA rond de FP cassette. Bovendien, de primers bevatten ook 5'-extensie sequenties waarop homologie met het gist-gen van belang te hebben, wat de integratie van de cassette vergemakkelijkt in de genomische locus via homologe recombinatie (Figuur 1). Genspecifieke FP cassettes worden gegenereerd door PCR en vervolgens getransformeerd in Candida cellen die voor opname van DNA door behandeling met lithium acetaat.
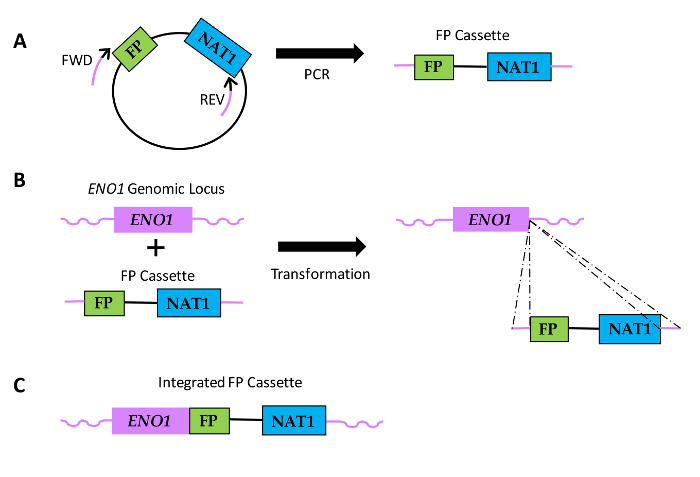
Figuur 1: Diagram van hoe FP reeks fusies worden gegenereerd in Candida soorten. (A) Plasmide DNA includes een FP-sequentie en een sequentie die codeert nourseothricin weerstand (NAT1). Relatieve locaties van de Vooruit (FWD) en achteruit (REV) primers worden getoond, met zwarte delen van de primers met vermelding van het gebied van homologie op het plasmide sequentie en de paarse delen aanduiding van de gen-specifieke homologie regio of primer extensie. (B) FP cassettes worden omgezet in Candida en te integreren binnen de ENO1 genomische locus via homologe recombinatie (stippellijnen). (C) Resulterend FP fusie sequentie aan het 3'-uiteinde van ENO1. Klik hier om een grotere versie van deze figuur te bekijken.
Hierin geven we een voorbeeld van eiwitfusie (Eno1-FP) constructies in Candida species. We maken gebruik van tagging plasmiden die de NAT1 transformatie marker-gen, samen met sequenties die coderen voor GFP, YFP, ofmCherry (Figuur 2). Deze plasmiden worden gebruikt in combinatie met primers in PCR te genspecifieke cassettes die fusie van KP vergemakkelijken het 3'-einde van ENO1 ontwikkeld, waardoor expressie van Eno1 gefuseerd aan KP bij de carboxy-terminus.
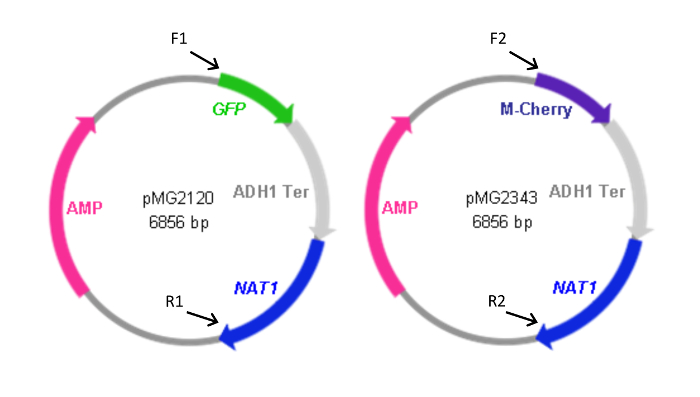
Figuur 2: Kaarten van FP-cassette die plasmiden. Voorwaartse (F) en reverse (R) primers gebruikt om de cassettes van de plasmiden worden aangeduid met de relatieve locatie van hun homologie met de plasmiden. Primer sequenties zoals vermeld in tabel 1. F1 en R1 werden gebruikt om de pYFP- NAT1 cassette genereren. Het plasmide dat de YFP- NAT1 cassette (pMG2263) is identiek aan pMG2120 behalve YFP in plaats van de GFP sequentie. Cassette maten: GFP-NAT1, 3,7 kbp; mCherry- NAT1, 3,2 kbp; YFP- NAT1, 3.7 kbp. Dit cijfer is gewijzigd ten opzichte van Gerami-Nejad, et al. 4 Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
Constructie van epitoop-gemerkte sequenties in Candida soort, volgens de PCR-gemedieerde genmodifcatie strategie hierboven beschreven kan worden samengevat als een drie-stappen. Eerst wordt een cassette door PCR dat codeert voor zowel de gewenste integratie en gebieden homoloog aan de plaats van insertie in het gist genoom. Ten tweede worden de gistcellen te transformeren chemisch competente met lithium acetaat en co-geïncubeerd met de cassette uit. Ten derde worden de cellen uitgeplaat op selectieve media om …
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Wij danken N. Dean voor het verstrekken van de oorspronkelijke mCherry FP-sequentie, M. Gerami-Nejad voor de constructie van plasmiden, B. Larson voor technische ondersteuning, en T. Heisel voor nuttig advies bij de ontwikkeling van dit project. JB werd gesteund door de European Research Council Geavanceerde Award 340.087 (RAPLODAPT). Microscopie en imaging systemen werden verstrekt door de Universiteit van Minnesota Kindergeneeskunde Stichting en de Universiteit van Minnesota Imaging Center.
Materials
| 100W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5ml, 1.5ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C ) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C ) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4°C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
Riferimenti
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. . Current Protocols in Molecular Biology. , (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).

