Genoom bewerken in zoogdieren cellijnen met behulp van CRISPR-Cas
Summary
CRISPR-Cas is een krachtige technologie om ingenieur van het complex genoom van planten en dieren. Hier, detail we een protocol bij het menselijk genoom met behulp van verschillende Cas enzym efficiënt te bewerken. We benadrukken belangrijke overwegingen en ontwerpparameters bewerken efficiëntie te optimaliseren.
Abstract
De geclusterde regelmatig interspaced korte palindromische herhaalt (CRISPR) systeem functioneert uiteraard in bacteriële adaptieve immuniteit, maar heeft met succes zijn voorzien voor genoom engineering in vele andere levende organismen. Meestal de wildtype CRISPR verbonden 9 (Cas9) of Cas12a endonuclease wordt gebruikt om specifieke locaties in het genoom, waarna de pauze van de double-stranded DNA is gerepareerd via het niet-homologe einde deelname aan (NHEJ) traject of de homologie geleide reparatie (klieven HDR) traject afhankelijk van of een donor-sjabloon is afwezig of respectievelijk presenteren. Tot op heden, is CRISPR systemen van verschillende bacteriesoorten gebleken kunnen bewerkingen genoom toe te passen in zoogdiercellen. Ondanks de schijnbare eenvoud van de technologie moeten meerdere ontwerpparameters echter worden overwogen, waardoor vaak gebruikers verbijsterd over het beste uit hun genoom bewerken experimenten te voeren. Hier beschrijven we een complete workflow van experimenteel design tot identificatie van cel klonen die gewenste DNA wijzigingen, met het doel van het vergemakkelijken van de succesvolle uitvoering van experimenten in zoogdieren cellijnen bewerken genoom dragen. We benadrukken belangrijke overwegingen voor gebruikers nota te nemen van, met inbegrip van de keuze van het CRISPR systeem, de lengte van de spacer en het ontwerp van een sjabloon van de donor single-stranded oligodeoxynucleotide (ssODN). Wij voorzien dat deze werkstroom zal zitten nuttig voor gene knockout studies, ziekte modellering van inspanningen, of de generatie van verslaggever cellijnen.
Introduction
De mogelijkheid om ingenieur van het genoom van elk levend organisme heeft vele biomedische en biotechnologische toepassingen, zoals de correctie van de ziekte-veroorzakende mutaties, bouw van accurate cellulaire modellen voor ziekte studies of generatie van landbouw gewassen met wenselijke eigenschappen. Sinds het begin van de eeuw, verschillende technologieën zijn ontwikkeld voor genoom engineering in zoogdiercellen, met inbegrip van meganucleases1,2,3, elektrolytisch vinger nucleasen4,5, of transcriptie activator-achtige effector nucleasen (TALENs)6,7,8,9. Echter zijn deze eerdere technologieën moeilijk te programma of vervelend te monteren, waardoor hun wijdverspreide goedkeuring in onderzoek en de industrie te belemmeren.
In de afgelopen jaren de geclusterde regelmatig interspaced korte palindromische herhaalt (CRISPR) – CRISPR-geassocieerde (Cas) systeem heeft ontpopt als een krachtige nieuwe genoom engineering technologie10,11. Oorspronkelijk een adaptieve immuunsysteem in bacteriën, is met succes ingezet voor het wijzigen van het genoom in planten en dieren, inclusief de mens. Een primaire reden waarom CRISPR-Cas heeft populariteit zo veel in zo’n korte tijd is dat het element dat de belangrijkste Cas-endonuclease, zoals Cas9 of Cas12a brengt (ook bekend als Cpf1), naar de juiste locatie in het genoom is gewoon een klein stukje chimeer één gids RN A (sgRNA), die is ontwerp eenvoudig en goedkoop te synthetiseren. Na wordt gerekruteerd om de doelsite, het Cas-enzym functioneert als een paar van moleculaire schaar en cleaves het afhankelijke DNA met haar RuvC, HNH of NOC domeinen12,13,14. De resulterende dubbele gestrande onderbreking (DSB) wordt later hersteld door de cellen via niet-homologe einde deelname aan (NHEJ) of de homologie geleide reparatie (HDR) traject. Bij gebrek aan een reparatie-sjabloon, wordt de DSB hersteld door de vergissing-geneigd NHEJ traject, die aanleiding tot pseudo-willekeurige invoeging of schrapping van nucleotiden (microdeleties) op de gesneden site geven kan, mogelijk veroorzaakt frameshift mutaties in eiwit-codeert genen. Echter in de aanwezigheid van een donor-sjabloon die de gewenste DNA wijzigingen bevat, wordt de DSB hersteld door het traject van de HDR high fidelity. Gangbare typen van donor sjablonen zijn single-stranded oligonucleotides (ssODNs) en plasmiden. De voormalige wordt meestal gebruikt als de beoogde DNA veranderingen klein (bijvoorbeeld wijziging van een enkelvoudige basenpaar), zijn terwijl de laatste meestal gebruikt wordt als men het wil invoegen een relatief lange reeks (bijvoorbeeld de codering opeenvolging van een groen fluorescent proteïne of GFP) in de locus van het doel.
De endonuclease activiteit van het eiwit van Cas vereist de aanwezigheid van een protospacer aangrenzende motief (PAM) op de target website15. De PAM van Cas9 is aan de 3′-eind van de protospacer, terwijl de PAM van Cas12a (ook wel Cpf1 genoemd) in plaats daarvan aan het einde 5′16. Het Cas-guide RNA complex is niet in staat om een DSB als de PAM afwezig17 is. Vandaar, de PAM legt een beperking op de genomic locaties waar een bepaalde Cas nuclease vermag klieven. Gelukkig, Cas nucleasen van verschillende bacteriesoorten vertonen meestal verschillende PAM eisen. Vandaar, door de integratie van verschillende systemen van de CRISPR-Cas in onze engineering toolbox, we kunnen uit te breiden het bereik van sites die in een genoom kunnen worden gericht. Bovendien kan een natuurlijke Cas enzym worden ontworpen of geëvolueerd om te herkennen van de alternatieve opeenvolgingen van het PAM, verdere uitbreiding van het toepassingsgebied van genomic doelstellingen toegankelijk voor manipulatie18,19,20.
Hoewel meerdere CRISPR-Cas systemen beschikbaar voor technische doeleinden genoom zijn, hebben de meeste gebruikers van de technologie vertrouwd voornamelijk op de Cas9 nuclease van Streptococcus pyogenes (SpCas9) om meerdere redenen. Ten eerste, het vereist een relatief eenvoudig NGG PAM, in tegenstelling tot veel andere certificeringsinstanties eiwitten die kunnen alleen in de aanwezigheid van meer complexe PAMs klieven. Ten tweede, het is de eerste Cas endonuclease om te worden geïmplementeerd in menselijke cellen21,22,23,24. Ten derde, SpCas9 is veruit het beste gekarakteriseerd enzym tot nu toe. Als een onderzoeker wil gebruiken een ander Cas nuclease, zou hij of zij vaak onduidelijk over de beste manier om het experiment en hoe goed de andere enzymen zal presteren in verschillende biologische contexten, in vergelijking met SpCas9 te ontwerpen.
Om duidelijkheid aan de relatieve prestaties van de verschillende systemen van de CRISPR-Cas, hebben we onlangs uitgevoerd een systematische vergelijking van vijf Cas enzym-SpCas9, het Cas9-enzym van Staphylococcus aureus (SaCas9), het enzym van de Cas9 van Neisseria meningitidis (NmCas9), het Cas12a-enzym van Acidaminococcus sp. BV3L6 (AsCas12a), en het enzym van de Cas12a van de Lachnospiraceae bacterie ND2006 (LbCas12a)25. We geëvalueerd voor een eerlijke vergelijking, de diverse Cas nucleasen met behulp van dezelfde set doel sites en andere experimentele omstandigheden. De studie ook afgebakend ontwerpparameters voor elk CRISPR-Cas-systeem, die als een nuttige referentie voor gebruikers van de technologie dienen zou. Hier, beter in staat stellen onderzoekers gebruik te maken van de CRISPR-Cas systeem, wij bieden een stapsgewijze protocol voor optimale genoom engineering met verschillende Cas9 en Cas12a enzymen (Zie Figuur 1). Het protocol omvat niet alleen experimentele details maar ook belangrijke ontwerpoverwegingen te maximaliseren van de kans op een succesvolle genoom engineering resultaat in zoogdiercellen.
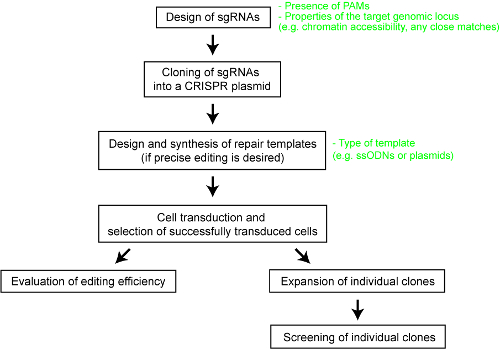
Figuur 1 : Een overzicht van de workflow voor het genereren van genoom bewerkt menselijke cellijnen. Klik hier voor een grotere versie van dit cijfer.
Protocol
Representative Results
Discussion
Het CRISPR-Cas-systeem is een krachtige, revolutionaire technologie om ingenieur van het genoom en de transcriptomes van planten en dieren. Talrijke bacteriesoorten bleken te bevatten van de CRISPR-Cas systemen, die potentieel kunnen worden aangepast voor genoom en transcriptome engineering doeleinden44. Hoewel de endonuclease van de Cas9 van Streptococcus pyogenes (SpCas9) was het eerste enzym met succes worden ingezet in menselijke cellen21,<sup class=…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
M.H.T. wordt ondersteund door een Agentschap voor wetenschap, technologie en onderzoek de gezamenlijke Raad Office grant (1431AFG103), een nationale Medical Research Council verlenen (OFIRG/0017/2016), National Research Foundation verleent (NRF2013-THE001-046 en NRF2013-THE001-093), een Ministerie van onderwijs Tier 1 grant (RG50/17 (S)), een startup verlenen vanaf Nanyang Technological University, en fondsen voor de internationale genetisch Engineering Machine (iGEM) concurrentie van Nanyang Technological University.
Materials
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | – | – | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
Riferimenti
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetica. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).

