التقييم الوظيفي لمتغيرات BRCA1 باستخدام محررات قاعدة CRISPR-بوساطة
Summary
الأشخاص الذين يعانون من طفرات BRCA1 لديهم خطر أعلى للإصابة بالسرطان ، مما يستدعي تقييمًا دقيقًا لوظيفة بدائل BRCA1. هنا، وصفنا بروتوكول للتقييم الوظيفي لمتغيرات BRCA1 باستخدام محررات قاعدة السيتوسين بوساطة CRISPR التي تمكن من C:G إلى T: تحويل في الخلايا الحية.
Abstract
وقد بحثت الدراسات الحديثة في المخاطر المرتبطة بالطفرات الجينية BRCA1 باستخدام أساليب تقييم وظيفية مختلفة مثل مقايسات المراسل الفلورية، مقايسات الخلايا الجذعية الجنينية، ومقايسات الحساسية العلاجية المستندة إلى الأدوية. على الرغم من أنها قد أوضحت الكثير من المتغيرات BRCA1، ترتبط هذه المقايسات التي تنطوي على استخدام بدائل BRCA1 أعرب عنها خارجيا مع قضايا فرط التعبير ولا يمكن تطبيقها على ما بعد النسخ. لحل هذه القيود، أبلغنا سابقا عن طريقة للتحليل الوظيفي لمتغيرات BRCA1 عبر محرر قاعدة السيتوسين بوساطة CRISPR التي تحفز استبدال النيوكليوتيدات المستهدفة في الخلايا الحية. باستخدام هذه الطريقة، حددنا المتغيرات التي لا تزال وظائفها غامضة، بما في ذلك c.-97C> T، c.154C> T، c.3847C> T، c.5056C> T، وc.4986+5G> A، وأكد أن محررات قاعدة CRISPR بوساطة هي أدوات مفيدة لإعادة تصنيف المتغيرات ذات الأهمية غير المؤكدة في BRCA1. هنا، ونحن وصف بروتوكول للتحليل الوظيفي للمتغيرات BRCA1 باستخدام محرر قاعدة cytosine مقرها CRISPR. ويوفر هذا البروتوكول مبادئ توجيهية لاختيار المواقع المستهدفة، والتحليل الوظيفي وتقييم المتغيرات BRCA1.
Introduction
سرطان الثدي نوع 1 الجينات القابلية للحساسية(BRCA1) هو معروف على نطاق واسع الجين القامع الورم. لأن جين BRCA1 يرتبط بإصلاح تلف الحمض النووي ، فإن الطفرات في هذا الجين ستؤدي إلى خطر أكبر من تطور السرطان في الفرد1. سرطانات الثدي, المبيض, البروستاتا, وسرطان البنكرياس ترتبط بالطفرات الموروثة فقدان وظيفة (LOF) من الجين BRCA1 2. التقييم الوظيفي وتحديد المتغيرات BRCA1 قد تساعد في الوقاية من الأمراض المختلفة وتشخيصها. لمعالجة وظيفة من المتغيرات BRCA1، وقد تم تطوير عدة طرق وتستخدم على نطاق واسع للتحقيق في الإمراضية من المتغيرات BRCA1 مثل المقايسات الخلايا الجذعية الجنينية البقاء، المقايسات مراسل الفلورسنت، والعلاجية المخدرات القائمة على الحساسية المقايسات3،4،5،6. على الرغم من أن هذه الطرق قد قيمت وظيفة الكثير من المتغيرات BRCA1، والأساليب التي تنطوي على بدائل BRCA1 أعرب عنها خارجيا تشكل قيودا من حيث فرط التعبير التي قد تؤثر على تنظيم المصب، والجرعة الجينية، والبروتين للطي7. وعلاوة على ذلك ، لا يمكن تسخير هذه المقايسات لتنظيم ما بعد الوصف مثل الربط مرنا ، واستقرار نسخة ، وتأثير المنطقة غير المترجمة8،9.
CRISPR-Cas9 نظام تمكن من تحرير الجينوم المستهدفة في الخلايا الحية والكائنات الحية10. من خلال RNA دليل واحد، يمكن أن تحفز Cas9 فواصل مزدوجة حبلا (DSBs) في الحمض النووي الكروموسومات في loci الجينوم محددة من أجل تنشيط مسارين إصلاح الحمض النووي: غيرhomologous غير عرضة للخطأ نهاية الانضمام (NHEJ) مسار وإصلاح homology خالية من الأخطاء الموجهة (HDR) مسار11. تقرير التنمية البشرية هو آلية دقيقة لإصلاح; ومع ذلك، DSBs الناجمة عن نيكلياز Cas9 لHDR غالباً ما يؤدي إلى إدخال غير المرغوب فيها والحذف (indel) طفرة. بالإضافة إلى ذلك، فإنه يحتاج إلى قوالب الحمض النووي المانحة متجانسة لإصلاح تلف الحمض النووي ولها كفاءة منخفضة نسبيا. في الآونة الأخيرة، وقد تم دمج Cas9 نيكاسي (nCas9) مع المجالات cytidine deaminase لاستهداف C:G إلى T:A استبدالات، دون الحاجة إلى قوالب الحمض النووي متماثل والحمض النووي كسر حبلا مزدوج12،13،14،15. باستخدام محرر قاعدة cytosine ، وضعنا طريقة جديدة للتحليل الوظيفي للبدائل BRCA116.
في هذه الدراسة، استخدمنا كريسبر بوساطة محرر قاعدة السيتوسين، BE314، مما يدفع كفاءة C:G إلى T:A الطفرات نقطة، لتنفيذ التقييم الوظيفي للمتغيرات BRCA1 وتحديد بنجاح وظائف عدة متغيرات BRCA1 (الشكل 1).
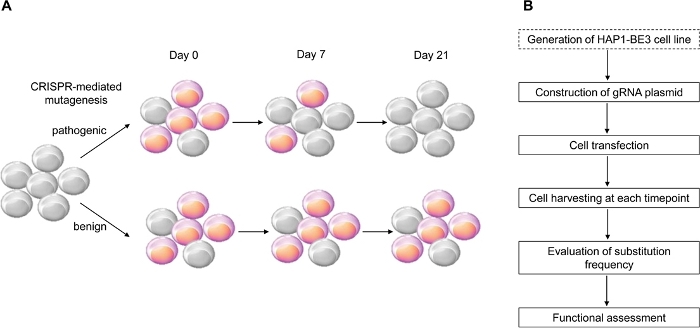
الشكل 1: نظرة عامة على سير العمل للتقييم الوظيفي. (A) التخطيطي تبين التقييم الوظيفي لBRCA1. لأن LOF من BRCA1 يؤثر على صلاحية الخلية، عندما طفرة BRCA1 هو المسببة للأمراض، تموت الخلايا كما يزيد عدد مرور. (ب) مراحل التقييم الوظيفي لـ BRCA1. مربع منقط اختياري. ويمكن استبداله بtrans-transfection من GRNA التعبير عن و BE3 التعبير عن dna plasmids. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
Representative Results
Discussion
يصف هذا البروتوكول طريقة بسيطة للتقييمات الوظيفية لمتغيرات BRCA1 باستخدام محرر قاعدة السيتوسين المتأملة CRISPR. ويصف البروتوكول أساليب تصميم الـ gRNAs عند موضع الهدف وبناء الـ DNAs البلازميد التي يتم التعبير عنها منها. المحررات قاعدة Cytosine حث تحويل النيوكليوتيدات في نافذة نشطة (في حالة BE3, الن?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
وقد تم دعم هذا العمل من قبل المؤسسة الوطنية للبحوث في كوريا (المنح 2017M3A9B4062419، 2019R1F1A1057637، و 2018R1A5A2020732 إلى Y.K.).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
Riferimenti
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Ricerca sul cancro. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

