Évaluation fonctionnelle des variantes BRCA1 à l’aide d’éditeurs de base à base de CRISPR
Summary
Les personnes atteintes de mutations BRCA1 ont un risque plus élevé de développer un cancer, ce qui justifie une évaluation précise de la fonction des variantes BRCA1. Ci-après, nous avons décrit un protocole pour l’évaluation fonctionnelle des variantes BRCA1 à l’aide d’éditeurs de base de cytosine à base crispr- médiatisés qui permettent la conversion ciblée de C:G à T:A dans les cellules vivantes.
Abstract
Des études récentes ont étudié les risques associés aux mutations du gène BRCA1 à l’aide de diverses méthodes d’évaluation fonctionnelles telles que les essais de reporter fluorescent, les essais de viabilité des cellules souches embryonnaires et les tests thérapeutiques de sensibilité à base de médicaments. Bien qu’ils aient clarifié beaucoup de variantes BRCA1, ces analyses impliquant l’utilisation de variantes BRCA1 exprimées exogènement sont associées à des problèmes de surexpression et ne peuvent pas être appliquées à la régulation post-transcriptionnelle. Pour résoudre ces limitations, nous avons précédemment rapporté une méthode pour l’analyse fonctionnelle des variantes BRCA1 par l’intermédiaire de l’éditeur de base de cytosine CRISPR-mentté qui induisent la substitution ciblée de nucléotide dans les cellules vivantes. En utilisant cette méthode, nous avons identifié des variantes dont les fonctions restent ambiguës, y compris c.-97C>T, c.154C>T, c.3847C>T, c.5056C>T, et c.4986+5G>A, et a confirmé que les éditeurs de base crispr-médiatisés sont des outils utiles pour reclassifier les variantes d’importance incertaine dans BRCA1. Ici, nous décrivons un protocole pour l’analyse fonctionnelle des variantes BRCA1 utilisant l’éditeur de base de cytosine crispr-basé. Ce protocole fournit des lignes directrices pour la sélection des sites cibles, l’analyse fonctionnelle et l’évaluation des variantes BRCA1.
Introduction
Le gène de susceptibilité de type 1 du cancer du sein (BRCA1) est un gène suppresseur de tumeur largement connu. Puisque le gène BRCA1 est lié à la réparation des dommages d’ADN, les mutations dans ce gène conduirait à un plus grand risque de développement de cancer dans unindividu 1. Les cancers du sein, de l’ovaire, de la prostate et du pancréas sont liés à des mutations héréditaires de perte de fonction (LOF) du gène BRCA1 2. L’évaluation fonctionnelle et l’identification des variantes BRCA1 peuvent aider à prévenir et à diagnostiquer les diverses maladies. Pour répondre à la fonction des variantes BRCA1, plusieurs méthodes ont été développées et largement utilisées pour étudier la pathogénie des variantes BRCA1 telles que les essais de viabilité des cellules souches embryonnaires, les essais de reporter fluorescent et les essais thérapeutiques de sensibilité à base demédicaments 3,4,5,6. Bien que ces méthodes aient évalué la fonction d’un grand nombre de variantes BRCA1, les méthodes impliquant des variantes BRCA1 exprimées exogènement posent des limites en termes de surexpression qui pourraient affecter la régulation en aval, la posologie des gènes et le pliage desprotéines 7. En outre, ces analyses ne peuvent pas être exploitées au règlement posttranscriptionnel tel que l’épissage d’ARNm, la stabilité de transcription, et l’effet de la régionnon traduite 8,9.
Le système CRISPR-Cas9 permet l’édition ciblée du génome dans les cellules vivantes et lesorganismes 10. Grâce à un ARN à guide unique, Cas9 peut induire des ruptures à double brin (DSB) dans l’ADN chromosomique à des loci génomiques spécifiques afin d’activer deux voies de réparation de l’ADN : la voie de jointage final non homologue sujette aux erreurs (NHEJ) et la voie de réparation dirigée par homologie sans erreur (HDR)11. HDR est un mécanisme de réparation précis; cependant, les DSBs induits par la nucléase de Cas9 pour HDR ont souvent comme conséquence l’insertion et la mutation non désirées d’insertion et de suppression (indélébile). En outre, il a besoin de modèles d’ADN de donneur homologue pour réparer les dommages causés par l’ADN et a une efficacité relativement faible. Récemment, Cas9 nickase (nCas9) ont été fusionnés avec des domaines deaminase de cytidine pour cibler les substitutions C:G à T:A, sans avoir besoin de modèles d’ADN homologues et de ruptures de double brin d’ADN12,13,14,15. En utilisant l’éditeur de base de cytosine, nous avons développé une nouvelle méthode pour l’analyse fonctionnelle des variantes BRCA116.
Dans cette étude, nous avons utilisé l’éditeur de base de cytosine crispr-négocié, BE314, qui induit des mutations efficaces de C:G à T:A point, pour mettre en œuvre l’évaluation fonctionnelle des variantes BRCA1 et a identifié avec succès les fonctions de plusieurs variantes BRCA1 (Figure 1).
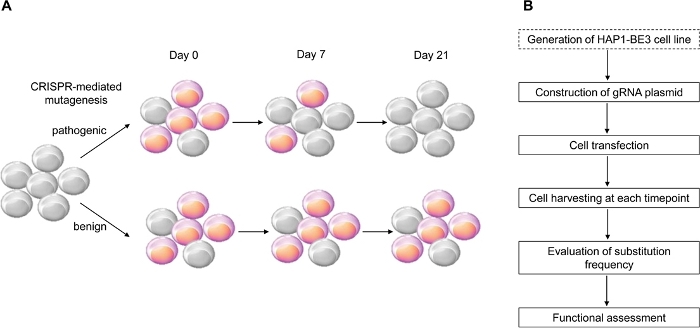
Figure 1 : Aperçu du flux de travail pour l’évaluation fonctionnelle. (A) Schéma montrant l’évaluation fonctionnelle de BRCA1. Puisque le LOF de BRCA1 affecte la viabilité cellulaire, quand la mutation BRCA1 est pathogène, les cellules meurent pendant que le nombre de passage augmente. (B) Étapes de l’évaluation fonctionnelle de BRCA1. La boîte pointillée est facultative. Il peut être remplacé par la co-transfection de l’expression de l’ARN g et be3 exprimant l’ADN des plasmides. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Protocol
Representative Results
Discussion
Ce protocole décrit une méthode simple pour les évaluations fonctionnelles des variantes BRCA1 à l’aide de l’éditeur de base de cytosine médité par CRISPR. Le protocole décrit les méthodes de conception des ARN à locus cible et de construction des ADN plasmides à partir de laquelle ils sont exprimés. Les éditeurs de base de cytosine induisent la conversion nucléotide dans une fenêtre active (en cas de BE3, nucléotides 4-8 dans l’extrémité PAM-distal des séquences cibles de gRNA). Le cher…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Ces travaux ont été soutenus par la National Research Foundation of Korea (subventions 2017M3A9B4062419, 2019R1F1A1057637, et 2018R1A5A2020732 à Y.K.).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
Riferimenti
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Ricerca sul cancro. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

