Sondas de tensión de ADN para mapear las fuerzas transitorias del receptor de piconewton por las células inmunes
Summary
Este documento describe un protocolo detallado para usar sondas de tensión basadas en ADN para obtener imágenes de las fuerzas receptoras aplicadas por las células inmunes. Este enfoque puede mapear las fuerzas del receptor >4.7pN en tiempo real y puede integrar fuerzas a lo largo del tiempo.
Abstract
Las fuerzas mecánicas transmitidas en la unión entre dos células vecinas y en la unión entre las células y la matriz extracelular son críticas para regular muchos procesos que van desde el desarrollo hasta la inmunología. Por lo tanto, desarrollar las herramientas para estudiar estas fuerzas a escala molecular es fundamental. Nuestro grupo desarrolló un conjunto de sensores de tensión molecular para cuantificar y visualizar las fuerzas generadas por las células y transmitidas a ligandos específicos. La clase más sensible de sensores de tensión molecular se compone de horquillas de bucle madre-bucle de ácido nucleico. Estos sensores utilizan pares fluoróforo-apagador para informar sobre la extensión mecánica y el despliegue de horquillas de ADN bajo fuerza. Un desafío con los sensores de tensión de horquilla de ADN es que son reversibles con un rápido repliegue de horquilla al finalizar la tensión y, por lo tanto, las fuerzas transitorias son difíciles de registrar. En este artículo, describimos los protocolos para preparar sensores de tensión de ADN que pueden “bloquearse” y evitar que se vuelvan a plegar para permitir el “almacenamiento” de información mecánica. Esto permite el registro de fuerzas piconewton altamente transitorias, que posteriormente pueden ser “borradas” mediante la adición de ácidos nucleicos complementarios que eliminan la cerradura. Esta capacidad de alternar entre el mapeo de tensión en tiempo real y el almacenamiento de información mecánica revela fuerzas débiles, de corta duración y menos abundantes, que son comúnmente empleadas por las células T como parte de sus funciones inmunes.
Introduction
Las células inmunes se defienden contra patógenos y células cancerosas rastreando y escaneando continuamente las superficies de las células diana en busca de antígenos, tachonando su superficie 1,2. El reconocimiento de antígenos se inicia tras la unión entre el receptor de células T (TCR) y el complejo MHC del complejo de histocompatibilidad péptido-mayor (pMHC) expresado en la superficie de las células diana. Debido a que el reconocimiento TCR-pMHC ocurre en la unión entre dos células móviles, durante mucho tiempo se ha sospechado que experimenta fuerzas mecánicas. Además, esto condujo al modelo mecanosensor de activación TCR, que sugiere que las fuerzas TCR contribuyen a su función 3,4. Para comprender cuándo, dónde y cómo las fuerzas mecánicas contribuyen a la función de las células T, es imperativo desarrollar herramientas para visualizar las fuerzas moleculares transmitidas por las células T. Tradicionalmente, métodos como la microscopía de fuerza de tracción (TFM) y las matrices de micropilares se utilizan para investigar las fuerzas celulares 5,6. Sin embargo, la sensibilidad a la fuerza de las matrices TFM y micropilares está en la escala nanonewton (nN) y, por lo tanto, a menudo es insuficiente para estudiar las fuerzas moleculares de piconewton (pN) transmitidas por los receptores celulares7. Para mejorar la fuerza y la resolución espacial para la detección, nuestro laboratorio fue pionero en el desarrollo de sondas de tensión molecular, que inicialmente se sintetizaron utilizando polímeros de polietilenglicol (PEG)7. Las sondas de tensión molecular están compuestas por un “resorte” molecular extensible (PEG, proteína, ADN) flanqueado por un fluoróforo y un quencher y están anclados en una superficie. Las fuerzas aplicadas al extremo de la sonda conducen a su extensión, separando el fluoróforo y el exprimidor, generando así una fuerte señal de fluorescencia (Figura 1A)8,9,10.
Durante la última década hemos desarrollado una biblioteca de diferentes clases de sondas de tensión molecular con elementos de resorte hechos de ácidos nucleicos11, proteínas10 y polímeros8. Entre estos, las sondas de tensión basadas en ADN proporcionan la mayor relación señal / ruido y la mayor sensibilidad de fuerza, que se sintoniza fácilmente desde unos pocos pN hasta ~ 20 pN11. Hemos utilizado estas sondas de tensión de ADN en tiempo real para estudiar las fuerzas moleculares generadas por muchos tipos de células diversas, incluidos fibroblastos, células cancerosas, plaquetas y células inmunes11,12,13. Este manuscrito describirá protocolos para sintetizar y ensamblar sondas de tensión de ADN en una superficie para mapear las fuerzas del receptor molecular con resolución de fuerza pN utilizando un microscopio de fluorescencia convencional. Si bien el procedimiento actual incluye modificaciones químicas al ácido nucleico para introducir el reportero fluorescente (Figura 1B), es importante tener en cuenta que muchos de los pasos de modificación y purificación pueden subcontratarse a compañías personalizadas de síntesis de ADN. Por lo tanto, la tecnología de sondas de tensión de ADN es fácil y accesible para las comunidades más amplias de biología celular y mecanobiología.
Brevemente, para ensamblar sensores de tensión de ADN, una horquilla de ADN se hibrida con una hebra de ligando fluorescente en un brazo y una hebra de anclaje de quencher en el otro brazo y luego se inmoviliza en un sustrato de vidrio (Figura 1C, tensión en tiempo real). En ausencia de fuerza mecánica, la horquilla se cierra y, por lo tanto, la fluorescencia se apaga. Sin embargo, cuando la fuerza mecánica aplicada es mayor que F1/2 (la fuerza en equilibrio que conduce a una probabilidad del 50% de despliegue), la horquilla se derrite mecánicamente y se genera una señal fluorescente.
Sobre la base del sensor de tensión de ADN en tiempo real, también describimos protocolos para mapear las fuerzas acumuladas, lo que es particularmente útil para estudiar las interacciones entre los receptores en las células inmunes y su ligando natural. Esto se debe a que los receptores inmunes a menudo muestran enlaces de corta duración 3,14. Las fuerzas acumuladas se visualizan utilizando una hebra de “bloqueo” que se une preferentemente a horquillas de ADN abiertas y permite el almacenamiento de señales de fluorescencia asociadas con eventos de tracción mecánica (Figura 1C, tensión bloqueada). La hebra de bloqueo está diseñada para unir un sitio de unión críptico que se expone al derretir mecánicamente la horquilla inducida mecánicamente y bloquear la horquilla en estado abierto bloqueando el plegado de la horquilla, almacenando así la señal de tensión y generando un mapa de tensión acumulada. Además, la hebra de bloqueo está diseñada con un punto de apoyo de ocho nucleótidos, que permite una reacción de desplazamiento de la hebra mediada por la puntera con su complemento completo, la hebra de “desbloqueo”. Con la adición de la hebra de desbloqueo, la hebra de bloqueo atada se quita de la construcción de horquilla, borrando la señal de tensión almacenada y restableciendo la horquilla al estado en tiempo real.
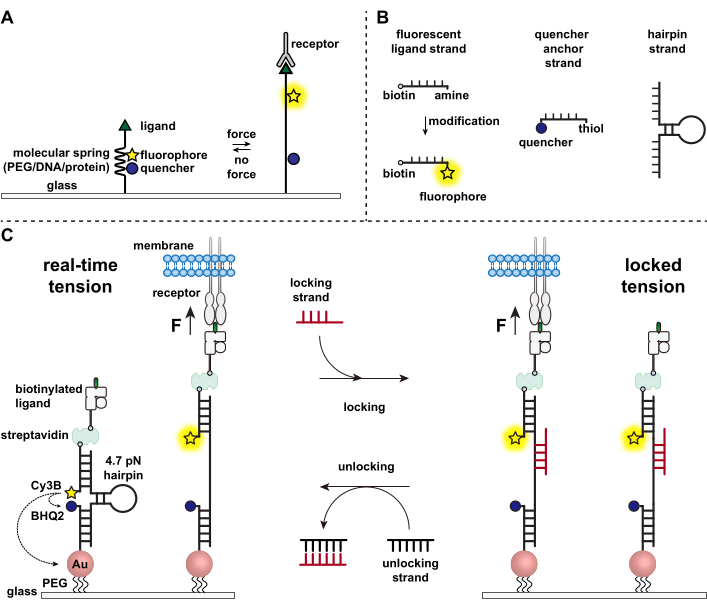
Figura 1: Esquema de las sondas de tensión molecular de última generación. (A) Diseño general de sondas de tensión molecular en tiempo real, (B) hebras para la construcción de la sonda de tensión basada en ADN, y (C) sondas de tensión basadas en ADN diseñadas y su alternancia entre el estado en tiempo real y el estado bloqueado. Haga clic aquí para ver una versión más grande de esta figura.
El protocolo principal consta de cuatro secciones principales: preparación de oligonucleótidos, preparación de superficies, imágenes y análisis de datos. Este protocolo ha sido demostrado con éxito por nuestro laboratorio y otros en células T OT-1 CD8 + ingenuas y activadas, células OT-II CD4 +, así como hibridomas, y se puede aplicar para interrogar diferentes receptores de células inmunes, incluidos el receptor de células T, el receptor de muerte celular programada (PD1) y las fuerzas del antígeno 1 asociado a la función de los linfocitos (LFA-1). Las células T OT-1 CD8 + no naïve se utilizan como una línea celular de ejemplo en este documento.
Protocol
Representative Results
Discussion
Con los procedimientos detallados proporcionados aquí, se pueden preparar sustratos de sonda de tensión de horquilla de ADN para mapear y cuantificar la tensión del receptor producida por las células inmunes. Cuando las células se colocan en el sustrato de la sonda de tensión de horquilla de ADN, aterrizan, se unen y se propagan a medida que los receptores detectan los ligandos tanto química como mecánicamente, el último de los cuales es detectado por nuestras sondas. Sin embargo, en algunos casos las células p…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Este trabajo fue apoyado por las subvenciones R01GM131099 de los NIH, NIH R01GM124472 y NSF CAREER 1350829. Agradecemos a la Instalación de Tetrámeros de los NIH por los ligandos pMHC. Este estudio fue apoyado, en parte, por el Emory Comprehensive Glycomics Core.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Riferimenti
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

