مجسات توتر الحمض النووي لرسم خريطة لقوى مستقبلات بيكونيوتن العابرة بواسطة الخلايا المناعية
Summary
تصف هذه الورقة بروتوكولا مفصلا لاستخدام مجسات التوتر القائمة على الحمض النووي لتصوير قوى المستقبلات التي تطبقها الخلايا المناعية. يمكن لهذا النهج تعيين قوى المستقبلات >4.7pN في الوقت الفعلي ويمكنه دمج القوى بمرور الوقت.
Abstract
تعتبر القوى الميكانيكية التي تنتقل عند التقاطع بين خليتين متجاورتين وعند التقاطع بين الخلايا والمصفوفة خارج الخلية ضرورية لتنظيم العديد من العمليات التي تتراوح من التطور إلى علم المناعة. لذلك ، فإن تطوير الأدوات اللازمة لدراسة هذه القوى على المستوى الجزيئي أمر بالغ الأهمية. طورت مجموعتنا مجموعة من مستشعرات التوتر الجزيئي لتحديد وتصور القوى التي تولدها الخلايا وتنتقل إلى روابط محددة. تتكون الفئة الأكثر حساسية من مستشعرات التوتر الجزيئي من دبابيس شعر حلقة جذعية للحمض النووي. تستخدم هذه المستشعرات أزواج الفلوروفور والتبريد للإبلاغ عن الامتداد الميكانيكي وتكشف دبابيس شعر الحمض النووي تحت القوة. يتمثل أحد التحديات التي تواجه مستشعرات شد دبوس الشعر في الحمض النووي في أنها قابلة للانعكاس مع إعادة طي دبوس الشعر السريع عند انتهاء التوتر وبالتالي يصعب تسجيل القوى العابرة. في هذه المقالة ، نصف بروتوكولات إعداد مستشعرات شد الحمض النووي التي يمكن “قفلها” ومنعها من إعادة الطي لتمكين “تخزين” المعلومات الميكانيكية. وهذا يسمح بتسجيل قوى piconewton العابرة للغاية ، والتي يمكن “محوها” لاحقا عن طريق إضافة الأحماض النووية التكميلية التي تزيل القفل. تكشف هذه القدرة على التبديل بين رسم خرائط التوتر في الوقت الفعلي وتخزين المعلومات الميكانيكية عن قوى ضعيفة وقصيرة العمر وأقل وفرة ، والتي تستخدمها الخلايا التائية بشكل شائع كجزء من وظائفها المناعية.
Introduction
تدافع الخلايا المناعية ضد مسببات الأمراض والخلايا السرطانية عن طريق الزحف المستمر ومسح أسطح الخلايا المستهدفة بحثا عن المستضدات ، وترصيع سطحها 1,2. يبدأ التعرف على المستضد عند الارتباط بين مستقبل الخلايا التائية (TCR) ومركب التوافق النسيجي الببتدي الرئيسي MHC (pMHC) المعبر عنه على سطح الخلايا المستهدفة. نظرا لأن التعرف على TCR-pMHC يحدث عند التقاطع بين خليتين متحركتين ، فقد اشتبه منذ فترة طويلة في أنه يعاني من قوى ميكانيكية. علاوة على ذلك ، أدى ذلك إلى نموذج المستشعر الميكانيكي لتنشيط TCR ، مما يشير إلى أن قوى TCR تساهم في وظيفتها 3,4. لفهم متى وأين وكيف تساهم القوى الميكانيكية في وظيفة الخلايا التائية ، من الضروري تطوير أدوات لتصور القوى الجزيئية التي تنتقل بواسطة الخلايا التائية. تقليديا ، يتم استخدام طرق مثل مجهر قوة الجر (TFM) ومصفوفات micropillar للتحقيق في القوى الخلوية 5,6. ومع ذلك ، فإن حساسية القوة لمصفوفات TFM و micropillar تكون على مقياس nanonewton (nN) ، وبالتالي فهي غالبا ما تكون غير كافية لدراسة قوى piconewton الجزيئية (pN) التي تنتقل بواسطة مستقبلات الخلايا7. لتحسين القوة والدقة المكانية للكشف ، كان مختبرنا رائدا في تطوير مجسات التوتر الجزيئي ، والتي تم تصنيعها في البداية باستخدام بوليمرات البولي إيثيلين جلايكول (PEG)7. تتكون مجسات التوتر الجزيئي من “زنبرك” جزيئي قابل للتمديد (PEG ، بروتين ، حمض نووي) محاط بفلوروفور ومروي ومثبت على سطح. تؤدي القوى المطبقة على نهاية المسبار إلى امتداده ، وفصل الفلوروفور والتبريد ، وبالتالي توليد إشارة مضان قوية (الشكل 1 أ)8،9،10.
على مدار العقد الماضي ، قمنا بتطوير مكتبة من فئات مختلفة من مجسات التوتر الجزيئي مع عناصر زنبركية مصنوعة من الأحماض النووية11 والبروتينات10 والبوليمرات8. من بين هذه ، توفر مجسات التوتر القائمة على الحمض النووي أعلى نسبة إشارة إلى الضوضاء وأكبر حساسية للقوة ، والتي يمكن ضبطها بسهولة من بضعة pN إلى ~ 20 pN11. لقد استخدمنا مجسات توتر الحمض النووي في الوقت الفعلي لدراسة القوى الجزيئية الناتجة عن العديد من أنواع الخلايا المتنوعة ، بما في ذلك الخلايا الليفية والخلايا السرطانية والصفائح الدموية والخلايا المناعية11،12،13. ستصف هذه المخطوطة بروتوكولات لتجميع وتجميع مجسات توتر الحمض النووي على سطح لرسم خريطة لقوى المستقبلات الجزيئية بدقة قوة pN باستخدام مجهر مضان تقليدي. في حين أن الإجراء الحالي يتضمن تعديلات كيميائية على الحمض النووي لإدخال مراسل الفلورسنت (الشكل 1 ب) ، من المهم ملاحظة أن العديد من خطوات التعديل والتنقية يمكن الاستعانة بمصادر خارجية لشركات تخليق الحمض النووي المخصصة. لذلك ، فإن تقنية مجسات توتر الحمض النووي سهلة ، ويمكن الوصول إليها من قبل مجتمعات بيولوجيا الخلية والبيولوجيا الميكانيكية الأوسع.
باختصار ، لتجميع مستشعرات توتر الحمض النووي ، يتم تهجين دبوس شعر الحمض النووي إلى حبلا يجند فلوري على ذراع واحدة وشريط مرساة مروي على الذراع الأخرى ثم يجمد على ركيزة زجاجية (الشكل 1 ج ، التوتر في الوقت الفعلي). في حالة عدم وجود قوة ميكانيكية ، يتم إغلاق دبوس الشعر ، وبالتالي يتم إخماد التألق. ومع ذلك ، عندما تكون القوة الميكانيكية المطبقة أكبر من F1/2 (القوة عند التوازن التي تؤدي إلى احتمال 50٪ للتكشف) ، يذوب دبوس الشعر ميكانيكيا ، ويتم إنشاء إشارة فلورسنت.
بناء على مستشعر توتر الحمض النووي في الوقت الفعلي، نصف أيضا بروتوكولات لرسم خريطة للقوى المتراكمة، وهو أمر مفيد بشكل خاص لدراسة التفاعلات بين المستقبلات على الخلايا المناعية ورباطها الطبيعي. وذلك لأن المستقبلات المناعية غالبا ما تعرض روابط قصيرة العمر 3,14. يتم تصوير القوى المتراكمة باستخدام حبلا “قفل” يرتبط بشكل تفضيلي بفتح دبابيس شعر الحمض النووي ويسمح بتخزين إشارات التألق المرتبطة بأحداث السحب الميكانيكية (الشكل 1C ، التوتر المقفل). تم تصميم حبلا القفل لربط موقع ربط خفي يتعرض عند ذوبان دبوس الشعر المستحث ميكانيكيا وقفل دبوس الشعر في الحالة المفتوحة عن طريق منع إعادة طي دبوس الشعر ، وبالتالي تخزين إشارة التوتر ، وتوليد خريطة توتر متراكمة. علاوة على ذلك ، تم تصميم حبلا القفل بإصبع قدم مكون من ثمانية نيوكليوتيدات ، مما يتيح تفاعل إزاحة حبلا بوساطة إصبع القدم مع مكمله الكامل ، حبلا “فتح”. مع إضافة حبلا الفتح ، يتم تجريد حبلا القفل المربوط من بنية دبوس الشعر ، مما يؤدي إلى محو إشارة التوتر المخزنة وإعادة ضبط دبوس الشعر إلى حالة الوقت الفعلي.
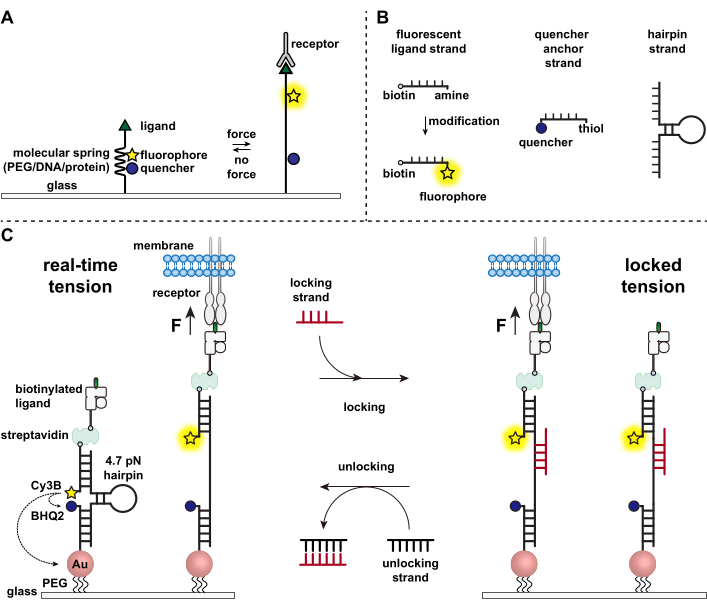
الشكل 1: مخطط أحدث مجسات التوتر الجزيئي. (أ) التصميم العام لمسبار التوتر الجزيئي في الوقت الفعلي، (ب) خيوط بناء مسبار التوتر القائم على الحمض النووي، (ج) مجسات التوتر القائمة على الحمض النووي (DNA) وتبديلها بين الحالة في الوقت الفعلي والحالة المقفلة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
يتكون البروتوكول الرئيسي من أربعة أقسام رئيسية – إعداد قليل النوكليوتيد ، وإعداد السطح ، والتصوير ، وتحليل البيانات. تم إثبات هذا البروتوكول بنجاح من قبل مختبرنا وغيره في خلايا OT-1 CD8 + T الساذجة والمنشطة ، وخلايا OT-II CD4 + ، وكذلك الأورام الهجينة ، ويمكن تطبيقه لاستجواب مستقبلات الخلايا المناعية المختلفة بما في ذلك مستقبلات الخلايا التائية ، ومستقبلات موت الخلايا المبرمجة (PD1) ، وقوى المستضد 1 المرتبطة بوظيفة الخلايا الليمفاوية (LFA-1). تستخدم الخلايا التائية الساذجة OT-1 CD8 + كمثال على خط الخلية في هذه الورقة.
Protocol
Representative Results
Discussion
من خلال الإجراءات التفصيلية المقدمة هنا ، يمكن للمرء إعداد ركائز مسبار شد دبوس الشعر للحمض النووي لرسم خريطة وقياس توتر المستقبلات التي تنتجها الخلايا المناعية. عندما يتم طلاء الخلايا على ركيزة مسبار شد دبوس الشعر DNA ، فإنها تهبط وتلتصق وتنتشر عندما تستشعر المستقبلات الروابط كيميائيا وم?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
تم دعم هذا العمل من قبل منح المعاهد الوطنية للصحة R01GM131099 و NIH R01GM124472 و NSF CAREER 1350829. نشكر مرفق المعاهد الوطنية للصحة Tetramer على روابط pMHC. تم دعم هذه الدراسة ، جزئيا ، من قبل Emory Comprehensive Glycomics Core.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Riferimenti
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

