DNA-spændingssonder til at kortlægge immuncellernes forbigående piconewtonreceptorkræfter
Summary
Dette papir beskriver en detaljeret protokol til brug af DNA-baserede spændingsprober til at afbilde receptorkræfterne anvendt af immunceller. Denne tilgang kan kortlægge receptorkræfter >4,7 pN i realtid og kan integrere kræfter over tid.
Abstract
Mekaniske kræfter, der overføres ved krydset mellem to naboceller og ved krydset mellem celler og den ekstracellulære matrix, er kritiske for regulering af mange processer lige fra udvikling til immunologi. Derfor er det afgørende at udvikle værktøjerne til at studere disse kræfter på molekylær skala. Vores gruppe udviklede en række molekylære spændingssensorer til at kvantificere og visualisere de kræfter, der genereres af celler og overføres til specifikke ligander. Den mest følsomme klasse af molekylære spændingssensorer består af nukleinsyrestamme-loop hårnåle. Disse sensorer bruger fluorofor-quencherpar til at rapportere om den mekaniske forlængelse og udfoldning af DNA-hårnåle under kraft. En udfordring med DNA-hårnålespændingssensorer er, at de er reversible med hurtig hårnål, der foldes sammen igen efter afslutning af spændingen, og derfor er forbigående kræfter vanskelige at registrere. I denne artikel beskriver vi protokollerne til forberedelse af DNA-spændingssensorer, der kan “låses” og forhindres i at folde sammen igen for at muliggøre “lagring” af mekanisk information. Dette muliggør registrering af stærkt forbigående piconewtonkræfter, som efterfølgende kan “slettes” ved tilsætning af komplementære nukleinsyrer, der fjerner låsen. Denne evne til at skifte mellem spændingskortlægning i realtid og mekanisk informationslagring afslører svage, kortvarige og mindre rigelige kræfter, der almindeligvis anvendes af T-celler som en del af deres immunfunktioner.
Introduction
Immunceller forsvarer sig mod patogener og kræftceller ved kontinuerligt at kravle og scanne overfladerne af målceller for antigener og studse deres overflade 1,2. Antigengenkendelse initieres ved binding mellem T-cellereceptoren (TCR) og peptid-major histokompatibilitetskomplekset MHC (pMHC) kompleks udtrykt på overfladen af målceller. Fordi TCR-pMHC-genkendelse sker ved krydset mellem to mobile celler, har det længe været mistænkt for at opleve mekaniske kræfter. Desuden førte dette til mekanosensormodellen for TCR-aktivering, hvilket tyder på, at TCR-kræfter bidrager til dens funktion 3,4. For at forstå, hvornår, hvor og hvordan mekaniske kræfter bidrager til T-cellefunktionen, er det bydende nødvendigt at udvikle værktøjer til at visualisere de molekylære kræfter, der transmitteres af T-celler. Traditionelt bruges metoder som traction force mikroskopi (TFM) og micropillar arrays til at undersøge cellulære kræfter 5,6. Imidlertid er kraftfølsomheden af TFM og mikrosøjle arrays på nanonewton (nN) skalaen og er derfor ofte utilstrækkelig til at studere de molekylære piconewton (pN) kræfter, der transmitteres af cellereceptorer7. For at forbedre kraften og den rumlige opløsning til detektion var vores laboratorium banebrydende for udviklingen af molekylære spændingsprober, som oprindeligt blev syntetiseret ved hjælp af polyethylenglycol (PEG) polymerer7. Molekylspændingssonder består af en udvidelig molekylær “fjeder” (PEG, protein, DNA) flankeret af en fluorofor og slukker og er forankret på en overflade. Kræfter, der påføres sondens endestation, fører til dens forlængelse, adskiller fluoroforen og slukkeren og genererer således et stærkt fluorescenssignal (figur 1A)8,9,10.
I løbet af det sidste årti har vi udviklet et bibliotek af forskellige klasser af molekylære spændingsprober med fjederelementer fremstillet af nukleinsyrer11, proteiner10 og polymerer8. Blandt disse giver de DNA-baserede spændingssonder det højeste signal / støjforhold og den største kraftfølsomhed, som let indstilles fra nogle få pN op til ~ 20 pN11. Vi har brugt disse DNA-spændingssonder i realtid til at studere de molekylære kræfter, der genereres af mange forskellige celletyper, herunder fibroblaster, kræftceller, blodplader og immunceller11,12,13. Dette manuskript vil beskrive protokoller til syntetisering og samling af DNA-spændingssonder på en overflade for at kortlægge molekylære receptorkræfter med pN-kraftopløsning ved hjælp af et konventionelt fluorescensmikroskop. Mens den nuværende procedure inkluderer kemiske modifikationer af nukleinsyren for at introducere den fluorescerende reporter (figur 1B), er det vigtigt at bemærke, at mange af modifikations- og oprensningstrinnene kan outsources til brugerdefinerede DNA-syntesevirksomheder. Derfor er DNA-spændingssondeteknologi let og tilgængelig for de bredere cellebiologi- og mekanobiologiske samfund.
Kort sagt, for at samle DNA-spændingssensorer, hybridiseres en DNA-hårnål til en fluorescerende ligandstreng på den ene arm og en slukningsankerstreng på den anden arm og immobiliseres derefter på et glassubstrat (figur 1C, realtidsspænding). I mangel af mekanisk kraft lukkes hårnålen, og fluorescensen slukkes således. Men når den påførte mekaniske kraft er større end F1/2 (kraften ved ligevægt, der fører til en 50% sandsynlighed for udfoldelse), smelter hårnålen mekanisk, og der genereres et fluorescerende signal.
Med udgangspunkt i DNA-spændingssensoren i realtid beskriver vi også protokoller til kortlægning af akkumulerede kræfter, hvilket er særligt nyttigt til at studere interaktioner mellem receptorer på immunceller og deres naturlige ligand. Dette skyldes, at immunreceptorer ofte viser kortvarige bindinger 3,14. Akkumulerede kræfter afbildes ved hjælp af en “låsende” streng, der fortrinsvis binder til åbne DNA-hårnåle og muliggør lagring af fluorescenssignaler forbundet med mekaniske trækhændelser (figur 1C, låst spænding). Låsestrengen er designet til at binde et kryptisk bindingssted, der eksponeres ved mekanisk induceret smeltning af hårnålen og låse hårnålen i åben tilstand ved at blokere hårnålens foldning igen, således at spændingssignalet lagres og genererer et akkumuleret spændingskort. Desuden er låsestrengen designet med et otte-nukleotid-tåhold, som muliggør en tåholdsmedieret strengforskydningsreaktion med sit fulde komplement, “oplåsningsstrengen”. Med tilføjelsen af oplåsningsstrengen fjernes den bundne låsestreng fra hårnålekonstruktionen, hvilket sletter det lagrede spændingssignal og nulstiller hårnålen tilbage til realtidstilstanden.
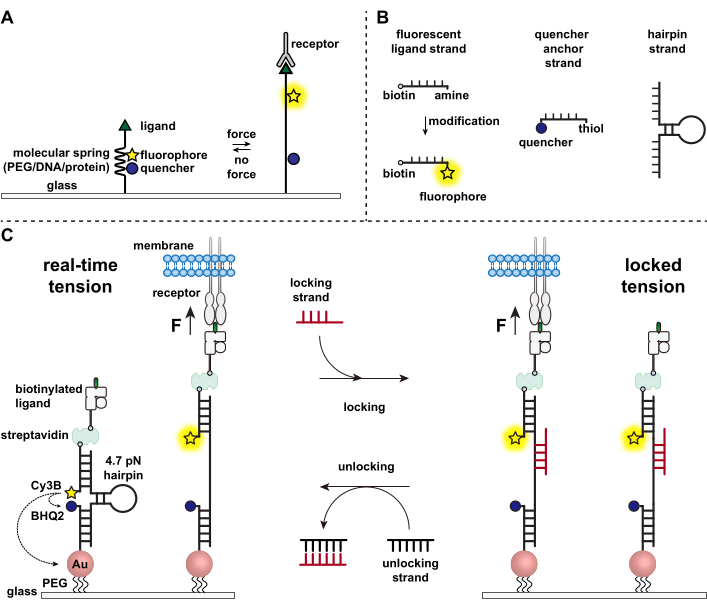
Figur 1: Skema for de nyeste molekylære spændingssonder. (A) Generelt design af molekylær spændingssonde i realtid, (B) strenge til den DNA-baserede spændingssondekonstruktion og (C) konstruerede DNA-baserede spændingssonder og deres skift mellem realtidstilstand og låst tilstand. Klik her for at se en større version af denne figur.
Hovedprotokollen består af fire hovedafsnit – oligonukleotidpræparation, overfladebehandling, billeddannelse og dataanalyse. Denne protokol er med succes blevet demonstreret af vores laboratorium og andre i naive og aktiverede OT-1 CD8 + T-celler, OT-II CD4 + celler samt hybridomer og kan anvendes til at forhøre forskellige immuncellereceptorer, herunder T-cellereceptor, programmeret celledødsreceptor (PD1) og lymfocytfunktionsassocieret antigen 1 (LFA-1) kræfter. OT-1 CD8+ naive T-celler bruges som eksempel på cellelinje i dette papir.
Protocol
Representative Results
Discussion
Med de detaljerede procedurer, der er angivet her, kan man forberede DNA-hårnålespændingssondesubstrater til at kortlægge og kvantificere receptorspændingen produceret af immunceller. Når celler belægges på DNA-hårnålespændingssondesubstratet, lander, fastgøres og spredes, når receptorerne registrerer liganderne både kemisk og mekanisk, hvoraf sidstnævnte detekteres af vores sonder. I nogle tilfælde kan celler dog ikke sprede sig (Figur 7A) eller undlader at frembringe spænd…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Dette arbejde blev støttet af NIH Grants R01GM131099, NIH R01GM124472 og NSF CAREER 1350829. Vi takker NIH Tetramer Facility for pMHC ligander. Denne undersøgelse blev delvist støttet af Emory Comprehensive Glycomics Core.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Riferimenti
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

