免疫細胞による一過性ピコニュートン受容体力をマッピングするためのDNA張力プローブ
Summary
この論文では、DNAベースのテンションプローブを使用して免疫細胞によって加えられる受容体力を画像化するための詳細なプロトコルについて説明します。このアプローチは、受容体の力>4.7pNをリアルタイムでマッピングし、時間の経過とともに力を積分することができます。
Abstract
隣接する2つの細胞間の接合部および細胞と細胞外マトリックス間の接合部で伝達される機械的な力は、発生から免疫学に至るまでの多くのプロセスを調節するために重要です。したがって、分子スケールでこれらの力を研究するためのツールを開発することが重要です。私たちのグループは、細胞によって生成され、特定のリガンドに伝達される力を定量化および視覚化するための一連の分子張力センサーを開発しました。最も感度の高いクラスの分子張力センサーは、核酸ステムループヘアピンで構成されています。これらのセンサーは、蛍光色素と消光物質のペアを使用して、力によるDNAヘアピンの機械的伸長と展開について報告します。DNAヘアピン張力センサーの課題の1つは、張力の終了時にヘアピンが急速に折りたたまれて可逆的であるため、過渡力を記録するのが難しいことです。本稿では、機械的情報の「保存」を可能にするために「ロック」して折り畳みを防止できるDNA張力センサーを準備するためのプロトコルについて説明します。これにより、非常に過渡的なピコネウトン力の記録が可能になり、ロックを除去する相補的核酸を添加することで、その後「消去」することができます。リアルタイムの張力マッピングと機械的情報保存を切り替えるこの能力は、T細胞が免疫機能の一部として一般的に使用する、弱く、短命で、存在量の少ない力を明らかにします。
Introduction
免疫細胞は、標的細胞の表面を連続的に這い回って抗原を探し、その表面をスタッディングすることにより、病原体や癌細胞から防御します1,2。抗原認識は、T細胞受容体(TCR)と標的細胞の表面に発現するペプチド主要組織適合遺伝子複合体MHC(pMHC)複合体との間の結合時に開始される。TCR-pMHC認識は2つのモバイルセル間の接合部で発生するため、機械的な力を受けることが長い間疑われてきました。さらに、これはTCR活性化のメカノセンサーモデルにつながり、TCR力がその機能に寄与することを示唆しています3,4。機械的な力がいつ、どこで、どのようにT細胞の機能に寄与するかを理解するには、T細胞によって伝達される分子力を視覚化するツールを開発することが不可欠です。伝統的に、牽引力顕微鏡(TFM)やマイクロピラーアレイなどの方法が細胞力を調べるために使用されます5,6。しかし、TFMおよびマイクロピラーアレイの力感度はナノニュートン(nN)スケールであるため、細胞受容体によって伝達される分子ピコニュートン(pN)力を研究するには不十分なことがよくあります7。検出力と空間分解能を向上させるために、私たちの研究室は、ポリエチレングリコール(PEG)ポリマーを使用して最初に合成された分子張力プローブの開発を開拓しました7。分子張力プローブは、蛍光色素と消光物質に隣接する拡張可能な分子「バネ」(PEG、タンパク質、DNA)で構成され、表面に固定されています。プローブの末端に力を加えるとプローブが伸長し、蛍光色素と消光物質が分離され、強い蛍光シグナルが生成されます(図1A)8,9,10。
過去10年間にわたり、核酸11、タンパク質10、およびポリマー8から作られたスプリングエレメントを備えたさまざまなクラスの分子張力プローブのライブラリを開発してきました。これらの中で、DNAベースのテンションプローブは、最高のS/N比と最大の力感度を提供し、数pNから~20pNまで簡単に調整できます11。これらのリアルタイムDNAテンションプローブを使用して、線維芽細胞、癌細胞、血小板、免疫細胞など、さまざまな種類の細胞によって生成される分子力を研究しています11,12,13。この原稿では、従来の蛍光顕微鏡を使用してpN力分解能で分子受容体力をマッピングするために、表面上でDNA張力プローブを合成および組み立てるためのプロトコルについて説明します。現在の手順には、蛍光レポーターを導入するための核酸の化学修飾が含まれていますが(図1B)、修飾および精製手順の多くはカスタムDNA合成会社に委託できることに注意することが重要です。したがって、DNA張力プローブ技術は容易であり、より広範な細胞生物学およびメカノバイオロジーコミュニティが利用できます。
簡単に言うと、DNA張力センサーを組み立てるために、DNAヘアピンを一方のアームの蛍光リガンド鎖ともう一方のアームのクエンチャーアンカー鎖にハイブリダイズし、ガラス基板に固定化します(図1C、リアルタイム張力)。機械的な力がない場合、ヘアピンは閉じられ、蛍光は消光します。しかし、加えられた機械的な力がF1/2 (50%の確率で展開する平衡時の力)よりも大きい場合、ヘアピンは機械的に溶け、蛍光シグナルが発生します。
リアルタイムDNA張力センサーに基づいて、免疫細胞上の受容体とその天然リガンドとの間の相互作用を研究するのに特に役立つ蓄積された力をマッピングするためのプロトコルについても説明します。これは、免疫受容体がしばしば短命結合を示すためである3,14。蓄積された力は、開いたDNAヘアピンに優先的に結合し、機械的引っ張りイベントに関連する蛍光シグナルの保存を可能にする「ロック」鎖を使用して画像化されます(図1C、ロック張力)。ロッキングストランドは、ヘアピンの機械的に誘発された融解時に露出する不可解な結合部位を結合し、ヘアピンの再折り畳みをブロックして張力信号を格納し、蓄積された張力マップを生成することによってヘアピンを開いた状態にロックするように設計されています。さらに、ロッキング鎖は8ヌクレオチドのトーホールドで設計されており、その完全な補体である「ロック解除」鎖とのトーホールドを介した鎖置換反応を可能にします。ロック解除ストランドを追加すると、バインドされたロックストランドがヘアピン構造から剥がされ、保存されているテンション信号が消去され、ヘアピンがリアルタイム状態にリセットされます。
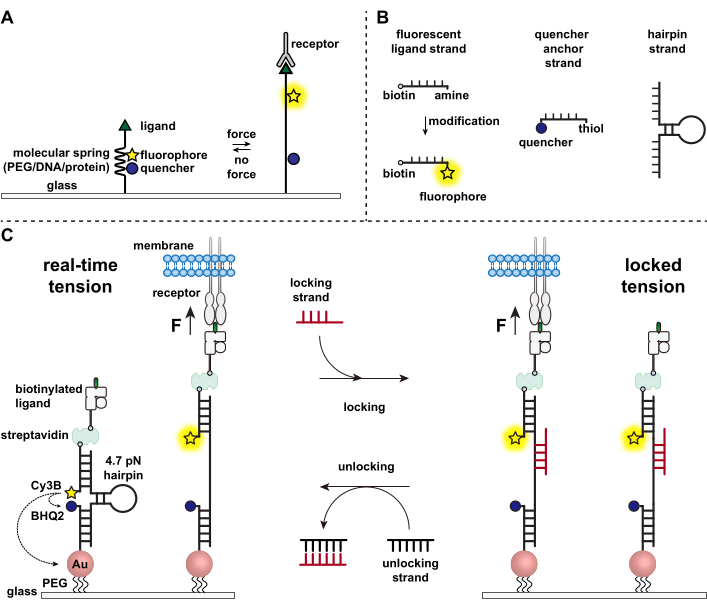
図1:最先端の分子張力プローブのスキーム 。 (A)リアルタイム分子張力プローブの一般的な設計、(B)DNAベースの張力プローブ構築のための鎖、および(C)設計されたDNAベースの張力プローブと、リアルタイム状態とロック状態の間のそれらの切り替え。 この図の拡大版を表示するには、ここをクリックしてください。
メインプロトコルは、オリゴヌクレオチド調製、表面処理、イメージング、およびデータ分析の4つの主要なセクションで構成されています。このプロトコルは、ナイーブで活性化されたOT-1 CD8+ T細胞、OT-II CD4 + 細胞、およびハイブリドーマにおいて私たちの研究室や他の人々によって首尾よく実証されており、T細胞受容体、プログラム細胞死受容体(PD1)、およびリンパ球機能関連抗原1(LFA-1)力を含むさまざまな免疫細胞受容体を調べるために適用できます。OT-1 CD8+ ナイーブT細胞は、この論文の細胞株の例として使用されています。
Protocol
Representative Results
Discussion
ここで提供される詳細な手順を使用して、免疫細胞によって産生される受容体張力をマッピングおよび定量するためのDNAヘアピン張力プローブ基板を調製することができます。細胞がDNAヘアピンテンションプローブ基板にプレーティングされると、受容体が化学的および機械的にリガンドを感知し、後者はプローブによって検出されるため、細胞は着地、付着、拡散します。ただし、場合に?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
この作業は、NIH Grants R01GM131099、NIH R01GM124472、およびNSF CAREER 1350829の支援を受けました。pMHCリガンドに関するNIHテトラマーファシリティに感謝します。この研究は、Emory Comprehensive Glycomics Coreによって部分的にサポートされました。
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Riferimenti
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

