DNA-spenningssonder for å kartlegge immuncellers forbigående piconeton-reseptorkrefter
Summary
Dette papiret beskriver en detaljert protokoll for bruk av DNA-baserte spenningsprober for å avbilde reseptorkreftene som påføres av immunceller. Denne tilnærmingen kan kartlegge reseptorkrefter >4.7pN i sanntid og kan integrere krefter over tid.
Abstract
Mekaniske krefter overført ved krysset mellom to naboceller og ved krysset mellom celler og den ekstracellulære matrisen er kritiske for å regulere mange prosesser som spenner fra utvikling til immunologi. Derfor er det viktig å utvikle verktøyene for å studere disse kreftene på molekylær skala. Vår gruppe utviklet en pakke med molekylære spenningssensorer for å kvantifisere og visualisere kreftene som genereres av celler og overføres til bestemte ligander. Den mest følsomme klassen av molekylære spenningssensorer består av nukleinsyrestamsløyfehårnåler. Disse sensorene bruker fluorofor-slukkerpar for å rapportere om mekanisk forlengelse og utfoldelse av DNA-hårnåler under kraft. En utfordring med DNA-hårnålsspenningssensorer er at de er reversible med rask hårnålsfolding ved avslutning av spenningen, og dermed er forbigående krefter vanskelige å registrere. I denne artikkelen beskriver vi protokollene for å forberede DNA-spenningssensorer som kan “låses” og forhindres i å brettes på nytt for å muliggjøre “lagring” av mekanisk informasjon. Dette muliggjør opptak av svært forbigående piconewton-krefter, som deretter kan “slettes” ved tilsetning av komplementære nukleinsyrer som fjerner låsen. Denne evnen til å veksle mellom sanntids spenningskartlegging og mekanisk informasjonslagring avslører svake, kortvarige og mindre rikelig krefter, som ofte brukes av T-celler som en del av deres immunfunksjoner.
Introduction
Immunceller forsvarer seg mot patogener og kreftceller ved kontinuerlig å krype og skanne overflatene til målceller for antigener, og pigge overflaten 1,2. Antigengjenkjenning initieres ved binding mellom T-cellereseptoren (TCR) og peptid-major histokompatibilitetskomplekset MHC (pMHC) komplekset uttrykt på overflaten av målceller. Fordi TCR-pMHC-gjenkjenning skjer i krysset mellom to mobilceller, har det lenge vært mistanke om å oppleve mekaniske krefter. Videre førte dette til mekanosensormodellen for TCR-aktivering, noe som antyder at TCR-krefter bidrar til funksjonen 3,4. For å forstå når, hvor og hvordan mekaniske krefter bidrar til T-cellefunksjon, er det viktig å utvikle verktøy for å visualisere molekylære krefter som overføres av T-celler. Tradisjonelt brukes metoder som trekkraftmikroskopi (TFM) og mikropillararrays for å undersøke cellulære krefter 5,6. Imidlertid er kraftfølsomheten til TFM og mikropilarrayer på nanonewton (nN) -skalaen og er derfor ofte utilstrekkelig til å studere molekylære piconewton (pN) -krefter overført av cellereseptorer7. For å forbedre kraften og romlig oppløsning for deteksjon, var laboratoriet vårt banebrytende for utviklingen av molekylære spenningsprober, som opprinnelig ble syntetisert ved bruk av polyetylenglykol (PEG) polymerer7. Molekylære spenningsprober består av en utvidbar molekylær “fjær” (PEG, protein, DNA) flankert av en fluorofor og slukker og er forankret på en overflate. Krefter påført sondens endepunkt fører til dens forlengelse, separerer fluoroforen og slukkeren, og genererer dermed et sterkt fluorescenssignal (figur 1A) 8,9,10.
I løpet av det siste tiåret har vi utviklet et bibliotek med forskjellige klasser av molekylære spenningsprober med fjærelementer laget av nukleinsyrer11, proteiner10 og polymerer8. Blant disse gir de DNA-baserte spenningsprobene det høyeste signal-støyforholdet og den største kraftfølsomheten, som enkelt justeres fra noen få pN opp til ~20 pN11. Vi har brukt disse sanntids DNA-spenningsprobene til å studere molekylære krefter generert av mange forskjellige celletyper, inkludert fibroblaster, kreftceller, blodplater og immunceller11,12,13. Dette manuskriptet vil beskrive protokoller for å syntetisere og sette sammen DNA-spenningsprober på en overflate for å kartlegge molekylære reseptorkrefter med pN-kraftoppløsning ved hjelp av et konvensjonelt fluorescensmikroskop. Mens den nåværende prosedyren inkluderer kjemiske modifikasjoner av nukleinsyren for å introdusere fluorescerende reporter (figur 1B), er det viktig å merke seg at mange av modifikasjons- og rensetrinnene kan outsources til tilpassede DNA-syntesefirmaer. Derfor er DNA-spenningssondeteknologien lett og tilgjengelig for de bredere cellebiologi- og mekanobiologimiljøene.
Kort sagt, for å montere DNA-spenningssensorer, hybridiseres en DNA-hårnål til en fluorescerende ligandstreng på den ene armen og en slukkeankerstreng på den andre armen og immobiliseres deretter på et glasssubstrat (figur 1C, sanntidsspenning). I fravær av mekanisk kraft er hårnålen lukket, og dermed slokkes fluorescensen. Men når den påførte mekaniske kraften er større enn F1/2 (kraften ved likevekt som fører til en 50% sannsynlighet for å utfolde seg), smelter hårnålen mekanisk, og et fluorescerende signal genereres.
Ved å bygge på sanntids DNA-spenningssensor beskriver vi også protokoller for å kartlegge akkumulerte krefter, noe som er spesielt nyttig for å studere interaksjoner mellom reseptorer på immunceller og deres naturlige ligand. Dette skyldes at immunreseptorer ofte viser kortvarige bindinger 3,14. Akkumulerte krefter avbildes ved hjelp av en “låsende” streng som fortrinnsvis binder seg til åpne DNA-hårnåler og muliggjør lagring av fluorescenssignaler assosiert med mekaniske trekkhendelser (figur 1C, låst spenning). Låsestrengen er designet for å binde et kryptisk bindingssted som eksponeres ved mekanisk indusert smelting av hårnålen og låse hårnålen i åpen tilstand ved å blokkere hårnålsfolding, og dermed lagre spenningssignalet og generere et akkumulert spenningskart. Videre er låsestrengen designet med et åtte-nukleotidtåhold, som muliggjør en tåholdsmediert strengforskyvningsreaksjon med sitt fulle komplement, den “låsende” strengen. Med tillegg av opplåsingsstrengen fjernes den bundne låsestrengen av hårnålskonstruksjonen, sletter det lagrede spenningssignalet og tilbakestiller hårnålen tilbake til sanntidstilstanden.
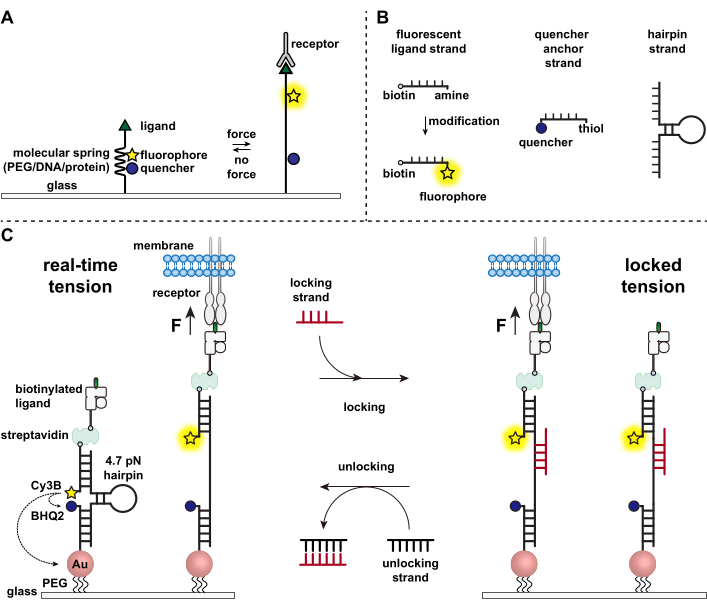
Figur 1: Skjema for state-of-art molekylære spenningsprober. (A) Generell utforming av sanntids molekylær spenningssonde, (B) Tråder for DNA-basert spenningssondekonstruksjon, og (C) konstruerte DNA-baserte spenningsprober og deres veksling mellom sanntidstilstand og låst tilstand. Klikk her for å se en større versjon av denne figuren.
Hovedprotokollen består av fire hovedseksjoner – oligonukleotidpreparasjon, overflatebehandling, avbildning og dataanalyse. Denne protokollen har blitt vellykket demonstrert av laboratoriet vårt og andre i naive og aktiverte OT-1 CD8 + T-celler, OT-II CD4 + -celler, samt hybridomer, og kan brukes til å forhøre forskjellige immuncellereseptorer, inkludert T-cellereseptor, programmert celledødsreseptor (PD1) og lymfocyttfunksjonsassosiert antigen 1 (LFA-1) krefter. OT-1 CD8+ naive T-celler brukes som eksempelcellelinje i denne artikkelen.
Protocol
Representative Results
Discussion
Med de detaljerte prosedyrene som er gitt her, kan man forberede DNA-hårnålsspenningssondesubstrater for å kartlegge og kvantifisere reseptorspenningen produsert av immunceller. Når celler blir belagt på DNA-hårnålsspenningssondesubstratet, lander, fester de seg og sprer seg når reseptorene sanser ligandene både kjemisk og mekanisk, hvorav sistnevnte oppdages av våre sonder. I noen tilfeller kan imidlertid celler mislykkes i å spre seg (Figur 7A) eller ikke produserer spenningssig…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Dette arbeidet ble støttet av NIH Grants R01GM131099, NIH R01GM124472 og NSF CAREER 1350829. Vi takker NIH Tetramer Facility for pMHC-ligander. Denne studien ble delvis støttet av Emory Comprehensive Glycomics Core.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Riferimenti
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

