Зонды натяжения ДНК для картирования переходных сил рецептора пиконьютона иммунными клетками
Summary
В этой статье описывается подробный протокол использования датчиков натяжения на основе ДНК для визуализации рецепторных сил, приложенных иммунными клетками. Этот подход может отображать рецепторные силы >4,7 пН в режиме реального времени и может интегрировать силы с течением времени.
Abstract
Механические силы, передаваемые на стыке между двумя соседними клетками и на стыке между клетками и внеклеточным матриксом, имеют решающее значение для регуляции многих процессов, начиная от развития и заканчивая иммунологией. Поэтому разработка инструментов для изучения этих сил на молекулярном уровне имеет решающее значение. Наша группа разработала набор датчиков молекулярного натяжения для количественной оценки и визуализации сил, генерируемых клетками и передаваемых конкретным лигандам. Наиболее чувствительный класс датчиков молекулярного натяжения состоит из шпилек со стволовыми петлями нуклеиновых кислот. Эти датчики используют пары флуорофор-гаситель, чтобы сообщать о механическом расширении и разворачивании шпилек ДНК под действием силы. Одна из проблем, связанных с датчиками натяжения шпилек ДНК, заключается в том, что они обратимы с быстрым складыванием шпильки после прекращения натяжения, и, таким образом, переходные силы трудно зарегистрировать. В этой статье мы описываем протоколы подготовки датчиков натяжения ДНК, которые можно «заблокировать» и предотвратить повторное складывание, чтобы обеспечить «хранение» механической информации. Это позволяет регистрировать высокопереходные пиконетонные силы, которые впоследствии могут быть «стерты» добавлением комплементарных нуклеиновых кислот, снимающих блокировку. Эта способность переключаться между картированием напряжения в реальном времени и механическим хранением информации выявляет слабые, кратковременные и менее обильные силы, которые обычно используются Т-клетками как часть их иммунных функций.
Introduction
Иммунные клетки защищаются от патогенов и раковых клеток, непрерывно ползая и сканируя поверхности клеток-мишеней на наличие антигенов, усеивая их поверхность 1,2. Распознавание антигена инициируется при связывании между Т-клеточным рецептором (TCR) и комплексом пептидно-главной гистосовместимости MHC (pMHC), экспрессируемым на поверхности клеток-мишеней. Поскольку распознавание TCR-pMHC происходит на стыке между двумя подвижными клетками, долгое время подозревалось, что он испытывает механические силы. Более того, это привело к механосенсорной модели активации TCR, которая предполагает, что силы TCR способствуют его функции 3,4. Чтобы понять, когда, где и как механические силы способствуют функционированию Т-клеток, необходимо разработать инструменты для визуализации молекулярных сил, передаваемых Т-клетками. Традиционно для исследования клеточных сил5,6 используются такие методы, как тракционно-силовая микроскопия (TFM) и микропиллярные матрицы. Однако силовая чувствительность TFM и микростолбовых матриц находится в масштабе наноньютона (nN) и, таким образом, часто недостаточна для изучения молекулярных пиконьютонных (pN) сил, передаваемых клеточными рецепторами7. Чтобы улучшить силу и пространственное разрешение для обнаружения, наша лаборатория впервые разработала зонды молекулярного натяжения, которые первоначально были синтезированы с использованием полимеров полиэтиленгликоля (ПЭГ)7. Зонды молекулярного натяжения состоят из выдвижной молекулярной «пружины» (ПЭГ, белок, ДНК), окруженной флуорофором и гасителем, и закреплены на поверхности. Силы, приложенные к концу зонда, приводят к его расширению, разделяя флуорофор и гаситель, и, таким образом, генерируя сильный сигнал флуоресценции (рис. 1А)8,9,10.
За последнее десятилетие мы разработали библиотеку различных классов зондов молекулярного натяжения с пружинными элементами, изготовленными из нуклеиновых кислот11, белков10 и полимеров8. Среди них датчики натяжения на основе ДНК обеспечивают самое высокое соотношение сигнал/шум и наибольшую чувствительность к силе, которая легко настраивается от нескольких пН до ~20 пН11. Мы использовали эти зонды натяжения ДНК в реальном времени для изучения молекулярных сил, генерируемых многими различными типами клеток, включая фибробласты, раковые клетки, тромбоциты и иммунные клетки11,12,13. В этой рукописи будут описаны протоколы синтеза и сборки зондов натяжения ДНК на поверхности для картирования молекулярных рецепторных сил с разрешением силы pN с использованием обычного флуоресцентного микроскопа. В то время как текущая процедура включает химические модификации нуклеиновой кислоты для введения флуоресцентного репортера (рис. 1B), важно отметить, что многие этапы модификации и очистки могут быть переданы на аутсорсинг компаниям, занимающимся синтезом ДНК. Таким образом, технология зондов натяжения ДНК проста и доступна для более широких сообществ клеточной биологии и механобиологии.
Вкратце, для сборки датчиков натяжения ДНК шпилька ДНК гибридизуется с флуоресцентной цепью лиганда на одном плече и якорной нитью гасителя на другом плече, а затем иммобилизуется на стеклянной подложке (рис. 1C, натяжение в реальном времени). При отсутствии механической силы шпилька закрывается, и таким образом флуоресценция гасится. Однако, когда приложенная механическая сила больше, чем F1/2 (сила в равновесии, которая приводит к 50% вероятности разворачивания), шпилька механически плавится, и генерируется флуоресцентный сигнал.
Основываясь на датчике натяжения ДНК в реальном времени, мы также описываем протоколы для отображения накопленных сил, что особенно полезно для изучения взаимодействия между рецепторами на иммунных клетках и их естественным лигандом. Это связано с тем, что иммунные рецепторы часто демонстрируют короткоживущие связи 3,14. Накопленные силы визуализируются с помощью «запирающей» цепи, которая преимущественно связывается с открытыми шпильками ДНК и позволяет хранить флуоресцентные сигналы, связанные с механическими событиями вытягивания (рис. 1C, заблокированное натяжение). Фиксирующая прядь предназначена для связывания загадочного места связывания, который подвергается воздействию при механически индуцированном плавлении шпильки, и блокировки шпильки в открытом состоянии путем блокировки повторного складывания шпильки, тем самым сохраняя сигнал натяжения и генерируя накопленную карту натяжения. Кроме того, блокирующая цепь имеет восьминуклеотидную опору, которая обеспечивает реакцию смещения цепи, опосредованную опорой на носок, с ее полным дополнением, «разблокирующей» нитью. С добавлением отпирающей пряди связанная фиксирующая прядь снимается с конструкции шпильки, стирая сохраненный сигнал натяжения и сбрасывая шпильку обратно в состояние реального времени.
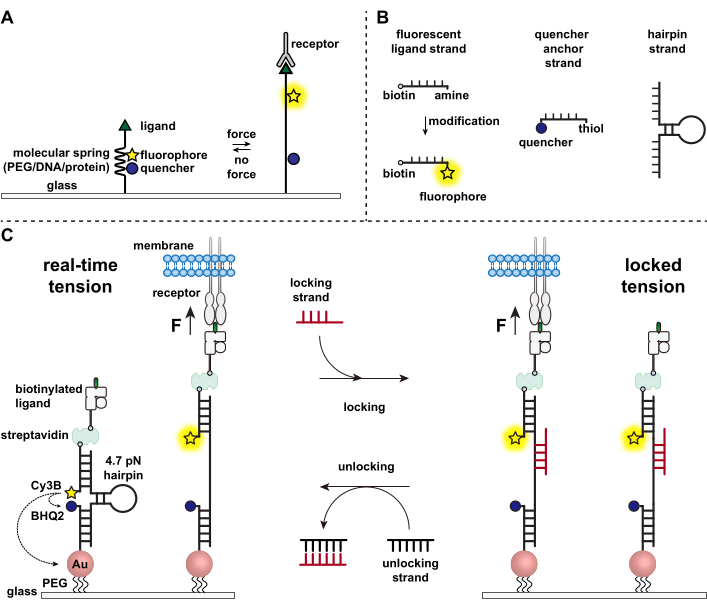
Рисунок 1: Схема современных датчиков молекулярного натяжения. (A) Общая конструкция датчика молекулярного натяжения в реальном времени, (B) Нити для конструкции датчика натяжения на основе ДНК и (C) спроектированные датчики натяжения на основе ДНК и их переключение между состоянием реального времени и заблокированным состоянием. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Основной протокол состоит из четырех основных разделов – подготовка олигонуклеотидов, подготовка поверхности, визуализация и анализ данных. Этот протокол был успешно продемонстрирован нашей лабораторией и другими в наивных и активированных OT-1 CD8+ Т-клетках, OT-II CD4+ клетках, а также гибридомах и может применяться для опроса различных рецепторов иммунных клеток, включая рецептор Т-клеток, рецептор запрограммированной клеточной смерти (PD1) и силы антигена 1, связанного с функцией лимфоцитов (LFA-1). OT-1 CD8+ наивные Т-клетки используются в качестве примера клеточной линии в этой статье.
Protocol
Representative Results
Discussion
С помощью подробных процедур, представленных здесь, можно подготовить субстраты зонда натяжения шпильки ДНК для картирования и количественной оценки напряжения рецепторов, продуцируемого иммунными клетками. Когда клетки наносятся на подложку зонда натяжения шпильки ДНК, они приземл…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Эта работа была поддержана грантами NIH R01GM131099, NIH R01GM124472 и NSF CAREER 1350829. Мы благодарим Центр тетрамеров NIH за лиганды pMHC. Это исследование было частично поддержано Emory Comprehensive Glycomics Core.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Riferimenti
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

