DNA-spänningssonder för att kartlägga de övergående piconewtonreceptorkrafterna av immunceller
Summary
Detta dokument beskriver ett detaljerat protokoll för att använda DNA-baserade spänningssonder för att avbilda receptorkrafterna som appliceras av immunceller. Detta tillvägagångssätt kan kartlägga receptorkrafter >4.7pN i realtid och kan integrera krafter över tid.
Abstract
Mekaniska krafter som överförs vid korsningen mellan två angränsande celler och vid korsningen mellan celler och den extracellulära matrisen är avgörande för att reglera många processer som sträcker sig från utveckling till immunologi. Därför är det viktigt att utveckla verktygen för att studera dessa krafter i molekylär skala. Vår grupp utvecklade en serie molekylära spänningssensorer för att kvantifiera och visualisera de krafter som genereras av celler och överförs till specifika ligander. Den känsligaste klassen av molekylära spänningssensorer består av nukleinsyrastamslinga hårnålar. Dessa sensorer använder fluorofor-släckarpar för att rapportera om den mekaniska förlängningen och utvecklingen av DNA-hårnålar som är i kraft. En utmaning med DNA-hårnålsspänningssensorer är att de är reversibla med snabb hårnålsvikning vid avslutad spänning och därmed är övergående krafter svåra att registrera. I den här artikeln beskriver vi protokollen för att förbereda DNA-spänningssensorer som kan “låsas” och förhindras från att vikas om för att möjliggöra “lagring” av mekanisk information. Detta möjliggör inspelning av mycket övergående piconewtonkrafter, som därefter kan “raderas” genom tillsats av komplementära nukleinsyror som tar bort låset. Denna förmåga att växla mellan spänningskartläggning i realtid och mekanisk informationslagring avslöjar svaga, kortlivade och mindre rikliga krafter, som vanligtvis används av T-celler som en del av deras immunfunktioner.
Introduction
Immunceller försvarar sig mot patogener och cancerceller genom att kontinuerligt krypa och skanna målcellernas ytor för antigener, studding deras yta 1,2. Antigenigenkänning initieras vid bindning mellan T-cellreceptorn (TCR) och det peptid-stora histokompatibilitetskomplexet MHC (pMHC) -komplexet uttryckt på ytan av målceller. Eftersom TCR-pMHC-igenkänning sker vid korsningen mellan två mobila celler har den länge misstänkts för att uppleva mekaniska krafter. Dessutom ledde detta till mekanosensormodellen för TCR-aktivering, vilket tyder på att TCR-krafter bidrar till dess funktion 3,4. För att förstå när, var och hur mekaniska krafter bidrar till T-cellernas funktion är det absolut nödvändigt att utveckla verktyg för att visualisera de molekylära krafter som överförs av T-celler. Traditionellt används metoder som dragkraftmikroskopi (TFM) och mikropelarrayer för att undersöka cellulära krafter 5,6. Emellertid är kraftkänsligheten hos TFM- och mikropelarrayer på nanonewton (nN) -skalan och är därför ofta otillräcklig för att studera molekylära piconewton (pN) -krafter som överförs av cellreceptorer7. För att förbättra kraften och den rumsliga upplösningen för detektion var vårt laboratorium banbrytande för utvecklingen av molekylära spänningsprober, som ursprungligen syntetiserades med användning av polyetylenglykol (PEG) polymerer7. Molekylära spänningsprober består av en utdragbar molekylär “fjäder” (PEG, protein, DNA) flankerad av en fluorofor och släckare och förankras på en yta. Krafter som appliceras på sondens ändpunkt leder till dess förlängning, separerar fluoroforen och släckaren och genererar därmed en stark fluorescenssignal (figur 1A)8,9,10.
Under det senaste decenniet har vi utvecklat ett bibliotek av olika klasser av molekylära spänningsprober med fjäderelement tillverkade av nukleinsyror11, proteiner10 och polymerer8. Bland dessa ger de DNA-baserade spänningsproberna det högsta signal-brusförhållandet och den största kraftkänsligheten, som enkelt ställs in från några pN upp till ~ 20 pN11. Vi har använt dessa DNA-spänningsprober i realtid för att studera de molekylära krafterna som genereras av många olika celltyper, inklusive fibroblaster, cancerceller, blodplättar och immunceller11,12,13. Detta manuskript kommer att beskriva protokoll för att syntetisera och montera DNA-spänningsprober på en yta för att kartlägga molekylära receptorkrafter med pN-kraftupplösning med hjälp av ett konventionellt fluorescensmikroskop. Medan det nuvarande förfarandet inkluderar kemiska modifieringar av nukleinsyran för att introducera den fluorescerande reportern (figur 1B), är det viktigt att notera att många av modifierings- och reningsstegen kan outsourcas till anpassade DNA-syntesföretag. Därför är DNA-spänningsprober teknik lätt och tillgänglig för de bredare cellbiologi- och mekanobiologiska samhällena.
Kortfattat, för att montera DNA-spänningssensorer, hybridiseras en DNA-hårnål till en fluorescerande ligandsträng på ena armen och en släckarankarsträng på den andra armen och immobiliseras sedan på ett glassubstrat (Figur 1C, realtidsspänning). I frånvaro av mekanisk kraft stängs hårnålen och fluorescensen släckes. Men när den applicerade mekaniska kraften är större än F1/2 (kraften vid jämvikt som leder till en 50% sannolikhet för utfällning) smälter hårnålen mekaniskt och en fluorescerande signal genereras.
Med utgångspunkt i DNA-spänningssensorn i realtid beskriver vi också protokoll för att kartlägga ackumulerade krafter, vilket är särskilt användbart för att studera interaktioner mellan receptorer på immunceller och deras naturliga ligand. Detta beror på att immunreceptorer ofta uppvisar kortlivade bindningar 3,14. Ackumulerade krafter avbildas med hjälp av en “låsande” sträng som företrädesvis binder till öppna DNA-hårnålar och möjliggör lagring av fluorescenssignaler associerade med mekaniska draghändelser (figur 1C, låst spänning). Låssträngen är utformad för att binda ett kryptiskt bindningsställe som exponeras vid mekaniskt inducerad smältning av hårnålen och låsa hårnålen i öppet tillstånd genom att blockera hårnålens omvikning, vilket lagrar spänningssignalen och genererar en ackumulerad spänningskarta. Dessutom är låssträngen utformad med en åtta-nukleotid-tåhållare, vilket möjliggör en tåhållmedierad strängförskjutningsreaktion med sitt fulla komplement, den “upplåsta” strängen. Med tillägget av upplåsningssträngen avlägsnas den bundna låssträngen från hårnålskonstruktionen, raderar den lagrade spänningssignalen och återställer hårnålen till realtidsläget.
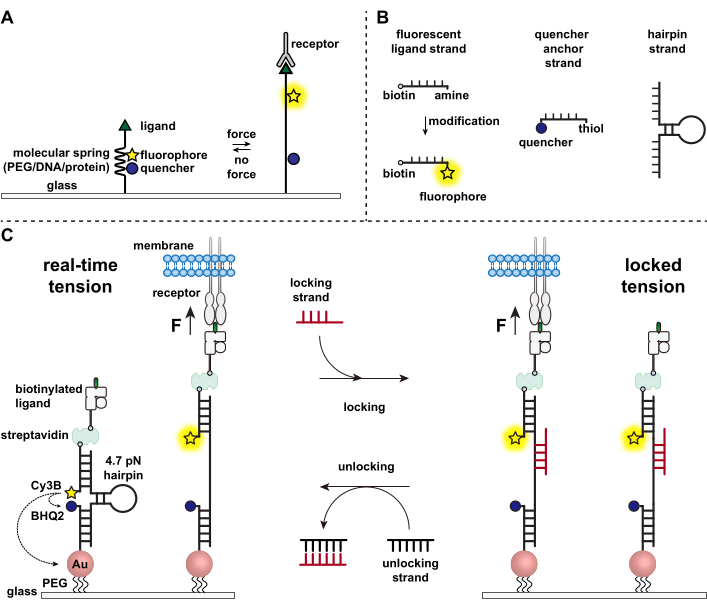
Figur 1: Schema för de toppmoderna molekylära spänningsproberna . (A) Allmän design av molekylär spänningssond i realtid, (B) strängar för den DNA-baserade spänningssondkonstruktionen, och (C) konstruerade DNA-baserade spänningssonder och deras växling mellan realtidstillstånd och låst tillstånd. Klicka här för att se en större version av denna figur.
Huvudprotokollet består av fyra huvudsektioner – oligonukleotidberedning, ytbehandling, avbildning och dataanalys. Detta protokoll har framgångsrikt demonstrerats av vårt laboratorium och andra i naiva och aktiverade OT-1 CD8 + T-celler, OT-II CD4 + -celler, såväl som hybridom, och kan tillämpas för att förhöra olika immuncellreceptorer inklusive T-cellreceptor, programmerad celldödsreceptor (PD1) och lymfocytfunktionsassocierad antigen 1 (LFA-1) krafter. OT-1 CD8 + naiva T-celler används som exempel cellinje i detta papper.
Protocol
Representative Results
Discussion
Med de detaljerade procedurerna som tillhandahålls här kan man förbereda DNA-hårnålsspänningssondsubstrat för att kartlägga och kvantifiera receptorspänningen som produceras av immunceller. När celler pläteras på DNA-hårnålsspänningssondsubstratet landar, fäster och sprider de sig när receptorerna känner av liganderna både kemiskt och mekaniskt, varav den senare detekteras av våra sonder. I vissa fall kan celler dock misslyckas med att sprida sig (Figur 7A) eller misslyck…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Detta arbete stöddes av NIH Grants R01GM131099, NIH R01GM124472 och NSF CAREER 1350829. Vi tackar NIH Tetramer Facility för pMHC-ligander. Denna studie stöddes delvis av Emory Comprehensive Glycomics Core.
Materials
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ |
AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification |
A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification |
GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC |
hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ |
TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ |
CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification |
TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Riferimenti
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).

