Expressão bacteriana e purificação da matriz humana Metaloproteinase-3 usando cromatografia de afinidade
Summary
Sua marca purificação, diálise e ativação são empregadas para aumentar os rendimentos da expressão proteica de domínio catalítico de matriz solúvel-3 em bactérias. As frações proteicas são analisadas através de géis SDS-PAGE.
Abstract
As metaloproteínas matricial (MMPs) pertencem à família de proteases metzincinas com papéis centrais na degradação e remodelagem da matriz extracelular (ECM), bem como interações com diversos fatores de crescimento e citocinas. A superexpressão de MMPs específicos é responsável por diversas doenças, como câncer, doenças neurodegenerativas e doenças cardiovasculares. Os MMPs têm sido o centro das atenções recentemente como metas para desenvolver terapêuticas que possam tratar doenças correlacionadas à superexpressão de MMP.
Para estudar o mecanismo MMP em solução, são necessários métodos de expressão e purificação de proteínas recombinantes mais fáceis e robustas para a produção de MMPs ativos e solúveis. No entanto, o domínio catallítico da maioria dos MMPs não pode ser expresso em Escherichia coli (E. coli) em forma solúvel devido à falta de máquinas pós-transicionais, enquanto os sistemas de expressão dos mamíferos são geralmente caros e têm rendimentos mais baixos. Os órgãos de inclusão de MMP devem passar pelo processo tedioso e trabalhoso de purificação extensiva e re dobra, reduzindo significativamente o rendimento de MMPs na conformação nativa. Este artigo apresenta um protocolo usando células Rosetta2(DE3)pLysS (doravante referida como R2DP) para produzir domínio catalítico matrix metalloproteinase-3 (MMP-3cd), que contém uma tag N-terminal Sua seguida por pró-domínio (Hisx6-pro-MMP-3cd) para uso na purificação de afinidade. As células R2DP aumentam a expressão de proteínas eucarióticas através de um plasmídeo resistente a clororamfenícol contendo códons normalmente raros em sistemas de expressão bacteriana. Em comparação com a linha celular tradicional de escolha para expressão de proteína recombinante, BL21(DE3), a purificação usando esta nova cepa melhorou o rendimento do Hisx6-pro-MMP-3cd purificado. Após a ativação e desalagem, o domínio pro é cortado juntamente com o N-terminal His-tag, fornecendo MMP-3cd ativo para uso imediato em inúmeras aplicações in vitro . Este método não requer equipamentos caros ou proteínas de fusão complexas e descreve a rápida produção de MMPs humanos recombinantes em bactérias.
Introduction
A maioria das proteínas eucarióticas complexas sofrem modificações pós-transicionais elaboradas após a expressão, exigindo dobramento de proteína altamente assistida e cofatores para serem funcionais1. Produzir grandes quantidades de proteína humana solúvel em um hospedeiro bacteriano continua sendo um desafio significativo devido aos altos custos e à falta de métodos robustos de expressão e purificação, mesmo para experimentos laboratoriais de menor escala2,3. OS MMPs, endopeptidases humanas com grande peso molecular, são geralmente expressos como corpos de inclusão insolúveis quando expressos em E. coli. A extração de MMPs humanos solúveis muitas vezes leva a um processo de solubilização e reabascamento laborioso e demorado4.
Os MMPs têm papéis críticos em processos fisiológicos e patogênicos. Os MMPs humanos são uma família de 23 endopeptidases de zinco, categorizadas pela especificidade de estrutura e substrato, e expressas diferencialmente apesar de um domínio catalítico altamente conservado5,6. Os MMPs são secretados como zymogens inativos, regulados via ativação pós-translacional e seus inibidores endógenos, inibidores de tecidos de metaloproteinas (TIMPs)7,8,9,10. Embora inicialmente reconhecidos por seu papel na rotatividade do ECM, os MMPs também foram implicados no desenvolvimento, morfogênese, reparação de tecidos e remodelação8. A desregulação dos MMPs tem sido notavelmente ligada ao câncer, juntamente com doenças neurodegenerativas, cardiovasculares e fibrosas, entre outras doenças5,7.
O desenvolvimento de métodos robustos de produção de MMP em larga escala é fundamental para garantir o sucesso de estudos futuros de mecanismos MMP por meio de ensaios bioquímicos e baseados em células. Vários MMPs foram previamente expressos em bactérias11, incluindo MMPs com marca Hisx6, sem alterar a atividade MMP12,13,14,15. No entanto, esses métodos incluem passos longos e tediosos que podem ser difíceis de replicar.
As células mamíferas também podem ser usadas para expressar muitas proteínas humanas diferentes, garantindo as modificações pós-transacionais adequadas16. Embora o sistema de expressão de mamíferos seja a escolha ideal para produzir proteínas humanas recombinantes com modificações pós-translacionais adequadas, as principais desvantagens deste método são os baixos rendimentos iniciais, meios de crescimento caros e reagentes, longas linhas temporais para alcançar linhas de expressão estáveis e risco de contaminação com outras espécies, como fungos ou bactérias2,11 . Além disso, a produção de MMP em linhas de células de mamíferos produz impurezas de proteínas celulares associadas, como TIMPs ou fibronectinas11. Ao contrário do lento crescimento celular observado em células mamíferas, o sistema de expressão bacteriana oferece produção de proteínas em larga escala em um curto período, juntamente com requisitos mais simples de mídia e crescimento. No entanto, devido à falta de outras proteínas celulares associadas (ou seja, TIMPs) em sistemas de expressão bacteriana, os MMPs ativos em concentrações mais elevadas estão sujeitos à degradação por meio da autoproteólise, resultando em baixo rendimento de MMP17.
Este artigo descreve um método detalhado para expressão bacteriana, purificação e ativação do recombinante Hisx6-pro-MMP-3cd usando E. coli como hospedeiro de expressão devido à sua acessibilidade, simplicidade e sucesso na produção de maiores rendimentos de MMPs2,3,18. Uma vez que a E. coli não tem o maquinário dobrável de proteínas e o processamento pós-transicional necessário para MMPs recombinantes e outras proteínas complexas, muitas cepas de E. coli foram projetadas para superar essas limitações, tornando a E. coli um hospedeiro mais adequado para a expressão de MMP-3cd humano recombinante,19,20 . Por exemplo, a cepa R2DP usada neste estudo aumenta a expressão eucariótica fornecendo um plasmídeo resistente a clororamfenícol contendo códons raramente usados em E. coli.
Como descrito neste protocolo, após a superexpressão de corpos de inclusão relativamente puros do vetor pET-3a (Figura 1) em células R2DP, proteínas do domínio catalítico Hisx6-pro-MMP-3 (MMP-3cd) são extraídas e desnaturadas4. Hisx6-pro-MMP-3cd3,19 foi purificado usando cromatografia de tag de afinidade. Após a redobramento e diálise, o pro-MMP-3cd (zymogen) foi ativado por acetato 4-aminofenilmerárico (APMA), e a análise SDS-PAGE é usada para avaliar os rendimentos e a necessidade de mais purificação5,21. Este protocolo descreve a expressão, purificação e ativação do MMP-3cd solúvel como exemplo. No entanto, também pode ser usado como guia para a expressão de outros MMPs e proteases humanas com expressão semelhante, e mecanismos de ativação (Figura 2). Para outras proteínas além do MMP-3cd, o leitor é aconselhado a determinar as composições e métodos de buffer ideais para sua proteína alvo antes de tentar este protocolo.
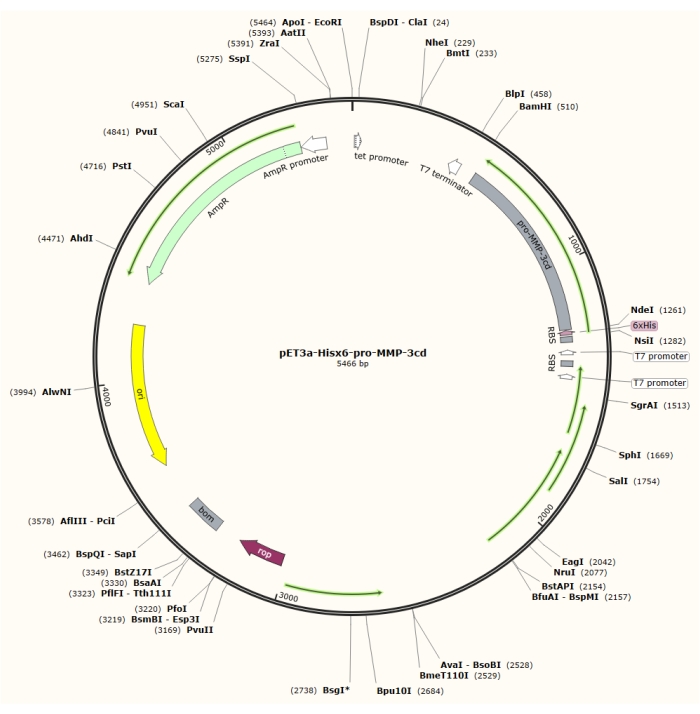
Figura 1: Mapa plasmídeo do pET-3a-Hisx6-pro-MMP-3cd plasmid. O vetor pET-3a inclui um gene de resistência à ampicilina. Uma sequência de tags Hisx6-terminal N é clonada no vetor baseado em pET-3a, incluindo pró-MMP-3cd, para produzir a construção pET-3a-Hisx6-pro-MMP-3cd sob controle do promotor T7 entre os locais de restrição BamHI e NdeI. Clique aqui para ver uma versão maior desta figura.
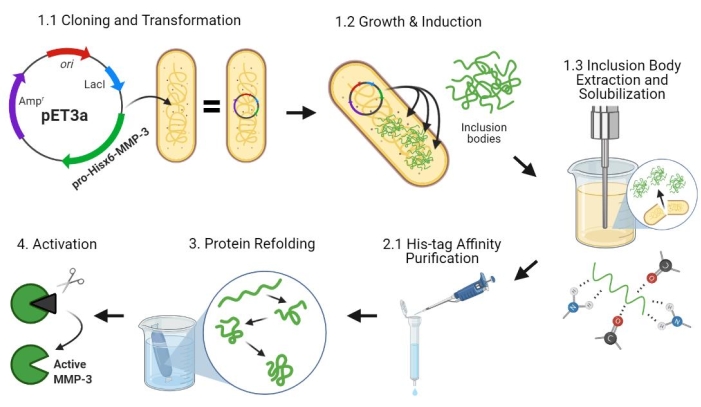
Figura 2: Expressão bacteriana de pro-MMP-3cd, purificação, repato e ativação. 1.1: pET-3a-Hisx6-pro-MMP-3cd plasmid foi transformado em células BL21(DE3) ou R2DP. 1.2: A expressão proteica Pró-MMP-3cd foi induzida por IPTG. 1.3: A lise química e a sônica são utilizadas para extrair proteínas Hisx6-pro-MMP-3cd que são principalmente insolúveis e encontradas nos corpos de inclusão. A ureia foi usada para desnaturar e solubilizar proteínas de corpos de inclusão. 2.1. A proteína Hisx6-pro-MMP-3cd foi purificada por meio da purificação da cromatografia de afinidade. 3. O Hisx6-pro-MMP-3cd elucido foi lentamente redobrado durante a diálise através da remoção gradual da ureia do buffer. 4. Finalmente, a proteína MMP-3cd repato foi ativada usando APMA removendo o domínio pró-peptídeo n-terminal. A APMA é posteriormente removida da solução através da desalting. Os números correspondem a seções de protocolo descrevendo essas etapas. Abreviaturas: MMP-3cd = Domínio catalítico matrix metaloproteinase-3; APMA = acetato aminofenilmerálico. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
A produção em larga escala de MMPs solúveis, humanos e recombinantes continua sendo uma tarefa desafiadora. As células mamíferas podem expressar MMPs funcionais a altos custos e longos tempos de espera, enquanto a E. coli produz rapidamente altas quantidades de corpos de inclusão de MMP que devem ser purificados e reobsecados11,16. As células R2DP aumentam significativamente o rendimento dos organismos de inclusão de MMP, permitindo um processo d…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Os autores gostariam de reconhecer a Dra. Evette Radisky e Alexandra Hockla na Clínica Mayo em Jacksonville, Flórida, por fornecer o pET-3a-pro-MMP-3cd plasmid como o modelo para clonagem do gene Hisx6pro-MMP-3cd, e seus comentários, juntamente com o Dr. Paul Hartley do Centro de Genômica de Nevada da Universidade de Nevada, Reno, para sequenciamento de DNA. Os autores também gostariam de agradecer Cassandra Hergenrader por ajudar com parte da expressão proteica. M.R.-S. gostaria de agradecer ao NIH-P20 GM103650-COBRE Integrative Neuroscience grant e ao Prêmio UNR P&D mICRO SEED Grant.
Materials
| 0.22 µm sterile filter | Sigma Aldrich | SLGP033RS | Used to remove some contaminants from the protein extract before purification, and prevent the Ni-NTA column from clogging |
| 1 L Erlenmeyer flasks | Thermo Fisher Scientific | S76106F | n/a |
| 1 L glass bottles | Thermo Fisher Scientific | 06-414-1D | n/a |
| 1.5 mL microfuge tubes | Thermo Fisher Scientific | 02-682-002 | n/a |
| 15 mL conical tubes | Thermo Fisher Scientific | 339650 | n/a |
| 18 G, 1-in. beveled needle | Amazon | B07S7VBHM2 | Used in combination with the dialysis casette |
| 2 mL desalting column | Thermo Fisher Scientific | 89890 | Removes APMA following activation |
| 2-(N-Morpholino)ethanesulfonic acid (MES) | Thermo Fisher Scientific | AAA1610422 | n/a |
| 250 mL conical bottle cushions | Thermo Fisher Scientific | 05-538-53A | Stabilize conical bottles during large-volume centrifugation |
| 250 mL conical bottles | Thermo Fisher Scientific | 05-538-53 | n/a |
| 400 mL stirred cell | Sigma Aldrich | UFSC40001 | Re-concentrates a much larger volume than the centrifugal filter unit. Rosetta2(DE3)pLysS cells produce high volumes of protein that may exceed the 15 mL limit of the centrifugal filter unit |
| 4-aminophenylmercuric acetate (APMA) | Sigma Aldrich | A9563-5G | Activates MMP-3 by cleaving the propeptide |
| 5 mL syringe | Thermo Fisher Scientific | NC0829167 | Used in combination with the dialysis casette |
| 50 mL conical tubes | Thermo Fisher Scientific | 339650 | Used for storage in many purification steps |
| 50 mL re-concentration tube | Sigma Aldrich | UFC901024D | Used for re-concentrating protein samples after dialysis or removing contaminants |
| Agar | Thermo Fisher Scientific | BP1423-500 | Buffer ingredient that solidifies autoclaved LB media upon cooling |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | Antibiotic used with pET3a vector; used at 100 µg/mL in LB media |
| BamHI | NEB | R3136S | Restriction enzyme to be used with the pET3a vector |
| Calcium chloride (CaCl2) | Thermo Fisher Scientific | 600-30-23 | The calcium ion stabilizes MMP structure |
| Cell spreaders | Thermo Fisher Scientific | 50-189-7544 | Can be used to spread cells across a petri dish after transformation |
| Chloramphenicol | Thermo Fisher Scientific | 22-055-125GM | Antibiotic used with pET3a vector; used at 34 µg/mL in LB media |
| Dialysis Buffer 1 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 4 M Urea. |
| Dialysis Buffer 2 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2, 1 µM ZnCl2, 2 M Urea. |
| Dialysis Buffer 3 | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 150 mM NaCl, 10 mM CaCl2 , 1 µM ZnCl2. |
| Dialysis clips | Thermo Fisher Scientific | 68011 | Used in combination with snakeskin dialysis tubing |
| Dialysis tubing | Thermo Fisher Scientific | 88243 | Alternative dialysis method that holds much larger sample volumes, but with higher risk of sample loss |
| Digest buffer | NEB | B7204S | Buffer used in digesting the pET3a vector |
| Disposable cuvettes | Thermo Fisher Scientific | 21-200-257 | Used to measure the bacterial culture OD during growth and expression |
| Dithiothreitol (DTT) | Thermo Fisher Scientific | D107125G | Assists with protein denaturation by reducing any disulfide bonds |
| DNA assembly mix | NEB | E2621S | Used to ligate the Hisx6-pro-MMP-3cd PCR product and digested pET3a vector |
| DNase I | NEB | M0303S | Endonuclease for degrading unfavorable DNA contaminants that could later affect protein purification |
| Ethanol | Thermo Fisher Scientific | A995-4 | n/a |
| Ethylenediaminetetraacetic acid (EDTA) | Thermo Fisher Scientific | J15694-AE | Used in denaturation. Prevents oxidation and subsequent formation of disulfide bonds |
| Gel recovery kit | Promega | A9281 | Isolates and purifies DNA from agarose gels |
| Glycerol | Thermo Fisher Scientific | G33-500 | Used for making glycerol stocks, which are frozen at -80 °C |
| Gravity flow column | BioRad | 7321010 | Used for Ni-NTA purification of recombinantly His-tagged proteins |
| Guanidine hydrochloride (GdnHCl) | Thermo Fisher Scientific | AAA135430B | Second chaotropic agent used for disrupting protein secondary structure. |
| High-transformation efficiency cells | NEB | C2987 | High-transformation efficiency cells with greater chance of success for cloning the N-terminal His-tag into the pET3a-pro-MMP-3cd construct |
| HT Elution Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 250 mM imidazole. Adjust pH to 7.4 |
| HT Equilibration Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea. Adjust pH to 7.4 |
| HT Regeneration Buffer | n/a | n/a | 20 mM MES, 0.1 M NaCl. Adjust pH to 5.0 |
| HT Wash Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 6 M urea, 25 mM imidazole. Adjust pH to 7.4 |
| Hydrochloric acid (HCl) | Thermo Fisher Scientific | A144C-212 | Used to pH buffers |
| Imidazole | Thermo Fisher Scientific | AAA1022122 | Mimics the histidine side group. Used to separate non-specifically binding proteins from the his-tagged target protein |
| Inclusion Body Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 5 mM DTT, 2% v/v Triton X 100, 0.5 M Urea. Adjust pH to 8.0 |
| Isopropyl-ß-D-thiogalactopyranoside (IPTG) | Thermo Fisher Scientific | FERR0392 | A reagent that induces target gene expression in pET3a. Make 0.5 mL 1 M aliquots, filter sterilize and store in -20 °C |
| LB Amp CamR media | n/a | n/a | To be poured into a sterible 1 L bottle or 1 L flask. For 1 L, add 25 g LB Broth. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Amp CamR plates | n/a | n/a | To be poured into sterile petri dishes. Pour until the petri dish lid is completely covered. 1 L of media yields 40-60 plates. For 1 L: 25 g LB Broth, 16 g Agar. Sterilize by autoclaving. Once cooled to below 50 °C, add ampicillin to 100 µg/mL and chloramphenicol to 34 µg/mL |
| LB Broth | Thermo Fisher Scientific | BP1426-2 | Pre-mixed with tryptone, yeast extract, and sodium chloride |
| Lysis Buffer | n/a | n/a | 50 mM Tris-HCl (pH 8.0), 1 mM EDTA, 100 mM NaCl, 0.133 g/mL lysozyme, 0.49% v/v Triton X-100. Adjust pH to 8.0 |
| Lysozyme | MP Biomedicals | 195303 | Used in protein extraction. Enzyme that lyses bacterial cell walls |
| Miniprep kit | Promega | A1330 | If successful, extracts the pET3a-pro-MMP-3cd construct from transformants |
| NdeI | NEB | R0111S | Restriction enzyme to be used with the pET3a vector |
| Ni-NTA resin | Thermo Fisher Scientific | PI88221 | Used to bind recombinant his-tagged proteins. This strong interaction can be displaced with higher concentrations of imidazole |
| PCR mix | NEB | M0492S | A PCR reagent for inserting an N-terminal his-tag into the pET3a-pro-MMP-3cd vector |
| pET plasmid | Addgene | n/a | The pET3a vector offers ampicillin resistance, inducible expression of a target gene, and sequencing with T7 primers |
| Petri dishes | VWR | 25384-342 | Used for plating transformants on LB agar media |
| R2DP cells | Novagen | 714033 | BL21 derivatives with enhanced expression of eukaryotic proteins. Contain tRNAs of codons found to be rare in e. coli |
| SOC growth media | NEB | B9020S | Non-selective growth media for rapid growth during transformation |
| Sodium chloride (NaCl) | Thermo Fisher Scientific | BP358-1 | Used in buffers and helps with protein stability |
| Sodium deoxycholate | Thermo Fisher Scientific | PI89905 | Detergent used in protein extraction. Lyses cell walls |
| Solubilization Buffer | n/a | n/a | 20 mM Tris-HCl (pH 8.0), 50 mM NaCl, 10 mM DTT, 6 M Urea. Adjust pH to 8.0 |
| Tris base | Thermo Fisher Scientific | BP152-1 | Common buffer used in the physiological pH range. Temperature-sensitive |
| Triton X-100 | Thermo Fisher Scientific | M1122980101 | Detergent used for cell lysis |
| Urea | Thermo Fisher Scientific | AAJ75826A7 | First chaotropic agent for disrupting protein secondary structure |
| Zinc chloride (ZnCl2) | Thermo Fisher Scientific | AAA162810E | Stabilizes MMP structure. The zinc ion is found in the catalytic site of MMP-3 |
Riferimenti
- Portolano, N., et al. Recombinant protein expression for structural biology in HEK 293F suspension cells: A novel and accessible approach. Journal of Visualized Experiments: JoVE. (92), e51897 (2014).
- Subedi, G. P., Johnson, R. W., Moniz, H. A., Moremen, K. W., Barb, A. High yield expression of recombinant human proteins with the transient transfection of HEK293 cells in suspension. Journal of Visualized Experiments: JoVE. (106), e53568 (2015).
- Nilvebrant, J., Alm, T., Hober, S. Orthogonal protein purification facilitated by a small bispecific affinity tag. Journal of Visualized Experiments: JoVE. (59), e3370 (2012).
- Yang, Z., et al. Highly efficient production of soluble proteins from insoluble inclusion bodies by a two-step-denaturing and refolding method. PLoS One. 6 (7), 22981 (2011).
- Hu, X., Beeton, C. Detection of functional matrix metalloproteinases by zymography. Journal of Visualized Experiments: JoVE. (45), e2445 (2010).
- Radisky, E. S., Raeeszadeh-Sarmazdeh, M., Radisky, D. C. Therapeutic potential of matrix metalloproteinase inhibition in breast cancer. Journal of Cellular Biochemistry. 118 (11), 3531-3548 (2017).
- Raeeszadeh-Sarmazdeh, M., Do, L. D., Hritz, B. G. Metalloproteinases and their inhibitors: Potential for the development of new therapeutics. Cells. 9 (5), 1313 (2020).
- Nagase, H., Visse, R., Murphy, G. Structure and function of matrix metalloproteinases and TIMPs. Cardiovascular Research. 69 (3), 562-573 (2006).
- Raeeszadeh-Sarmazdeh, M., et al. Directed evolution of the metalloproteinase inhibitor TIMP-1 reveals that its N- and C-terminal domains cooperate in matrix metalloproteinase recognition. Journal of Biological Chemistry. 294 (24), 9476-9488 (2019).
- Batra, J., et al. Matrix metalloproteinase-10 (MMP-10) interaction with tissue inhibitors of metalloproteinases TIMP-1 and TIMP-2. Journal of Biological Chemistry. 287 (19), 15935-15946 (2012).
- Singh, K. K., Jain, R., Ramanan, H., Saini, D. K., Galea, C. A. Expression and purification of matrix metalloproteinases in Escherichia coli. Matrix Metalloproteases. , 3-16 (2017).
- Manka, S. W., et al. Structural insights into triple-helical collagen cleavage by matrix metalloproteinase 1. Proceedings of the National Academy of Sciences of the United States of America. 109 (31), 12461-12466 (2012).
- Gomis-Ruth, F. X., et al. Mechanism of inhibition of the human matrix metalloproteinase stromelysin-1 by TIMP-1. Nature. 389 (6646), 77-81 (1997).
- Shirian, J., et al. Converting a broad matrix metalloproteinase family inhibitor into a specific inhibitor of MMP-9 and MMP-14. FEBS Letters. 592 (7), 1122-1134 (2018).
- Li, C., et al. Purification of recombinant histidine-tagged catalytic domain of MMP-13 in one step using affinity column and renaturation of it with histidine tag. Journal of Liquid Chromatography & Related Technologies. 37 (15), 2118-2130 (2014).
- Aydin, H., Azimi, F. C., Cook, J. D., Lee, J. E. A convenient and general expression platform for the production of secreted proteins from human cells. Journal of Visualized Experiments: JoVE. (65), e4041 (2012).
- McNiff, M. L., Haynes, E. P., Dixit, N., Gao, F. P., Laurence, J. S. Thioredoxin fusion construct enables high-yield production of soluble, active matrix metalloproteinase-8 (MMP-8) in Escherichia coli. Protein Expression and Purification. 122, 64-71 (2016).
- Maity, R., et al. GST-His purification: A two-step affinity purification protocol yielding full-length purified proteins. Journal of Visualized Experiments: JoVE. (80), e50320 (2013).
- Stefan, A., Ceccarelli, A., Conte, E., Montón Silva, A., Hochkoeppler, A. The multifaceted benefits of protein co-expression in Escherichia coli. Journal of Visualized Experiments: JoVE. (96), e52431 (2015).
- Yadavalli, R., Sam-Yellowe, T. HeLa based cell free expression systems for expression of Plasmodium rhoptry proteins. Journal of Visualized Experiments: JoVE. (100), e52772 (2015).
- Zeytuni, N., Zarivach, R. Purification of the M. magneticum strain AMB-1 magnetosome associated protein MamAΔ41. Journal of Visualized Experiments: JoVE. (37), e1844 (2010).

