تنقية بوليسوم من العقيدات التكافلية لفول الصويا
Summary
يصف هذا البروتوكول طريقة لتنقية متعدد حقيقيات النواة من عقيدات فول الصويا السليمة. بعد التسلسل ، يمكن استخدام خطوط الأنابيب القياسية لتحليل التعبير الجيني لتحديد الجينات المعبر عنها بشكل تفاضلي على مستويات النسخ و translatome.
Abstract
الهدف من هذا البروتوكول هو توفير استراتيجية لدراسة ترانسلاتوم حقيقيات النواة للعقيدات التكافلية لفول الصويا (الجلايسين ماكس). تصف هذه الورقة الطرق المحسنة لعزل البوليريبوسومات المشتقة من النباتات والحمض النووي الريبوزي المرسال المرتبط بها ليتم تحليلها باستخدام تسلسل الحمض النووي الريبي. أولا ، يتم الحصول على التحلل السيتوبلازمي من خلال التجانس في ظروف الحفاظ على الحمض النووي الريبي والحمض النووي الريبي من عقيدات فول الصويا الكاملة المجمدة. بعد ذلك ، يتم مسح الليزات عن طريق الطرد المركزي منخفض السرعة ، ويتم استخدام 15٪ من المادة الفائقة لعزل الحمض النووي الريبي الكلي (TOTAL). يتم استخدام المحللات المتبقية التي تم تطهيرها لعزل البوليسومات عن طريق الطرد المركزي الفائق من خلال وسادة السكروز المكونة من طبقتين (12٪ و 33.5٪). يتم تنقية الحمض النووي الريبوزي المرسال المرتبط بالبوليسومات (PAR) من الكريات متعددة الصبغيات بعد إعادة التعليق. يتم تقييم كل من TOTAL و PAR بواسطة الرحلان الكهربائي الشعري عالي الحساسية لتلبية معايير الجودة لمكتبات التسلسل ل RNA-seq. كمثال على تطبيق المصب ، بعد التسلسل ، يمكن استخدام خطوط الأنابيب القياسية لتحليل التعبير الجيني للحصول على جينات معبر عنها بشكل تفاضلي على مستويات النسخ و translatome. باختصار ، تسمح هذه الطريقة ، بالاقتران مع RNA-seq ، بدراسة التنظيم الانتقالي للحمض النووي الريبي المرسال حقيقيات النواة في نسيج معقد مثل العقيدات التكافلية.
Introduction
يمكن للنباتات البقولية ، مثل فول الصويا (Glycine max) ، أن تنشئ تكافلا مع بكتيريا تربة محددة تسمى ريزوبيا. هذه العلاقة المتبادلة تثير تشكيل أعضاء جديدة ، العقيدات التكافلية ، على جذور النباتات. العقيدات هي الأعضاء النباتية التي تستضيف البكتيريا وتتكون من خلايا مضيفة يستعمر السيتوبلازم الخاص بها مع شكل متخصص من الجذور يسمى البكتيريا. تحفز هذه البكتيريا على تقليل النيتروجين الجوي (N 2) إلى الأمونيا ، والتي يتم نقلها إلى النبات مقابل الكربوهيدرات 1,2.
على الرغم من أن هذا التكافل المثبت للنيتروجين هو واحد من أكثر التكافل بين النبات والميكروبات دراسة جيدة ، إلا أنه لا يزال يتعين فهم العديد من الجوانب بشكل أفضل ، مثل كيفية قيام النباتات المعرضة لظروف الإجهاد اللاأحيائي المختلفة بتعديل تفاعلها مع شريكها التكافلي وكيف يؤثر ذلك على استقلاب العقيدات. ويمكن فهم هذه العمليات بشكل أفضل من خلال تحليل ترانسلاتوم العقيدات (أي المجموعة الفرعية من الحمض النووي الريبي المرسال [mRNAs] التي تترجم بنشاط). البولي ريبوسومات أو البوليسومات هي مجمعات من الريبوسومات المتعددة المرتبطة بالحمض النووي الريبوزي المرسال ، وتستخدم عادة لدراسة الترجمة3. تتكون طريقة التنميط المتعدد من تحليل الحمض النووي الريبوزي المرسال المرتبط ب polysomes وقد تم استخدامه بنجاح لدراسة آليات ما بعد النسخ التي تتحكم في التعبير الجيني الذي يحدث في العمليات البيولوجية المتنوعة 4,5.
تاريخيا ، ركز تحليل التعبير الجيني في المقام الأول على تحديد وفرة الحمض النووي الريبوزي المرسال6،7،8،9. ومع ذلك ، هناك نقص في العلاقة بين مستويات النسخ والبروتين بسبب المراحل المختلفة لتنظيم ما بعد النسخ للتعبير الجيني ، وخاصة الترجمة 10،11،12. علاوة على ذلك ، لم يلاحظ أي اعتماد بين التغييرات على مستوى النسخ وتلك التي تحدث على مستوى translatome13. يسمح التحليل المباشر لمجموعة الحمض النووي الريبوزي المرسال التي يتم ترجمتها بقياس أكثر دقة واكتمالا للتعبير الجيني للخلية (الذي تكون نقطة نهايته وفرة البروتين) من ذلك الذي تم الحصول عليه عندما يتم تحليل مستويات الحمض النووي الريبوزي المرسال فقط14،15،16.
يصف هذا البروتوكول كيفية تنقية البوليسومات المشتقة من النباتات من عقيدات فول الصويا السليمة عن طريق الطرد المركزي التفاضلي من خلال وسادة السكروز المكونة من طبقتين (الشكل 1). ومع ذلك ، نظرا لأن الريبوسومات المشتقة من البكتيريا موجودة أيضا في العقيدات ، يتم تنقية مزيج من أنواع الريبوسومات والحمض النووي الريبي ، على الرغم من أن الأنواع حقيقية النواة تمثل الجزء الرئيسي (90٪ -95٪). كما يتم وصف عزل الحمض النووي الريبي اللاحق ، والقياس الكمي ، ومراقبة الجودة (الشكل 1). يجب أن يوفر هذا البروتوكول ، بالاقتران مع RNA-seq ، نتائج تجريبية حول التنظيم الانتقالي للحمض النووي الريبي المرسال حقيقيات النواة في نسيج معقد مثل العقيدات التكافلية.
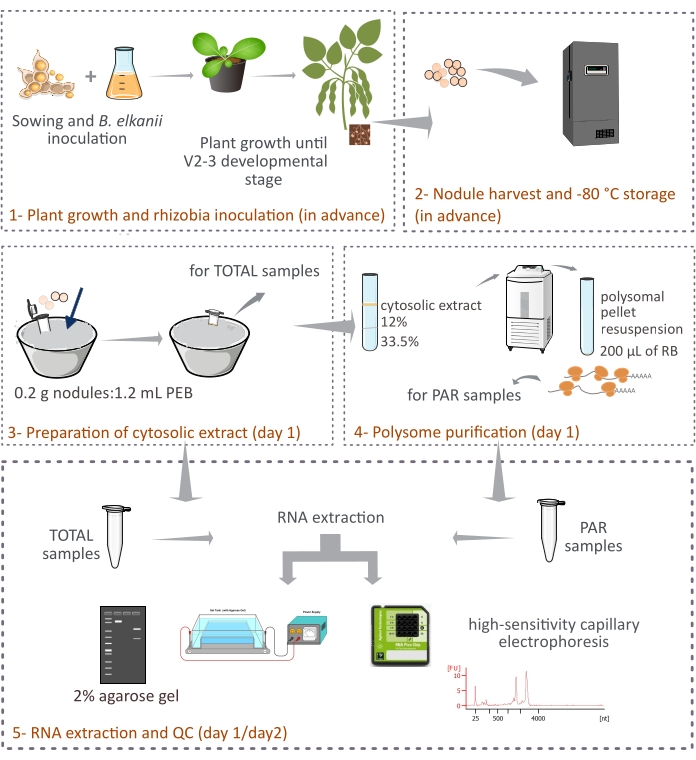
الشكل 1: نظرة عامة تخطيطية على المنهجية المقترحة لتنقية تنقية الصبغات متعددة حقيقيات النواة من العقيدات التكافلية. يقدم المخطط لمحة عامة عن الخطوات المتبعة في البروتوكول من (1) نمو النبات و (2) حصاد العقيدات إلى (3) إعداد المستخلصات الخلوية ، (3) الحصول على عينات TOTAL و (4) عينات PAR ، و (5) استخراج الحمض النووي الريبي ومراقبة الجودة. الاختصارات: PEB = مخزن مؤقت لاستخراج polysome ؛ RB = المخزن المؤقت لإعادة التعليق; المجموع = إجمالي الحمض النووي الريبي. PAR = الحمض النووي الريبوزي المرسال المرتبط بالبوليسومات. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
Representative Results
Discussion
تعد دراسة تنظيم التعبير الجيني على المستوى الانتقالي أمرا بالغ الأهمية لفهم العمليات البيولوجية المختلفة بشكل أفضل لأن نقطة النهاية للتعبير الجيني للخلية هي وفرة البروتين13,14. يمكن تقييم ذلك من خلال تحليل translatome للنسيج أو الكائن الحي محل الاهتمام الذي يجب ?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
تم تمويل هذا البحث من قبل منحة CSIC I + D 2020 رقم 282 ، ومنحة FVF 2017 رقم 210 ، و PEDECIBA (ماريا مارثا ساينز).
Materials
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
Riferimenti
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer’s disease model mice. Journal of Alzheimer’s Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).

