Фенотипическое профилирование дофаминергических нейронов среднего мозга человека, полученных из стволовых клеток человека
Summary
Этот протокол описывает культивирование клеток дофаминергических нейронов среднего мозга человека с последующим иммунологическим окрашиванием и генерацией нейрональных фенотипических профилей из полученных микроскопических изображений с высоким содержанием, позволяющих идентифицировать фенотипические вариации, обусловленные генетическими или химическими модуляциями.
Abstract
Болезнь Паркинсона (БП) связана с рядом клеточных биологических процессов, которые вызывают потерю дофаминергических нейронов среднего мозга (мДА). Многие современные клеточные модели БП in vitro недостаточно сложны и не учитывают множественные фенотипы. Фенотипическое профилирование нейронов мДА, полученных из индуцированных плюрипотентных стволовых клеток человека (iPSC), может устранить эти недостатки путем одновременного измерения ряда фенотипов нейронов в PD-релевантном типе клеток. В данной статье мы опишем протокол получения и анализа фенотипических профилей коммерчески доступных нейронов мДА человека. Нейрон-специфическая флуоресцентная окрашивающая панель используется для визуализации фенотипов, связанных с ядерным α-синуклеином, тирозингидроксилазой (ТГ) и микротрубочками-ассоциированным белком 2 (MAP2). Описанный протокол фенотипического профилирования является масштабируемым, так как в нем используются 384-луночные планшеты, автоматическая работа с жидкостями и высокопроизводительная микроскопия. Полезность протокола проиллюстрирована на примере здоровых донорских нейронов mDA и нейронов mDA, несущих PD-сцепленную мутацию G2019S в гене лейцин-богатой повторной киназы 2 (LRRK2). Обе клеточные линии обрабатывали ингибитором киназы LRRK2 PFE-360 и измеряли фенотипические изменения. Кроме того, мы демонстрируем, как многомерные фенотипические профили могут быть проанализированы с помощью кластеризации или методов классификации с учителем на основе машинного обучения. Описанный протокол будет особенно интересен исследователям, работающим над моделированием заболеваний нейронов или изучающими эффекты химических соединений в нейронах человека.
Introduction
При болезни Паркинсона (БП) нарушаются различные клеточные биологические процессы. Например, митохондриальная дисфункция, окислительный стресс, дефекты деградации белков, нарушение везикулярного транспорта и эндолизосомальной функции, связанные с потерей дофаминергических нейронов среднего мозга (мДА), обычно наблюдаются при БП1. Таким образом, болезнь Паркинсона, по-видимому, включает в себя несколько механизмов заболевания, которые могут взаимодействовать друг с другом и ухудшать друг друга. Одним из полезных способов исследования этого механистического взаимодействия является создание комплексного фенотипического отпечатка пальца или профиля дофаминергических нейронов среднего мозга (mDA).
Фенотипическое профилирование – это подход, который включает в себя создание профиля выборки на основе набора измеримых характеристик и, во-вторых, прогнозирование выборки на основе этого профиля 2,3. Цель профилирования состоит в том, чтобы зафиксировать широкий спектр признаков, некоторые из которых, возможно, ранее не были связаны с заболеванием или лечением3. В результате профилирование может выявить неожиданные биологические процессы. Фенотипическое профилирование, как правило, опирается на флуоресцентно окрашенные клетки, и для создания фенотипическихпрофилей были разработаны стандартизированные анализы, такие как Cell Painting. В последнее время фенотипическое профилирование, например, применяется для характеризации малых молекул или точного прогнозирования подтипов БП исключительно на основе фибробластов, полученных от пациентов 5,6. Несмотря на эти достижения, фенотипическое профилирование редко применялось к постмитотическим дифференцированным клеткам, таким как индуцированные плюрипотентные стволовые клетки человека (iPSC) нейроны mDA, которые экспрессируют PD-сцепленные мутации, такие как LRRK2 G2019S. К существенным проблемам моделей, полученных на основе ИПСК, относятся наличие малозаметных или вариабельных патологических признаков в группах дифференцировки или генотипах, а также тот факт, что изолированные фенотипы БП не охватывают всю сложность заболевания. Кроме того, несмотря на то, что нейронные модели ИПСК являются физиологически значимыми, они редко используются в процессах разработки лекарств для лечения БП из-за опасений по поводу технической сложности 7,8.
Ранее мы разработали надежную методологию для измерения нескольких патофизиологических фенотипов, связанных с болезнью Паркинсона, в нейронах мДА человека, которые чувствительны как к генетическим, так и к химическим соединениям фенотипическимизменениям. В данной статье подробно описывается дальнейшая оптимизированная версия этой методики для создания фенотипических профилей из нейронов mDA (рис. 1). Этот протокол имеет ряд преимуществ по сравнению с ранее описанными подходами к фенотипическому профилированию, такими как использование высококачественных нейронов mDA и техническая воспроизводимость. Впервые этот протокол позволяет осуществлять фенотипическое профилирование физиологически значимых постмитотических нейронов mDA после химических возмущений с высокой степенью масштабируемости. Полностью дифференцированные и криоконсервированные нейроны mDA коммерчески доступны, что значительно снижает вариабельность дифференцировки от партии к партии. Во-вторых, техническая вариабельность может быть дополнительно снижена за счет использования четко определенного плана эксперимента (т.е. продолжительности культивирования или отказа от краевых лунок), автоматизированной обработки жидкостей и автоматизированной микроскопии. Кроме того, здесь описаны начальные этапы анализа фенотипического профиля с использованием неконтролируемой кластеризации или контролируемой классификации, а также указано, как можно анализировать данные фенотипического профилирования. Этот протокол будет полезен исследователям, интересующимся фенотипическими изменениями нейронов mDA, индуцированными генетическими или химическими возмущениями, в частности, когда требуется высокомасштабируемая исследовательская установка, например, во время скрининговых кампаний или когда необходимо изучить влияние меньшего числа соединений, например, для определения токсических эффектов. Таким образом, предполагается, что применение фенотипического профилирования нейронов человека является ценным методом для изучения сложных фенотипов, связанных с заболеванием, и характеристики клеточных эффектов кандидатов в лекарственные препараты.
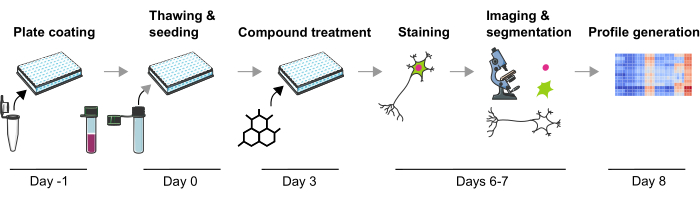
Рисунок 1: Схематическое изображение экспериментального протокола для создания фенотипических профилей на основе изображений из нейронов мДА человека, полученных из ИПСК. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
Protocol
Representative Results
Discussion
Фенотипическое профилирование — это метод измерения большого числа фенотипов в клетках с помощью флуоресцентного окрашивания, микроскопии и анализа изображений3. Фенотипические профили могут быть получены и сравнены между клеточными линиями или другими эксперименталь?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Авторы хотели бы поблагодарить всех коллег из «Ксилинк» за их ценную помощь и обсуждения, которые привели к разработке представленного протокола.
Materials
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse – Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit – Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
Riferimenti
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson’s disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson’s disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson’s Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson’s Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson’s disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson’s disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson’s disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson’s disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).

