Generación de fusiones de proteínas fluorescentes en<em> Candida</em> Especies
Summary
La modificación genética mediada por PCR se puede utilizar para generar fusiones de proteínas fluorescentes en especies de Candida, lo que facilita la visualización y cuantificación de células de levadura y las proteínas. Aquí, se presenta una estrategia para la construcción de una fusión proteína fluorescente (Eno1-FP) en Candida parapsilosis.
Abstract
Las especies de Candida, colonizadores frecuentes de los tractos intestinal y genitourinario, son la causa de la mayoría de las infecciones fúngicas invasivas en seres humanos. Por lo tanto, se necesitan herramientas moleculares y genéticos para facilitar el estudio de sus mecanismos de la patogénesis. la modificación genética mediada por PCR es un método sencillo y rápido para generar proteínas etiquetadas con epítopo para facilitar su detección. En particular, la proteína fluorescente (FP) fusiones son herramientas poderosas que permiten la visualización y cuantificación de ambas células de levadura y las proteínas por microscopía de fluorescencia y la inmunotransferencia, respectivamente. Los plásmidos que contienen secuencias de codificación de PF, junto con los genes marcadores nutricionales que facilitan la transformación de las especies Candida, se han generado con el propósito de la construcción FP y expresión en Candida. Aquí, se presenta una estrategia para la construcción de una fusión FP en una especie de Candida. Los plásmidos que contienen el sold nourseothricinna vez que el gen marcador de transformación (NAT1) junto con secuencias para cualquiera verde, amarillo, o de cerezas FPS (GFP, YFP, mCherry) se utilizan junto con los cebadores que incluyen secuencias de genes específicos en una reacción en cadena de la polimerasa (PCR) para generar un módulo de FP . Este casete de genes específicos tiene la capacidad de integrar en el extremo 3 'del locus del gen correspondiente a través de recombinación homóloga. El éxito de fusión en el marco de la secuencia de FP en el locus de gen de interés se verifica genéticamente, seguido por el análisis de la expresión de proteína de fusión por microscopía y / o métodos de inmunodetección. Además, para el caso de proteínas altamente expresados, fusiones exitosas pueden ser examinados para principalmente por técnicas de imagen de fluorescencia.
Introduction
Las especies de Candida son comensales hongos que colonizan el tracto intestinal y genitourinario de todos los seres humanos. En condiciones de inmunodeficiencia, tales como que se producen con el nacimiento prematuro o efectos inmunosupresores de los tratamientos para el cáncer, las especies de Candida pueden llegar a ser patógenos oportunistas. De las especies de Candida, Candida albicans es el colonizador fúngica más frecuente y causa la mayoría de las infecciones fúngicas invasivas. Otras especies de Candida, como C. glabrata, C. parapsilosis, C. tropicalis y C. También kruseii causan infecciones graves en pacientes inmunocomprometidos, con alguna resistencia intrínseca que presenta a los antibióticos comúnmente utilizados antifúngicos como fluconazol y anfotericina B. Por lo tanto, las infecciones con algunas de estas especies están siendo observados con mayor frecuencia, especialmente en pacientes que están siendo tratados profilácticamente con agentes anti-hongos. Incluso con una apropiada y oportunanti tratamiento de las infecciones fúngicas, infecciones invasivas por Candida continúan siendo asociado con morbilidad y mortalidad significativa 1. Debido a la importancia de especies de Candida en la salud humana, existe una necesidad de herramientas moleculares fácilmente disponibles que permiten el estudio y la elucidación de sus mecanismos de la patogénesis.
Una herramienta importante que permite a los investigadores visualizar y cuantificar las células microbianas y las proteínas que se expresan es la tecnología de fusión FP. Reacción en cadena de la polimerasa (PCR), la modificación genética mediada, tal como se describe en este documento, permite la construcción de fusiones, entre las secuencias de PF y una proteína de Candida secuencia codificante de interés en su locus genómico. La integración estable de la construcción facilita el análisis de la expresión de proteínas, así como la dinámica de localización de proteínas. Los plásmidos que contienen secuencias de PF, optimizados para la expresión en Candida albicans y que se pueden utilizar en la g mediada por PCRestrategia de modificación eno, se han construido previamente 2, 3, 4, 5. Los plásmidos contienen FP transformación "casetes": una secuencia FP ligado a un gen marcador nutricional que facilita la transformación de C. albicans y C. parapsilosis 2, 3, 4, 5, 6, 7. Actualmente plásmidos disponibles contienen una variedad de genes marcadores seleccionables nutricionales (URA3, His1, ARG4) para la transformación de las cepas auxotróficas, así como un marcador de resistencia dominante fármaco (NAT1), que facilita la transformación de cepas clínicas que carecen de auxotrophies. Además, los plásmidos que contienen opciones para hasta cuatro secuencias diferentes (FP verde [GFP], yellow [YFP], cian [PPC], y cereza [mCherry]) y, o bien una secuencia de terminación de ADH1 para la construcción de fusiones de proteínas carboxi-terminal, o una secuencia promotora para la construcción de fusiones de proteína amino-terminal. Los cebadores están diseñados con homología con el plásmido de ADN que rodea a la casete de FP. Además, los cebadores también contienen secuencias 5 'de extensión que llevan homología con el gen de levadura de interés para ser marcados, lo que facilita la integración del casete en el locus genómico mediante recombinación homóloga (Figura 1). Cassettes de PF de genes específicos son generados por PCR y luego se transformaron en células de Candida hechas competentes para la captación de ADN por tratamiento con acetato de litio.
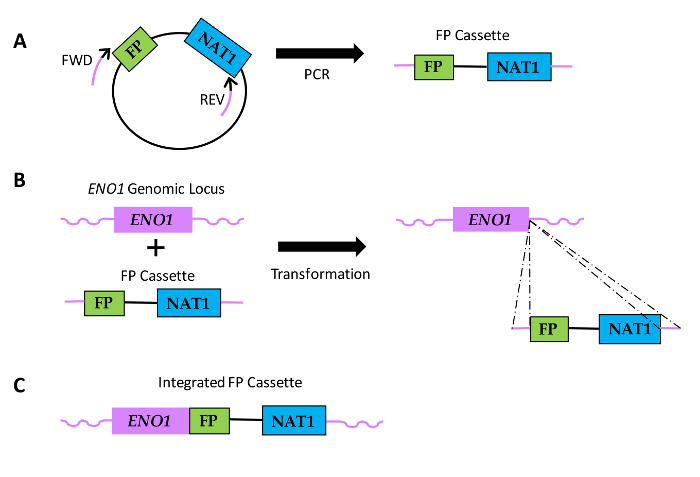
Figura 1: Diagrama de cómo FP fusiones de secuencia se generan en las especies de Candida. (A) El ADN del plásmido INCLUYENDOes una secuencia FP y una secuencia que codifica resistencia nourseothricin (NAT1). ubicaciones relativas de Forward (FWD) y atrás primers (REV) se muestran, con partes negras de los cebadores que indica la región de homología con la secuencia del plásmido y las porciones de color púrpura que denotan la región de homología con el gen específico o extensión del cebador. Casetes (B) de PF se transforman en Candida y se integran dentro del locus genómico ENO1 mediante recombinación homóloga (líneas de puntos). (C) resultante secuencia de fusión FP al extremo 3 'de ENO1. Haga clic aquí para ver una versión más grande de esta figura.
Aquí, se presenta un ejemplo de construcciones de proteínas de fusión (Eno1-FP) en especies de Candida. Nos utilizan referencia de plásmidos que contienen el gen marcador de la transformación NAT1 junto con secuencias que codifican GFP, YFP, omCherry (Figura 2). Estos plásmidos se utilizan junto con cebadores en PCR para generar casetes de genes específicos que facilitan la fusión de los programas marco para el extremo 3 'de ENO1, lo que resulta en la expresión de Eno1 fusionado a programas marco en su extremo carboxi-terminal.
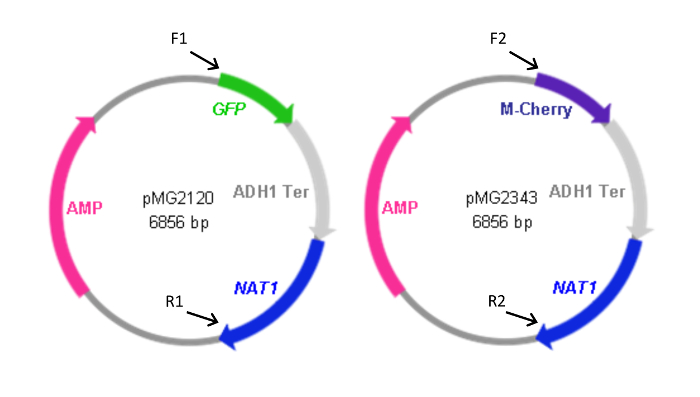
Figura 2: Mapas de los plásmidos que contienen cassettes-PF. Forward (F) y atrás primers (R) utilizados para generar los casetes de los plásmidos se indican junto con la ubicación relativa de su homología con los plásmidos. Las secuencias de cebador son como se indica en la Tabla 1. F1 y R1 también se utilizaron para generar el casete pYFP- NAT1. El plásmido que contiene el casete YFP- NAT1 (pMG2263) es idéntica a pMG2120 con la excepción de YFP en lugar de la secuencia de GFP. Los tamaños de casete: GFP-NAT1, 3,7 kbp; mCherry- NAT1, 3,2 kpb; YFP- NAT1, 3.7 kpb. Esta cifra ha sido modificado a partir de Gerami-Nejad, et al. 4 Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Construcción de epítopo etiquetado secuencias en las especies de Candida utilizando la estrategia de modificación del gen mediada por PCR descrito anteriormente se pueden resumir como un proceso de tres pasos. En primer lugar, un casete se hace por PCR que codifica tanto la secuencia deseada para la integración y regiones homólogas al locus de la inserción en el genoma de la levadura. En segundo lugar, las células de levadura para ser transformadas se hacen químicamente competente con acetato d…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Agradecemos a N. Dean para proporcionar la secuencia original mCherry FP, M. Gerami-Nejad para la construcción de plásmidos, B. Larson para la asistencia técnica, y T. Heisel útil para el asesoramiento durante el desarrollo de este proyecto. JB fue apoyado por el Premio del Consejo Europeo de Investigación Avanzada 340087 (RAPLODAPT). sistemas de microscopía e imagen fueron proporcionados por la Fundación Universidad de Minnesota Pediatría y la Universidad de Minnesota Centro de Imagen.
Materials
| 100W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5ml, 1.5ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C ) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C ) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4°C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
References
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. . Current Protocols in Molecular Biology. , (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).

