Generazione di fusioni proteina fluorescente in<em> Candida</em> Specie
Summary
Modificazione genetica PCR-mediata può essere utilizzato per generare fusioni di proteine fluorescenti a Candida specie, che facilita la visualizzazione e la quantificazione delle cellule di lievito e proteine. Qui, vi presentiamo una strategia per la costruzione di una fusione proteina fluorescente (Eno1-FP) in Candida parapsilosis.
Abstract
Specie di Candida, colonizzatori prevalente dei tratti intestinali e genito-urinario, sono la causa della maggior parte delle infezioni fungine invasive sugli esseri umani. Pertanto, sono necessari strumenti molecolari e genetici per facilitare lo studio dei loro meccanismi patogeni. PCR-mediata modificazione genetica è un metodo semplice e rapido per generare proteine epitopi-tag per facilitare la loro individuazione. In particolare, la proteina fluorescente (FP) fusioni sono potenti strumenti che consentono la visualizzazione e la quantificazione di entrambe le cellule di lievito e proteine da microscopia a fluorescenza e immunoblotting, rispettivamente. I plasmidi contenenti FP sequenze di codifica, insieme con geni marcatori nutrizionali che facilitano la trasformazione delle specie Candida, sono stati generati al fine di costruzione FP ed espressione in Candida. Qui, vi presentiamo una strategia per la costruzione di una fusione FP in una specie di Candida. I plasmidi contenenti il impermeabi nourseothricinnce gene marcatore di trasformazione (NAT1) insieme a sequenze per uno verde, giallo, o ciliegio PQ (GFP, YFP, mCherry) sono utilizzati insieme con gli iniettori che comprendono sequenze gene-specifici in una reazione a catena della polimerasi (PCR) per generare una cassetta FP . Questa cassetta-specific gene ha la capacità di integrarsi nel 3'-end del corrispondente locus genico mediante ricombinazione omologa. Successful fusione in-frame della sequenza FP nel locus gene di interesse viene verificato geneticamente, seguita da analisi dell'espressione della proteina di fusione mediante microscopia e / o metodi immuno-rilevazione. Inoltre, nel caso di proteine altamente espressi, fusioni successo possono essere esaminati per principalmente da tecniche di imaging di fluorescenza.
Introduction
Specie di Candida sono funghi commensali che colonizzano i tratti intestinali e genito-urinario di tutti gli esseri umani. In condizioni di immunodeficienza, come ad esempio che si verificano con parto prematuro o effetti immunosoppressivi di trattamenti per il cancro, specie Candida possono diventare patogeni opportunisti. Tra le specie di Candida, Candida albicans è il colonizzatore fungina più diffusa e provoca la maggior parte delle infezioni fungine invasive. Altre specie di Candida come C. glabrata, C. parapsilosis, C. tropicalis e C. kruseii anche causare gravi infezioni nei pazienti immunocompromessi, con qualche esibendo resistenza intrinseca alla usati comunemente antibiotici anti-fungine quali fluconazolo e amfotericina B. Quindi, infezioni con alcune di queste specie sono osservati più frequentemente, soprattutto nei pazienti in trattamento profilattico con farmaci anti-fungini. Anche con una appropriata e tempestivatrattamento NTI-fungine, infezioni da Candida invasive continuano ad essere associata a morbidità e mortalità 1 significativa. A causa del significato di specie Candida nella salute umana, vi è l'esigenza di strumenti molecolari prontamente disponibili che permettono lo studio e la spiegazione dei loro meccanismi patogeni.
Uno strumento importante che permette ai ricercatori di visualizzare e quantificare le cellule microbiche e le proteine che essi esprimono è tecnologia della fusione FP. Reazione a catena della polimerasi (PCR) mediata modificazione genetica, come descritto in questo documento, permette la costruzione di fusioni, tra le sequenze FP ed una proteina Candida sequenza codificante di interesse al suo locus genomico. Integrazione stabile del costrutto facilita l'analisi dell'espressione della proteina e dinamiche localizzazione della proteina. I plasmidi contenenti sequenze FP, ottimizzati per l'espressione in Candida albicans e che possono essere utilizzati nella g PCR-mediatastrategia modifica ene, è stato costruito in precedenza 2, 3, 4, 5. I plasmidi contengono FP trasformazione "cassette": una sequenza FP legato ad un gene marcatore nutrizionale che facilita la trasformazione di C. albicans e C. parapsilosis 2, 3, 4, 5, 6, 7. Attualmente plasmidi disponibili contengono una varietà di geni marcatori selezionabili nutrizionali (URA3, HIS1, Arg4) per la trasformazione di ceppi auxotrofi così come marcatore resistenza ai farmaci dominante (NAT1), che facilita la trasformazione di ceppi clinici privi auxotrophies. Inoltre, plasmidi contengono opzioni per un massimo di quattro differenti sequenze FP (verde [GFP], Yellow [YFP], ciano [CFP], e la ciliegia [mCherry]) e sia una sequenza di terminazione ADH1 per la costruzione di fusioni di proteine carbossi-terminale, o di una sequenza promotrice per la costruzione di fusioni di proteine amino-terminale. Primer sono progettati con omologia al DNA plasmidico che circonda la cassetta FP. Inoltre, i primer contengono anche sequenze 5'-estensione recanti omologia con il gene di lievito di interesse per essere etichettato, che facilita l'integrazione della cassetta nel locus genomico tramite ricombinazione omologa (Figura 1). Gene-specifici cassette FP sono generati mediante PCR e poi trasformati in cellule di Candida rese competenti per l'assorbimento di DNA mediante trattamento con acetato di litio.
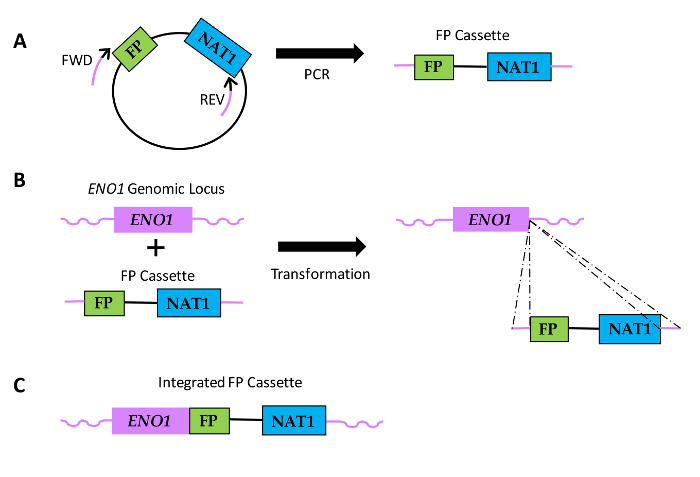
Figura 1: Diagramma di come FP fusioni sequenza sono generati in specie di Candida. (A) inclusa, plasmidi DNAes una sequenza FP e una codifica sequenza nourseothricin resistenza (NAT1). posizioni relative di Forward (FWD) e reverse primer (REV) sono mostrati, con porzioni nere dei primer che indica la regione di omologia alla sequenza plasmidi e le porzioni viola denotano la regione di omologia gene-specifica o estensione di primer. Cassette (B) FP si trasformano in Candida e si integrano all'interno del ENO1 locus genomico mediante ricombinazione omologa (linee tratteggiate). (C) risultante FP sequenza di fusione presso il 3'end di ENO1. Clicca qui per vedere una versione più grande di questa figura.
Qui, vi presentiamo un esempio di fusione di proteine (Eno1-FP) costruzioni in specie di Candida. Usiamo codifica plasmidi contenenti il gene marcatore NAT1 trasformazione insieme a sequenze codificanti GFP, YFP, omCherry (Figura 2). Questi plasmidi sono utilizzati insieme con primer in PCR per generare cassette gene-specifici che facilitano fusione PQ alla estremità 3 'del ENO1, con conseguente espressione di Eno1 fusa PQ al suo carbossi-terminale.
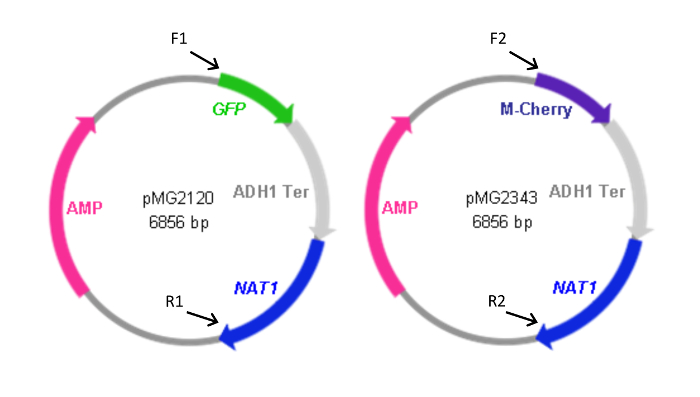
Figura 2: Mappe di FP plasmidi cassette contenenti. Forward (F) e reverse primer (R) utilizzati per generare le cassette dei plasmidi sono indicati nonché la posizione relativa della loro omologia con i plasmidi. Sequenze primer sono elencati nella tabella 1. F1 e R1 sono stati utilizzati anche per generare la cassetta pYFP- NAT1. Il plasmide contenente la cassetta YFP- NAT1 (pMG2263) è identica a pMG2120 con l'eccezione di YFP al posto della sequenza GFP. Dimensioni cassette: GFP-NAT1, 3.7 KBP; mCherry- NAT1, 3,2 kbp; YFP- NAT1, 3.7 kbp. Questo dato è stato modificato da Gerami-Nejad, et al. 4 Clicca qui per vedere una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Costruzione di epitopo etichettato sequenze in specie Candida utilizzando la strategia di modificazione genetica PCR-mediata sopra descritto possono essere riassunti come un processo in tre fasi. Innanzitutto, una cassetta è fatta da PCR che codifica sia la sequenza desiderata di integrazione e regioni omologa al locus di inserimento nel genoma di lievito. In secondo luogo, le cellule di lievito da trasformare sono chimicamente competenti con acetato di litio e co-incubate con la cassetta. Terzo, le cellule ve…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ringraziamo N. Dean per fornire la sequenza originale mCherry FP, M. Gerami-Nejad per la costruzione di plasmidi, B. Larson per l'assistenza tecnica, e T. Heisel per consigli utili durante lo sviluppo di questo progetto. JB è stato sostenuto dal premio Consiglio europeo della ricerca avanzata 340.087 (RAPLODAPT). sistemi di microscopia e di imaging sono stati forniti dalla University of Minnesota Pediatria Foundation e l'Università del Minnesota Imaging Center.
Materials
| 100W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5ml, 1.5ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C ) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C ) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4°C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
References
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. . Current Protocols in Molecular Biology. , (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).

