Laser-Capture Microdissection RNA-Sequencing voor ruimtelijke en temporale weefsel-specifieke genexpressie analyse in planten
Summary
Hier gepresenteerd is een protocol voor laser-capture microdissection (LCM) van plantaardige weefsels. LCM is een microscopische techniek voor het isoleren van gebieden van weefsel op een contaminatievrije manier. De procedure omvat weefselfixatie, paraffine inbedding, sectioning, LCM en RNA extractie. RNA wordt gebruikt in de downstream weefselspecifieke, tijdelijk opgeloste analyse van transcripto’s.
Abstract
De ontwikkeling van een complex meercellig organisme wordt beheerst door verschillende celtypen die verschillende transcriptieprofielen hebben. Om transcriptieregels te identificeren die ontwikkelingsprocessen regelen, is het noodzakelijk om de ruimtelijke en temporele genexpressieprofielen van deze individuele celtypen te meten. Daarom is inzicht in de spatio-temporele controle van genexpressie essentieel om inzicht te krijgen in hoe biologische en ontwikkelingsprocessen worden gereguleerd. Hier beschrijven we een laser-capture microdissection (LCM) methode om een klein aantal cellen te isoleren van drie gerst embryo organen tijdens een tijd-cursus tijdens de kiemkracht, gevolgd door transcript profilering. De methode bestaat uit weefselfixatie, weefselverwerking, paraffine inbedding, sectioning, LCM en RNA extractie gevolgd door real-time PCR of RNA-seq. Deze methode heeft ons in staat gesteld om ruimtelijke en temporele profielen van zaadorgaantranscripties te verkrijgen uit verschillende aantallen cellen (tientallen tot honderden), wat veel meer weefselspecificatie biedt dan typische bulkweefselanalyses. Uit deze gegevens konden we transcriptienetwerken definiëren en vergelijken en kandidaat-regelgevende transcriptiefactoren voor individuele weefsels voorspellen. De methode moet van toepassing zijn op andere plantaardige weefsels met minimale optimalisatie.
Introduction
Plantenontwikkeling en -groei omvatten de gecoördineerde actie van transcriptie-regelgevende netwerken binnen verschillende cellen die bestaan in een complexe cellulaire omgeving. Om de activiteit van deze regulerende netwerken te begrijpen, hebben we de kennis van ruimtelijke en temporele genexpressie binnen verschillende celtypen in ontwikkelingsstadia nodig. Echter, analyses van genexpressie worden vaker uitgevoerd in hele organen of bulk weefsel monsters als gevolg van de technische uitdaging van het isoleren en analyseren van kleine aantallen cellen. De methode die we hier beschrijven heeft het verkrijgen van ruimtelijke en temporele weefselspecifieke transcriptome analyse mogelijk gemaakt door LCM te koppelen aan RNA-seq.
LCM werd twee decennia geleden ontwikkeld door Emmert-Buck en collega’s1. De techniek stelde onderzoekers in staat om enkelcellen of clusters van cellen nauwkeurig te isoleren uit hun omgeving met behulp van directe microscopische visualisatie en manipulatie met een narrow beam laser1. Sindsdien is de methode op grote schaal gebruikt in kankerbiologie en pathologie2,3. Veel plantenonderzoeksgroepen hebben LCM ook aangepast voor het gebruik met verschillende plantensoorten en verschillende weefseltypen4,5,6,7,8,9,10,11. Onlangs hebben verschillende documenten ook LCM gebruikt op eudicot- en monocotzaden om embryo’s, endos en andere zaadstructuren te bestuderen tijdens zaadontwikkeling en kiemkracht10,12,13. De meeste andere veelgebruikte eencellige isolatiemethoden zoals micropipetting, celsortering, magnetische scheiding en microfluïde platforms zijn afhankelijk van de enzymatische spijsvertering of mechanische homogenisatie om cellen te scheiden. Dit kan verstoren genexpressie, de invoering van technische artefacten die gegevens interpretatieverstoren 14,15. Deze methoden vereisen ook eerdere kennis van marker genen voor elk celtype om de gescheiden cellen te relateren aan hun ruimtelijke locatie en echte cel-type. Een andere groep technieken hangt af van op affiniteit gebaseerde isolatie van subcellulaire structuren in plaats van hele cellen, bijvoorbeeld INTACT (Isolatie van nuclei’s gelabeld in celtypen) en TRAP (Vertalen ribosoom affiniteitszuivering)16,17. Affiniteit labelen en zuiveren van kernen of ribosomen zijn echter technisch uitdagend bij plantensoorten die geen gevestigde transformatieprotocollen hebben. LCM maakt gebruik van snelle weefselfixatie om transcriptieniveaus en conventionele histologische identificatie te behouden door directe visualisatie van cellen binnen hun normale weefsel/orgaancontext, waardoor afzonderlijke cellen in korte tijd18,19kunnen worden geïsoleerd.
Het hier gepresenteerde protocol is een geoptimaliseerde methode voor de isolatie van specifieke cellen of celtypen uit de weefselsecties van graanzaden, die kan worden toegepast op de meeste cellen die histologisch kunnen worden geïdentificeerd. LCM biedt een contactvrije methode van celisolatie, waardoor besmetting sterk wordt verminderd en de integriteit van teruggewonnen RNA wordt verhoogd. Bovendien illustreert de methode de kracht van LCM op grootschalige genoombrede studies te beginnen met kleine hoeveelheden biologische materialen. We beschrijven ook lineaire versterking van RNA voor het genereren van voldoende inputmateriaal voor downstream transcriptie/transcriptome analyses.
Er zijn tien belangrijke stappen in dit LCM RNA-seq protocol voor ruimtelijke en temporele weefselspecifieke transcriptomen, waaronder fixatie van weefselmonsters, uitdroging, paraffine-infiltratie, inbedding, sectie, LCM, RNA-extractie, RNA-versterking, RNA-kwantificering en qRT-PCR en/of RNA-seq(figuur 1).
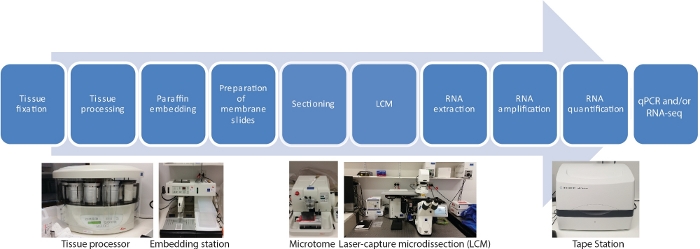
Figuur 1: Stroomdiagram van LCM gevolgd door RNA-seq of qRT-PCR. LCM is een ruimtelijk nauwkeurige en contactvrije techniek om cellen te verzamelen van vaste weefselsecties met behulp van een laserstraal onder microscopische visualisatie. Het proces begint met fixatie van weefselmonsters, gevolgd door uitdroging met behulp van een gradiënt reeks ethanol en xyleen, en afgewerkt met paraffine infiltratie. Het proces kan volledig geautomatiseerd worden met behulp van een tissue processor. Zodra het weefsel is geïnfiltreerd met paraffine, het is ingebed in een mal met gesmolten paraffine met behulp van een inbedding station. Sectioning wordt uitgevoerd met behulp van microtome ingesteld op de gewenste dikte. Dia’s worden voorbereid en LCM wordt vlak voor RNA uit gevangen cellen geëxtraheerd. RNA-extractie wordt direct gevolgd door twee rondes RNA-versterking voorafgaand aan qRT-PCR en/of RNA-seq. Klik hier om een grotere versie van dit cijfer te bekijken.
Protocol
Representative Results
Discussion
Veel weefselspecifieke genexpressiestudies zijn beperkt door handontleding van monsters, wat tijdrovend, arbeidsintensief is, een hoog risico op besmetting heeft en alleen monsters kan gebruiken die een menselijke agent voldoende behendig is om te oogsten. LCM is een nauwkeurige en contactloze techniek om cellen te verzamelen van vaste weefselsecties met behulp van een mechanisch bediende laserstraal onder microscopische visualisatie.
Een goede monstervoorbereiding is cruciaal voor LCM. Het pr…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dit werk werd ondersteund door het Australian Research Council Centre of Excellence in Plant Energy Biology (CE140100008) aan JW. M.G.L werd ondersteund door een startbeurs van de Universiteit van La Trobe. Wij danken het La Trobe Genomics Platform voor hun steun bij high-throughput sequencing en data-analyse. Wij danken universitair hoofddocent Matthew Tucker voor deskundig advies over de oprichting van LCM in ons lab.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
References
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

