Microsezione laser-cattura RNA-Sequenziamento per l'analisi dell'espressione genica spaziale e temporale specifica del tessuto nelle piante
Summary
Presentato qui è un protocollo per la microdissezione laser-cattura (LCM) di tessuti vegetali. LCM è una tecnica microscopica per isolare aree di tessuto in modo privo di contaminazione. La procedura include fissazione del tessuto, incorporamento di paraffina, sessazione, LCM e estrazione dell’RNA. L’RNA viene utilizzato nell’analisi dei trascrimi specifici del tessuto a valle e tempormente risolta.
Abstract
Lo sviluppo di un organismo multicellulare complesso è governato da tipi di cellule distinti che hanno diversi profili trascrizioni. Per identificare le reti di regolamentazione trascrizionali che governano i processi di sviluppo è necessario misurare i profili di espressione genica spaziale e temporale di questi singoli tipi di cellule. Pertanto, la comprensione del controllo spatio-temporale dell’espressione genica è essenziale per comprendere come sono regolati i processi biologici e di sviluppo. Qui, descriviamo un metodo di microdissezione laser-cattura (LCM) per isolare un piccolo numero di cellule da tre organi embrionali d’orzo in un corso di tempo durante la germinazione seguito dalla profilazione della trascrizione. Il metodo consiste nella fissazione dei tessuti, nell’elaborazione dei tessuti, nell’incorporazione di paraffina, nella sessazione, nell’estrazione di LCM e RNA seguita da PCR o RNA-seq in tempo reale. Questo metodo ci ha permesso di ottenere profili spaziali e temporali di trascrittori di organi di semi da un numero variabile di cellule (da decine a centinaia), fornendo una specificità del tessuto molto maggiore rispetto alle tipiche analisi dei tessuti sfusi. Da questi dati siamo stati in grado di definire e confrontare le reti normative trascrizioni, nonché prevedere i fattori di trascrizione normativa candidati per i singoli tessuti. Il metodo deve essere applicabile ad altri tessuti vegetali con ottimizzazione minima.
Introduction
Lo sviluppo e la crescita delle piante comportano l’azione coordinata delle reti di regolamentazione trascrizionali all’interno di diverse cellule esistenti in un ambiente cellulare complesso. Per comprendere l’attività di queste reti normative, abbiamo bisogno della conoscenza dell’espressione genica spaziale e temporale all’interno di diversi tipi di cellule attraverso le fasi di sviluppo. Tuttavia, le analisi dell’espressione genica sono più comunemente condotte in interi organi o campioni di tessuto sfusa a causa della sfida tecnica di isolare e analizzare un piccolo numero di cellule. Il metodo che descriviamo qui ha permesso di ottenere un’analisi del trascrittura specifica del tessuto spaziale e temporale accoppiando LCM con RNA-seq.
LCM è stato sviluppato due decenni fa da Emmert-Buck e colleghi1. La tecnica ha permesso ai ricercatori di isolare con precisione singole cellule o ammassi di cellule dal loro ambiente utilizzando la visualizzazione e la manipolazione microscopiche dirette con un laser a fasciostretto 1. Da allora il metodo è stato ampiamente utilizzato nella biologia del cancro e nellapatologia 2,3. Molti gruppi di ricerca vegetali hanno anche adattato LCM per l’uso con diverse specie vegetali e diversi tipi ditessuto 4,5,6,7,8,9,10,11. Recentemente, diversi documenti hanno anche usato LCM su semi di eudicot e monocot per studiare embrioni, endospermemi e altre strutture di semi durante lo sviluppo dei semi e lagerminazione 10,12,13. La maggior parte degli altri metodi di isolamento a cella singola comunemente utilizzati come micro-pipettazione, smistamento cellulare, separazione magnetica e piattaforme microfluidiche dipendono dalla digestione ezimatica o dall’omogeneizzazione meccanica per dissociare le cellule. Questo può perturbare l’espressione genica, introducendo artefatti tecnici che confondono l’interpretazionedei dati 14,15. Questi metodi richiedono anche una conoscenza precedente dei geni marcatori per ogni tipo di cellula per correlare le cellule dissociate alla loro posizione spaziale e al vero tipo di cellula. Un ulteriore gruppo di tecniche dipende dall’isolamento basato sull’affinità delle strutture subcellulari anziché delle celle intere, ad esempio INTACT (Isolation of Nuclei Tagged in Cell Types) e TRAP (Translating Ribosome Affinity Purification)16,17. Tuttavia, l’etichettatura di affinità e la purificazione di nuclei o ribosomi sono tecnicamente impegnative nelle specie vegetali che non hanno protocolli di trasformazione ben consolidati. LCM sfrutta la fissazione rapida dei tessuti per preservare i livelli di trascrizione e l’identificazione ircologica convenzionale mediante la visualizzazione diretta delle cellule all’interno del loro normale contesto di tessuto/organo, che consente alle cellule discrete di essere isolate in un breveperiodo di tempo 18,19.
Il protocollo qui presentato è un metodo ottimizzato per l’isolamento di cellule o tipi di cellule specifiche dalle sezioni tisssate dei semi di cereali, che possono essere applicate alla maggior parte delle cellule che possono essere identificate istologicamente. LCM fornisce un metodo senza contatto di isolamento cellulare, riducendo notevolmente la contaminazione e aumentando l’integrità dell’RNA recuperato. Inoltre, il metodo illustra la potenza della ML su larga scala su studi su tutto il genoma a partire da piccole quantità di materiali biologici. Descriviamo anche l’amplificazione lineare dell’RNA per generare materiale di input sufficiente per le analisi di trascrizione/trascrizione a valle.
Ci sono dieci passaggi principali in questo protocollo LCM RNA-seq per trascrittori spaziali e temporali specifici del tessuto, tra cui la fissazione di campioni di tessuto, disidratazione, infiltrazione di paraffina, incorporamento, sezionazione, LCM, estrazione dell’RNA, amplificazione dell’RNA, quantificazione dell’RNA e qRT-PCR e/o RNA-seq (Figura 1).
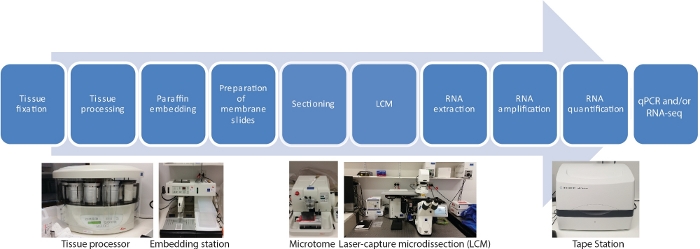
Figura 1: diagramma di flusso di LCM seguito da RNA-seq o qRT-PCR. LCM è una tecnica spazialmente precisa e senza contatto per raccogliere cellule da sezioni di tessuto fisse utilizzando un raggio laser sotto visualizzazione microscopica. Il processo inizia con la fissazione di campioni di tessuto, seguita da disidratazione utilizzando una serie di gradienti di etanolo e xylene, e finito con infiltrazione di paraffina. Il processo può essere completamente automatizzato utilizzando un processore di tessuto. Una volta che il tessuto è infiltrato con paraffina, è incorporato in uno stampo con paraffina fusa utilizzando una stazione di incorporamento. Il sessaggio viene eseguito utilizzando microtomo impostato allo spessore desiderato. I vetrini vengono preparati e LCM condotto immediatamente prima che l’RNA venga estratto dalle cellule catturate. L’estrazione dell’RNA è seguita direttamente da due cicli di amplificazione dell’RNA prima di qRT-PCR e/o RNA-seq. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Molti studi sull’espressione genica specifici dei tessuti sono stati limitati dalla dissezione manuale di campioni, che richiede molto tempo e richiede lavoro, ha un alto rischio di contaminazione e può utilizzare solo campioni che un operatore umano è sufficientemente abile da raccogliere. LCM è una tecnica precisa e senza contatto per raccogliere cellule da sezioni di tessuto fisse utilizzando un raggio laser azionato meccanicamente sotto visualizzazione microscopica.
Una buona preparazio…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questo lavoro è stato sostenuto dall’Australian Research Council Centre of Excellence in Plant Energy Biology (CE140100008) a JW. M.G.L è stato sostenuto da una sovvenzione di partenza dell’Università di La Trobe. Ringraziamo la piattaforma Genomica La Trobe per il loro supporto nel sequenziamento ad alta velocità effettiva e nell’analisi dei dati. Ringraziamo il Professore Associato Matthew Tucker per la consulenza di esperti su come stabilire LCM nel nostro laboratorio.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
References
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

