Functionele beoordeling van BRCA1-varianten met CRISPR-gemedieerde basiseditors
Summary
Mensen met BRCA1-mutaties hebben een hoger risico op het ontwikkelen van kanker, wat een nauwkeurige evaluatie van de functie van BRCA1-varianten garandeert. Hierin beschreven we een protocol voor functionele beoordeling van BRCA1-varianten met behulp van CRISPR-gemedieerde cytosinebasiseditors die gerichte C:G naar T:A-conversie in levende cellen mogelijk maken.
Abstract
Recente studies hebben de risico’s onderzocht die gepaard gaan met BRCA1-genmutaties met behulp van verschillende functionele beoordelingsmethoden zoals fluorescerende reportertests, embryonale stamcel levensvatbaarheidstests en therapeutische op geneesmiddelen gebaseerde gevoeligheidstests. Hoewel ze veel BRCA1-varianten hebben verduidelijkt, worden deze tests met betrekking tot het gebruik van exogeen uitgedrukte BRCA1-varianten geassocieerd met overexpressieproblemen en kunnen ze niet worden toegepast op posttranscriptionele regelgeving. Om deze beperkingen op te lossen, rapporteerden we eerder een methode voor functionele analyse van BRCA1-varianten via CRISPR-gemedieerde cytosinebasiseditor die gerichte nucleotidesubstitutie in levende cellen induceren. Met behulp van deze methode identificeerden we varianten waarvan de functies dubbelzinnig blijven, inclusief c.-97C>T, c.154C>T, c.3847C>T, c.5056C>T en c.4986+5G>A, en bevestigde dat CRISPR-gemedieerde basiseditors nuttige hulpmiddelen zijn voor het herindelen van de varianten van onzekere betekenis in BRCA1. Hier beschrijven we een protocol voor functionele analyse van BRCA1-varianten met behulp van CRISPR-gebaseerde cytosinebasiseditor. Dit protocol biedt richtlijnen voor de selectie van doellocaties, functionele analyse en evaluatie van BRCA1-varianten.
Introduction
Het borstkanker type 1 gevoeligheidsgen (BRCA1) is een algemeen bekend tumoronderdrukkergen. Omdat het BRCA1-gen gerelateerd is aan het herstel van DNA-schade, zouden mutaties in dit gen leiden tot een groter risico op kankerontwikkeling bij een individu1. Borst-, eierstok-, prostaat- en pancreaskankers zijn gekoppeld aan erfelijke functieverliesmutaties (LOF) van het BRCA1-gen 2. Functionele beoordeling en identificatie van BRCA1-varianten kunnen helpen bij het voorkomen en diagnosticeren van de verschillende ziekten. Om de functie van BRCA1-varianten aan te pakken, zijn verschillende methoden ontwikkeld en in grote lijnen gebruikt voor het onderzoeken van de pathogeniteit van BRCA1-varianten zoals embryonale stamcel levensvatbaarheidstests, fluorescerende reportertests en therapeutische op geneesmiddelen gebaseerde gevoeligheidstests3,4,5,6. Hoewel deze methoden de functie van veel BRCA1-varianten hebben beoordeeld, stellen de methoden met exogeen uitgedrukte BRCA1-varianten beperkingen in termen van overexpressie die van invloed kunnen zijn op downstreamregulatie, gendosering en eiwitvouwen7. Bovendien kunnen deze tests niet worden gebruikt voor de posttranscriptionele regelgeving, zoals mRNA-splicing, transcriptstabiliteit en effect van onvertaald gebied8,9.
CRISPR-Cas9-systeem maakt gerichte genoombewerking in levende cellen en organismenmogelijk 10. Via een enkelgeleid RNA kan Cas9 dubbelstrengsbreuken (DSB’s) in chromosomale DNA induceren bij specifieke genomische loci om twee DNA-reparatiepaden te activeren: foutgevoelige niet-geologische end-joining (NHEJ) pathway en error-free homology-directed repair (HDR) pathway11. HDR is een nauwkeurig reparatiemechanisme; DSBs geïnduceerd door Cas9 nuclease voor HDR resulteert echter vaak in ongewenste invoeging en verwijdering (indel) mutatie. Bovendien heeft het homologe donor-DNA-sjablonen nodig voor het herstellen van DNA-schade en heeft het een relatief lage efficiëntie. Onlangs, Cas9 nickase (nCas9) zijn gesmolten met cytidine deaminase domeinen voor het richten van C:G naar T:A substituties, zonder de noodzaak van homologe DNA sjablonen en DNA dubbele streng breekt12,13,14,15. Met behulp van de cytosine base editor ontwikkelden we een nieuwe methode voor functionele analyse van BRCA1 varianten16.
In deze studie gebruikten we CRISPR-gemedieerde cytosinebasiseditor, BE314, die efficiënte C:G- tot T:A-puntmutaties induceert, voor de implementatie van de functionele beoordeling van BRCA1-varianten en identificeerden we met succes de functies van verschillende BRCA1-varianten (figuur 1).
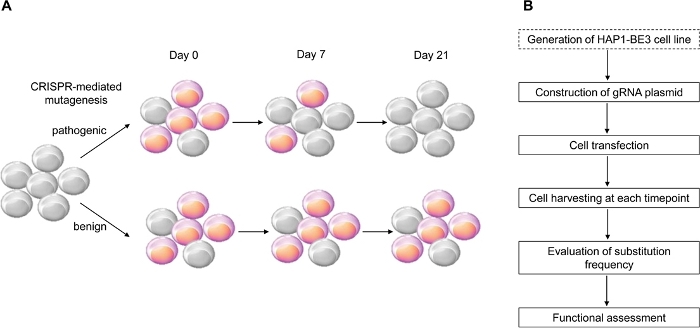
Figuur 1: Een overzicht van de workflow voor functionele beoordeling. (A) Schematisch met de functionele beoordeling van BRCA1. Omdat het LOF van BRCA1 de levensvatbaarheid van cellen beïnvloedt, wanneer de BRCA1-mutatie pathogeen is, sterven de cellen naarmate het passagenummer toeneemt. (B) Fasen van de functionele beoordeling van BRCA1. Gestippelde doos is optioneel. Het kan worden vervangen door co-transfectie van gRNA uitdrukken en BE3 uitdrukken plasmiden DNA. Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
Dit protocol beschrijft een eenvoudige methode voor functionele beoordelingen van BRCA1-varianten met behulp van CRISPR-gemediteerde cytosinebasiseditor. Het protocol beschrijft methoden voor het ontwerp van gRNA’s bij doel locus en de constructie van de plasmide DNA’s waaruit ze worden uitgedrukt. Cytosinebasiseditors induceren nucleotideconversie in een actief venster (in het geval van BE3, nucleotiden 4–8 in het PAM-distale uiteinde van de gRNA-doelsequenties). De onderzoeker moet zorgvuldig doelsequenties …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dit werk werd ondersteund door de National Research Foundation of Korea (subsidies 2017M3A9B4062419, 2019R1F1A1057637 en 2018R1A5A2020732 to Y.K.).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
References
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. 암 연구학. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

