CRISPR媒介ベース・エディタを用いた BRCA1 バリアントの機能評価
Summary
BRCA1突然変異を有する人々は、BRCA1変異体の機能の正確な評価を保証する癌を発症するリスクが高い。本明細書において、生細胞における標的C:GからT:A変換を可能にするCRISPR媒介シトシンベースエディタを用いたBRCA1変異体の機能評価用プロトコルについて説明した。
Abstract
最近の研究では、蛍光レポーターアッセイ、胚性幹細胞生存アッセイ、治療用薬物ベースの感受性アッセイなどの様々な機能評価方法を用いて 、BRCA1 遺伝子変異に関連するリスクを調査しています。彼らは多くの BRCA1 変異体を明らかにしているが、外因的に発現した BRCA1 変異体の使用を含むこれらのアッセイは過剰発現の問題に関連しており、転写後の調節には適用できない。これらの制限を解決するために、我々は以前、生細胞における標的ヌクレオチド置換を誘導するCRISPR媒介シトシン塩基エディタを介して BRCA1 変異体の機能分析方法を報告した。この方法を使用して、c.-97C>T、c.154C>T、c.3847C>T、c.5056C>T、c.4986+5G>Aなど、関数があいまいなままのバリアントを特定し、CRISPRメディア化された基本エディタが BR1の不確定な変異体の再分類に役立つことを確認しました。ここでは、CRISPRベースのシトシンベースエディタを用いた BRCA1 変異体の機能解析用プロトコルについて説明する。このプロトコルは、ターゲット・サイトの選択、機能分析、 および BRCA1 バリアントの評価に関するガイドラインを提供します。
Introduction
乳癌タイプ1感受性遺伝子(BRCA1)は、広く知られている腫瘍抑制遺伝子である。BRCA1遺伝子はDNA損傷の修復に関連しているため、この遺伝子の変異は、個体1における癌発症のリスクが高くなる。乳癌、卵巣癌、前立腺癌、および膵臓癌はBRCA1遺伝子2の遺伝性機能喪失(LOF)突然変異に関連している。BRCA1変異体の機能評価および同定は、様々な疾患の予防および診断に役立つ可能性がある。BRCA1変異体の機能に対処するために、胚性幹細胞生存アッセイ、蛍光レポーターアッセイ、および治療用薬物ベースの感受性アッセイ3、4、5、6などのBRCA1変異体の病原性を調査するために、いくつかの方法が開発され、広く使用されている。これらの方法は多くのBRCA1変異体の機能を評価しているが、外因的に発現したBRCA1変異体を含む方法は、下流の調節、遺伝子投与量、およびタンパク質折りたたみ7に影響を及ぼす可能性のある過剰発現の点で制限をもたらす。さらに、これらのアッセイは、mRNAスプライシング、転写安定性、および非翻訳領域8,9の効果などの転写後の調節に利用することができない。
CRISPR-Cas9システムは、生きている細胞および生物10における標的ゲノム編集を可能にする。単一ガイドRNAを介して、Cas9は、エラーが起こりやすい非相同性末端結合(NHEJ)経路およびエラーを起こしやすい相同性指向修復(HDR)経路11の2つのDNA修復経路を活性化するために、特定のゲノム遺伝子座で染色体DNA中の二本鎖切断(DSB)を誘導することができる。HDR は、正確な修復メカニズムです。しかし、HDRのCas9ヌクレアーゼによって誘導されたDSBは、しばしば望ましくない挿入および欠失(indel)突然変異をもたらす。さらに、DNA損傷を修復するための相同なドナーDNAテンプレートが必要であり、効率が比較的低い。最近、Cas9ニッカーゼ(nCas9)は、C:GからT:Aの置換を標的とするシチジンデアミネーゼドメインと融合しており、相同なDNAテンプレートおよびDNA二重鎖破断12、13、14、15を必要としない。シトシンベースエディタを用いて、BRCA1バリアント16の機能解析のための新しい方法を開発しました。
本研究では、BRCA1変異体の機能評価を実施するために、効率的なC:GからT:A点突然変異を誘導するCRISPR媒介シトシンベースエディタBE314を用いて、いくつかのBRCA1変異体の機能を同定することに成功した(図1)。
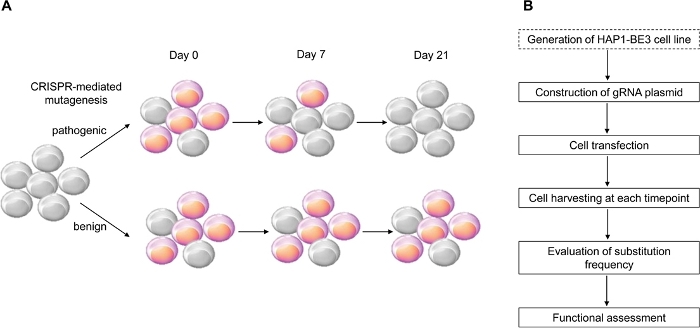
図 1: 機能評価のワークフローの概要(A) BRCA1の機能評価を示す回路図。 BRCA1 のLOFは細胞の生存率に影響を与えるので 、BRCA1 突然変異が病原性である場合、細胞は通過数が増加するにつれて死ぬ。(B) BRCA1の機能評価の段階 。ドット ボックスはオプションです。それは、プラスミドDNAを発現するgRNAとBE3の共トランスフェクションに置き換えることができる。 この図の大きなバージョンを表示するには、ここをクリックしてください。
Protocol
Representative Results
Discussion
このプロトコルは、CRISPR-瞑想されたシトシンベースエディタを用いた BRCA1 変異体の機能評価のための簡単な方法を説明する。プロトコルは、標的遺伝子座におけるgRNAの設計とそれらが発現するプラスミド・ドナの構築のための方法を記述する。シトシン塩基エディタは、活性ウィンドウでヌクレオチド変換を誘導する(BE3の場合、gRNA標的配列のPAM-遠位末端のヌクレオチド4〜8)。アク?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
この研究は、韓国国立研究財団(助成金2017M3A9B4062419、2019R1F1F1F1057637、2018R1A5A2020732 Y.K.に支援されました)。
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
References
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. 암 연구학. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

