Büyüme ve Gen İfadesinde Heterojenliği Ölçmek için Mikrokolonların Yüksek Verimli Canlı Görüntülemesi
Summary
Maya büyüme fenotipleri, mikrokolonlara dönüşen hareketsiz hücrelerin son derece paralel zaman atlamalı görüntülemesi ile tam olarak ölçülür. Aynı zamanda, stres toleransı, protein ekspresyasyonu ve protein lokalizasyonu izlenebilir, çevresel ve genetik farklılıkların yanı sıra izojenik hücreler arasındaki gen ekspresyon heterojenliğinin büyümeyi nasıl modüle ettiğini incelemek için entegre veri kümeleri üretilebilir.
Abstract
Mikrobiyal büyüme oranlarındaki suş içi heterojenitenin hassas ölçümleri, stres toleransı, patojenite ve zindeliğin diğer temel bileşenlerine genetik ve çevresel girdileri anlamak için gereklidir. Bu makale, deney başına yaklaşık 105 Saccharomyces cerevisiae mikrokolonyasını izleyen mikroskop tabanlı bir tahlil tanımlamaktadır. Mayanın çok katlı bir plakada hareketsiz olarak otomatik olarak zaman atlamalı görüntülenmesi sonrasında, mikrokolony büyüme oranları özel görüntü analiz yazılımı ile kolayca analiz edilir. Her mikrokolony için floresan proteinlerin ekspresyonu ve lokalizasyonu ve akut stresin sağkalımları da izlenebilir. Bu tahlil, suşların ortalama büyüme oranlarının kesin olarak tahmin edilmesine ve klonal popülasyonlarda büyüme, gen ekspresyasyonu ve stres toleransında heterojenliğin kapsamlı bir şekilde ölçülmesine izin verir.
Introduction
Büyüme fenotipleri maya zindeliği için kritik katkıda bulunur. Doğal seleksiyon, 108 kişiyi aşabilen etkili nüfus büyüklüğünün tersi ile farklılık gösteren büyüme oranları ile soyları verimli bir şekilde ayırt edebilir1. Ayrıca, bir popülasyondaki bireyler arasında büyüme oranlarının değişkenliği evrimsel olarak ilgili bir parametredir, çünkü bahis koruma 2 , 3,4,5,6gibi hayatta kalma stratejilerinin temelini oluşturabilir. Bu nedenle, büyüme fenotiplerinin ve dağılımlarının son derece doğru ölçümlerine izin veren tahliller mikroorganizmaların incelenmesi için çok önemlidir. Burada açıklanan mikrokolony büyüme tahlili, deney başına ~105 mikrokolon için bireysel büyüme hızı ölçümleri oluşturabilir. Bu nedenle bu test, maya evrimsel genetiği ve genomikleri incelemek için güçlü bir protokol sağlar. Genetik olarak özdeş tek hücre popülasyonları içindeki değişkenliğin nasıl üretildiğini, sürdürüldüğünü ve popülasyon kondisyonu 7 ,8,9,10’anasıl katkıda bulunduğunu test etmek için özellikle iyi bir şekilde kendini ödünç verir.
Burada açıklanan yöntem (Şekil 1), mikrokolonlara büyümeyi izlemek için 96 veya 384 kuyulu bir cam tabanlı plaka üzerinde sıvı ortamda yetişen hücrelerin periyodik olarak yakalanan, düşük büyütmeli parlak alan görüntülerini kullanır. Hücreler, mikroskop plakasının altını kaplayan ve iki boyutlu koloniler oluşturan A lektin kontanvalinine yapışır. Mikrokolonlar monolayerde büyüdüğünden, mikrokolony alanı7numaralı hücre ile oldukça ilişkilidir. Bu nedenle, her bir mikrokolony alanının değişim hızını izleyen özel görüntü analizi yazılımı ile mikrokolony büyüme hızı ve gecikme süresi hakkında doğru tahminler oluşturulabilir. Ayrıca, deneysel kurulum, bu mikrokolonlarda ifade edilen floresan etiketli proteinlerin bolluklarını ve hatta hücre altı lokalizasyonlarını izleyebilir. Bu mikrokolonyum büyüme testinden elde edilen verilerin aşağı akış işlemesi, özel analiz veya Görüntüleri Kolayca İşleme (PIE)11, sağlam koloni alanı tanıma ve gitHub12aracılığıyla kullanılabilen düşük büyütmeli, parlak alan görüntülerinden yüksek verimli büyüme analizi için bir algoritma gibi mevcut görüntü analizi yazılımı ile elde edilebilir.
Mikrokolony-büyüme testinden elde edilen büyüme hızı tahminleri çok sayıda tek koloni ölçümünden oluşturulduğundan, makul büyüklükte bir deney için tahminlerden birkaç büyüklük sırasıyla standart hatalarla son derece doğrudur. Bu nedenle, tahlillerin farklı genotipler, tedaviler veya çevresel koşullar arasındaki büyüme hızı farklarını tespit etme gücü yüksektir. Multiwell-plate formatı, çok sayıda farklı ortam ve genotip kombinasyonunun tek bir deneyde karşılaştırılmasını sağlar. Suşlar farklı floresan belirteçleri tam olarak ifade ederse, aynı kuyuda karıştırılabilir ve sonraki görüntü analizi ile ayırt edilebilir, bu da iyi veri normalleştirmesine izin vererek gücü daha da artırabilir.
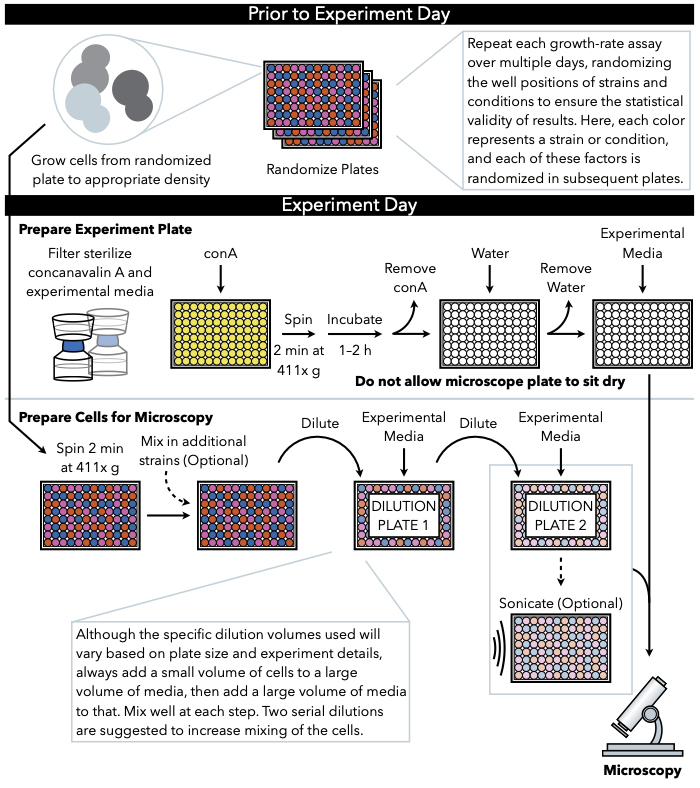
Şekil 1: Protokolün şematik gösterimi. Bu protokol, deneysel plakanın hazırlanması ve hücrelerin görüntüye hazırlanması olan iki ana adımı izler. Plakaların randomizasyonu ve hücrelerin büyümesi deney gününden önce ve bu güne kadar yapılmalıdır. Seyreltme sırasında her adımda hücrelerin tekrar tekrar karıştırılması, kaplamaya kadar olan adımlarda zorunludur ve bu nedenle deneysel plakanın ilk olarak hazırlanması önerilir, böylece hücre seyreltmenin tamamlanmasından hemen sonra kaplamaya hazırdır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
Burada açıklanan protokol, hücre büyümesi ve gen ekspresyonunun aynı anda bireysel mikrokolonlar düzeyinde izlenmesini sağlayan çok yönlü bir testtir. Bu iki modaliteyi birleştirmek benzersiz biyolojik içgörüler sağlar. Örneğin, önceki çalışmalar bu tahlili, aynı anda7,10’unher ikisini de ölçerek Izojenik wildtype hücrelerinde TSL1 geninin ekspresyonu ile mikrokolony büyüme hızı arasında negatif bir korelasyon göstermek i?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Naomi Ziv, Sasha Levy ve Shuang Li’ye bu protokolün geliştirilmesine katkılarından dolayı, David Gresham’a paylaşılan ekipmanlar için ve Marissa Knoll’a video prodüksiyon konusunda yardımları için teşekkür ederiz. Bu çalışma Ulusal Sağlık Enstitüleri hibesi R35GM118170 tarafından desteklendi.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
References
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. 유전학. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. 유전학. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. 유전학. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

