오픈 소스 smfBox를 사용하여 정확하고 정확한 단일 분자 FRET 측정
Summary
이 문서에서는 스위치 켜기부터 정렬 및 초점, 데이터 수집 및 분석에 이르기까지 오픈 소스, 저렴한 smfBox를 사용하여 생체 분자를 자유롭게 확산시키는 개별에 대해 완전히 수정된 정확한 FRET 측정을 만들기 위한 단계별 지침을 제공합니다.
Abstract
smfBox는 단일 분자 Förster 공명 에너지 전송 (smFRET)을위한 최근 개발 된 비용 효율적인 오픈 소스 기기로, 자유롭게 확산 생체 분자에 대한 측정을 보다 쉽게 사용할 수 있습니다. 이 개요에는 샘플 준비, 계측기 설정 및 정렬, 데이터 수집 및 완전한 분석 루틴의 세부 사항을 포함하여 이중 DNA 샘플에서 정확한 FRET 효율성을 측정하기 위해 이 계측기를 사용하기 위한 단계별 프로토콜이 포함되어 있습니다. 정확한 FRET 파생 거리 측정에 필요한 모든 보정 요소를 결정하는 방법을 포함하는 제시된 접근 방식은 표준 프로토콜 및 분석 접근 방식을 설정하는 것을 목표로 하는 FRET 커뮤니티 전반에 걸쳐 최근 협력 작업의 큰 본체를 기반으로 합니다. 다양한 생체 분자 시스템에 쉽게 적응할 수 있는 이 프로토콜은 광범위한 과학 커뮤니티를 위한 smFRET 민주화에 대한 노력이 증가하고 있습니다.
Introduction
단일 분자 Förster 공명 에너지 전송 (smFRET)는 개별 분자의 수준에서 두 염료 -a 기증자와 수용자 사이의 FRET 효율을 측정하는 기술이다. FRET는 두 염료의 중복 에너지 스펙트럼에서 발생하는 광물리 학적 과정입니다 : 기증자는 특정 파장의 빛에 흥분하고 수용자에서 방출의 결과로, 수용자로 비 방사 적으로 에너지를 전송합니다. 이 전송의 효율은 두 염료 사이의 거리의 여섯 번째 힘에 반비례하므로 전송 효율은 distance1에 따라 다릅니다. 따라서, 이러한 FRET 효율은 염료가 부착되는 분자에 대한 공간 정보를 결정하는 데 사용될 수 있다. 이러한 규모와 FRET 효율의 변화가 Angstrom 분자 운동에 민감하다는 사실은, 핵산 및 단백질과 같은 생체 분자에 대한 구조적 정보를 평균 4,5,6의 합병증없이 조사하는 데 적합한 기술을 만든다. 상대적인 FRET 효율성의 변화는 생체 분자 상호 작용 및 형성 역학을 모니터링하는 데 사용될 수 있지만, 단백질(un)접기, 전사 및 DNA 복제 및 수리와 같은 주요 세포 공정에 빛을 흘리는 반면, 절대 FRET 효율은 생체 분자 구조 결정에 대한 정확한 거리를 결정하는 데 사용되어 왔습니다7,8,9,10,111 , 다른 구조적 방법에 필요한 결정화 또는 동결의 필요성을 극복4,12.
smFRET 실험은 가장 일반적으로 두 가지 형태를 취, 공초점 또는 총 내부 반사 형광 (TIRF) 현미경 검사. 두 접근 사이 생체 분자의 분자 역학은 전형적으로 pico-to millisecond (공초점, 자유롭게 확산 분자)에서 시간까지 시간 척도에서 조사 할 수 있습니다 (TIRF, 표면 고정 분자). 이는 각 기술에 관련된 다양한 설정 때문입니다. TIRF 현미경 검사법에서 분자는 슬라이드 표면에 고정되어 에반센드 파에 흥분됩니다(그림 1A). 여기서, 그러나, 초점은 이 smfBox의 형식이기 때문에 공초점 현미경 검사법에 있습니다. 공초점 현미경 검사에서 분자는 고숫자 조리개 렌즈를 통해 레이저 빔을 솔루션 내의 일부 지정된 깊이에서 스팟으로 집중하여 형성된 공초점 부피(~1fL)를 통해 브라우니아 운동을 통해 자유롭게 확산된다(그림 1B). 생성된 방출은 동일한 조리개를 통해 다시 초점을 맞추고 이색 거울을 통해 필터링됩니다(전체 회로도의 그림 1C ). 그런 다음 초점이 맞지 않는 빛을 제거하고 눈사태 포토다이오드(APD)에 초점을 맞춥니다. APD가 광자를 감지하면 TTL 펄스를 출력하며, 타이밍은 최대 피코초 해상도로 기록될 수 있습니다. 공초점 부피 부근내에서 자유롭게 확산되는 분자의 관찰 시간은 일반적으로 밀리초 의 순서 내에 있습니다.
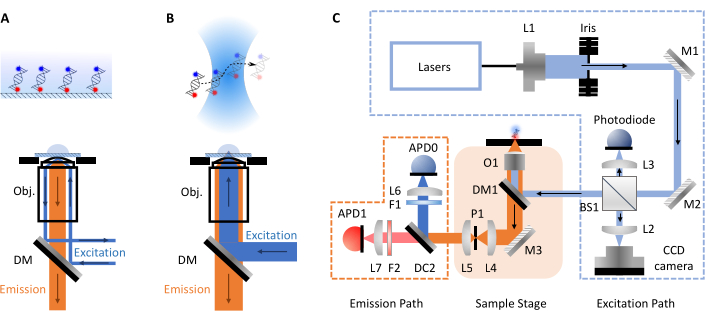
그림 1: 현미경 검사법과 smfBox 설정의 원리를 보여주는 회로도. (A) 총 내부 반사 형광 (TIRF) 현미경 원리 : 흥분 빛은 목표 (Obj.)의 가장자리로 지시하고 기하급수적으로 부패 하는 에반스 엔스 필드를 생성하는 커버 슬립 버퍼 인터페이스에서 총 내부 반사를 겪는다. (B) 공초점 현미경 검사: 분자를 자유롭게 확산시키는 것은 시료에 초점을 맞춘 거의 회절 제한 지점에 의해 흥분됩니다. (C) 눈사태 포토다이오드(APD), 빔 스플리터(BS), 이색 거울(DM), 필터(F), 미러(M), 목표(O) 및 핀홀(P)의 모든 주요 구성 요소를 보여주는 이 프로토콜에 사용되는 smfBox 설정. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
최근에는 smFRET 기술이 두 가지 색 여기를 통합하여 기증자와 수용자 내분 파장을 일치시키는 레이저가 번갈아 5입니다. 이것은 두 가지 방법 중 하나에서 수행 할 수 있습니다, KHz 기간에 연속 파 레이저를 변조하여 첫 번째, 이는 교대 레이저 여기로 알려진 (ALEX)13,14. 두 번째 방법은 MHz 시간 척도에서 빠른 펄스를 인터링합니다. 이는 나노초-ALEX15 또는 펄스 인터리브 된 여기 (PIE)16입니다. 이러한 모든 접근법에서, 수용자 레이저의 정보는 소위 stoichiometry의 계산으로 이끌어 냅니다, 이는 낮은 FRET 효율을 가진 분자와 수용자가 부족한 사람들 (불완전한 라벨링 또는 광표백을 통해)를 구별할 수 있는. PIE/ns-ALEX를 사용하면 단일 분자 수준에서 형광 수명에 추가로 접근할 수 있으며, 편광 광학과 결합될 때 이산화정도를 측정할 수 있습니다. 이러한 측정 조합은 다중 파라미터 형광 검출(MFD)9라고 합니다.
smFRET의 많은 장점에도 불구 하 고, 그것은 널리 상업 악기의 높은 비용과 간단한의 부족으로 전문 실험실 외부에서 사용 되지 않습니다., 자체 빌드 대안. 저가형 오픈소스 현미경 검사법의 개발을 향한 추세가 증가하고 있으며 플랑크톤코프17, 오픈플렉스어마이크로스코프18, 플렉시코프19, miCube20, liteTIRF21 및 Squid22를 포함한 다른 플랫폼이 최근 등장했습니다. 본 연구에서는 단일 분자를 자유롭게 확산시키는 데 대한 두 염료 사이의 FRET 효율을 측정할 수 있는 최근 개발된 비용 효율적인 공초점 설정인 smfBox를 사용하기 위한 프로토콜을 설명합니다. 자세한 빌드 지침과 필요한 모든 운영 소프트웨어는 https://craggslab.github.io/smfBox/ 23에서 자유롭게 사용할 수 있습니다. smfBox의 광학 배열은 저렴하고 널리 접근 가능한 제조업체에서 구입한 쉽게 사용할 수 있는 구성 요소에서 조립되며, 현미경 본체(표준 공초점 설정에서 비용의 대부분을 담당함)는 사용자 정의 조명 이밀한 양극산화 알루미늄 상자로 대체되었습니다(주변 조명 조건에서 측정할 수 있음). 이 상자에는 여기 이색, 목표 및 핀홀, 기계식 레이저 인터록을 포함한 주요 광학 부품이 포함되어 있어 Class I 레이저 제품으로 안전한 작동을 가능하게 합니다(전체 회로도의 그림 1C 참조). smfBox는 ALEX를 사용하여 염료 스토이치오메트리의 유효성을 검사하고 정확한 FRET 보정 요인을 결정합니다. 데이터 수집의 모든 측면을 제어하고 많은 타사 분석 도구와 호환되는 오픈 소스 광자-HDF5 format24의 데이터를 출력하는 맞춤형 오픈 소스 소프트웨어(smOTTER)를 사용하여 작동됩니다. smfBox 및 수집 및 데이터 분석 프로토콜은 최근 멀티 랩 블라인드 연구에서 >20 개의 다른 기기 (공초점 및 TIRF 모두)에 대해 테스트되었습니다25. 얻은 FRET 효율성은 smfBox가 시판되는 설정 가격의 일부만 비용에도 불구하고 다른 모든 악기와 우수한 계약을 맺었습니다.
여기서 단계별 프로토콜은 smfBox를 사용하여 DNA 듀플렉스를 자유롭게 확산시키는 데 있어 정확하고 절대적인 FRET 효율성을 획득하고 분석하기 위해 설명되어 있으며, 전원켜기부터 정렬 및 초점, 데이터 수집 및 분석에 이르기까지 모든 방법을 제공합니다. 여기서 사용되는 샘플은 세계 적인 맹목적인 연구에서 평가된 3개의 이중 DN(고, 중간 및 저FRET 효율성을 전시하고, 표 1참조)입니다; 그러나, 이 방법은 단백질 및 기타 핵산을 포함한 많은 분자 시스템에 적응할 수 있다. 희망은 이러한 상세한 프로토콜, smfBox23에 대한 기존의 기존 빌드 지침과 함께, 실험실의 넓은 범위에 더 액세스 할 수있는이 강력한 기술을 만드는 데 도움이 될 것입니다.
Protocol
Representative Results
Discussion
프로토콜에서 가장 중요한 단계는 현미경의 정렬및 정확한 희석에 샘플 농도를 조정하는 것입니다. 정렬이 꺼진 경우 버스트를 식별하고 히스토그램을 플롯하는 신호가 충분하지 않을 수 있으며 샘플 간에 정렬이 잘못 발생하면 누설 및 감지/흥분 효율성의 변화로 인해 정확한 FRET 보정이 실패할 수 있습니다. 적절한 농도의 사용은 또한 중요 하다, 너무 높은 농도 일치 된 버스트를 줄 것 이다, 잠재적으로 다른 FRET 효율성 또는 라벨 stoichiometries와 여러 분자를 포함. 농도가 너무 낮을 때 강력한 데이터 분석을 위해 너무 적은 버스트를 줄 수 있습니다.
여기에 설명된 프로토콜은 정적 단일 FRET 종에서 거리를 측정하기 위한 것입니다. 샘플이 FRET 효율 히스토그램에 두 개 이상의 피크를 가지고 있거나 피크가 광범위하게 나타나면 (동적 종으로 발생할 수 있음), 히스토그램을 동일한 정도의 정밀도에 맞추기 위해 더 많은 버스트가 필요할 수 있습니다. 잘 분리된 두 개의 피크의 경우 약 두 배의 데이터가 필요하지만 인구가 약간 겹치면 더 많은 데이터가 필요합니다.
두 집단이 실험의 시간 척도에서 상호 변환하는 경우 시스템의 역학 및 운동을 잠재적으로 결정할 수 있습니다. BVA27 및 2CDE28과 같은 테스트는 중간 버스트가 본질적으로 역동적임을 확인할 수 있는 반면 dPDA29,30 또는 H2MM31을 포함한 분석은 상호 변환 속도를 결정할 수 있습니다. BVA 및 2CDE용 Jupyter 노트북은 FRETBursts26 웹 사이트에서 사용할 수 있으며 MATLab 기반 소프트웨어 PAM32는 BVA, 2CDE 및 PDA 분석을 실행할 수 있습니다.
공초점 단일 분자 FRET는 TIRF보다 훨씬 더 곧 살았던 상태 (~1 ms)를 쉽게 관찰 할 수 있습니다. 그러나, 확산에 의해 제한되는 짧은 관측 시간은 분자 기록을 주지 않으며, 표면 고정 실험이 할 수 있는 방식으로 더 이상 거주 시간 또는 복잡한 전환 네트워크를 결정할 수 없습니다.
프로토콜은 매우 낮은 농도에서 분자를 자유롭게 확산측정하므로 동일한 분자에서 분자 내 거리를 측정할 때 가장 잘 작동합니다. 두 분자의 Kd가 실험(~100 pM)에 요구되는 낮은 작업 농도에서 상당한 양으로 존재하는 복합체가 충분히 낮을 때 과도 결합 분자 사이의 분자 간 거리를 측정할 수 있다. Kd가 이것보다 훨씬 높으면, 노래로 표지된 분자만 볼 수 있습니다. 이 문제는 미세 유체학을 사용하여 두 개의 라벨이 부착된 구성 요소를 고농도로 혼합한 다음 복잡한 해리33,34 전에 목표를 빠르게 희석하고 흐르면 극복할 수 있습니다.
단일 분자 수준에서 FRET 효율성을 측정하는 것은 앙상블 실험에서 평균이 될 이질적인 하위 집단에 알기 때문에 앙상블 기술에 비해 상당한 이점이 있습니다. 또한 ALEX를 갖춘 단일 분자 FRET는 정확한 FRET 효율성에 대한 액세스를 제공하므로 정확한 거리로 변환할 수 있습니다. 이를 통해 상대 거리 변화를 단순히 조사하는 대신 보다 자세한 구조 정보를 결정할 수 있습니다. smfBox는 이러한 모든 이점과 기능을 제공하지만 공초점 smFRET23이 가능한 유사한 상용 현미경보다 훨씬 낮은 예산으로 구성될 수 있습니다.
smfBox는 smFRET 기술에 대한 진입장벽이 훨씬 낮기 때문에 연구원들이 형성적 변화를 측정할 수 있게 해주며, 단백질과 핵산 내및 핵산 사이의 정확한 거리7,8,9,10,11,35를 측정할 수 있습니다.
Disclosures
The authors have nothing to disclose.
Acknowledgements
저자는 감사하게 다음과 같은 자금 출처를 인정: BBSRC (BB / T008032/1); EPSRC (B.A.로의 학생) 및 MRC (A. R.-T.로의 학생).
Materials
| Amino modified oligonucleotide | Eurogentec | N/A | May be ordered from various suppliers or synthesised; amino modification enables labeling with NHS-ester modified dyes |
| Avalanche photodiode (APD) | Excelitas | SPCM-AQRH-14 | Two APDs are required for the smfBox setup |
| Bovine Serum Albumin (BSA) | Merck | A2153 | System dependant; imaging buffer component (0.1 mg/mL in buffer) |
| Compact Laser Combiner | OMICRON | LightHUB-2 | 515 nm (80 mW) and 638 nm (100 mW) lasers |
| Coverglass | VWR | 630-2742 | Thickness: 0.17 ± 0.01 mm, LxW: 22×22 mm |
| Cy3B | Cytiva | PA63101 | 1 mg, PA63100 (5 mg), PA96106 (25 mg) |
| FRETBursts Python Package | N/A | N/A | Open-source python package for burst analysis of freely-diffusing single-molecule FRET data: https://fretbursts.readthedocs.io |
| Imaging Buffer | N/A | System dependant; 5 mM NaCl, 20 mM MgCl2, 5 mM Tris pH 7.5 and 0.1 mg/mL BSA | |
| Immersion Oil | Olympus | IMMOIL-F30CC | |
| Jupyter notebooks | Project Jupyter | N/A | Open-source web application to create and share documents that contain live code, equations, visualizations and text; data analysis notebooks for smfBox can be found in the SI |
| Lens Tissue | ThorLabs | MC-5 | MC-50E is same item in bulk |
| Magnesium Chloride | Merck | M2670 | System dependant; imaging buffer component (20 mM in buffer) |
| MilliQ/Ultrapure water | N/A | ||
| Nanopoistioner | Piezoconcept | FOC300 | Nanopositioner for accurate positioning of microscope objective |
| NHS-ester modified ATTO-550 | ATTO-TEC | AD 550-31 | 1 mg, AD 550-35 (5 mg) |
| NHS-ester modified ATTO-647N | ATTO-TEC | AD 647N-31 | 1 mg, AD 647N-35 (5 mg) |
| Objective lens | Olympus | N1480700 | Olympus objective series from orignal smfBox discontinued; replaced by N5702300 |
| OMICRON Control Center (OCC)- laser control center | OMICRON | N/A | v3.5.34 – OMICRON laser driver software |
| Press-To-Seal silicone isolator | Grace Bio-Labs | 664201 | 8-9 mm Diameter x 1.7 mm Depth |
| smOTTER | N/A | N/A | Open-source acquisition software for the Craggs Lab smfBox: https://github.com/craggslab/smOTTER |
| Sodium Chloride | Merck | S7653 | System dependant; imaging buffer component (5 mM in buffer) |
| Tris base | Merck | 93362 | System dependant; imaging buffer component (5 mM, pH 7.5 in buffer) |
| Type I ultrapure water | Merck | ZIQ7000T0 | Milli-Q® IQ 7000 Ultrapure Water System |
References
- Forster, T. Intermolecular energy migration and fluorescence. Annals of Physics. 437 (1-2), 55-75 (1948).
- Stryer, L., Haugland, R. P. Energy transfer: a spectroscopic ruler. Proceedings of the National Academy of Sciences. 58 (2), 719-726 (1967).
- Hohlbein, J., et al. Conformational landscapes of DNA polymerase I and mutator derivatives establish fidelity checkpoints for nucleotide insertion. Nature Communications. 4 (1), 2131 (2013).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), (2018).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chemical Society Reviews. 43 (4), 1156-1171 (2014).
- Lerner, E., et al. The FRET-based structural dynamics challenge — community contributions to consistent and open science practices. arXiv. , (2020).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2017).
- Craggs, T. D., et al. Substrate conformational dynamics facilitate structure-specific recognition of gapped DNA by DNA polymerase. Nucleic Acids Research. 47 (20), 10788-10800 (2019).
- Tsytlonok, M., et al. Dynamic anticipation by Cdk2/Cyclin A-bound p27 mediates signal integration in cell cycle regulation. Nature Communications. 10 (1), 1676 (2019).
- Nagy, J., et al. Complete architecture of the archaeal RNA polymerase open complex from single-molecule FRET and NPS. Nature Communications. 6 (1), 6161 (2015).
- LeBlanc, S. J., et al. Coordinated protein and DNA conformational changes govern mismatch repair initiation by MutS. Nucleic Acids Research. 46 (20), 10782-10795 (2018).
- Segal, M., et al. High-throughput smFRET analysis of freely diffusing nucleic acid molecules and associated proteins. Methods. 169, 21-45 (2019).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences. 101 (24), 8936-8941 (2004).
- Kapanidis, A. N., et al. Alternating-laser excitation of single molecules. Accounts of Chemical Research. 38 (7), 523-533 (2005).
- Müller, B. K., Zaychikov, E., Brauchle, C., Lamb, D. C. Pulsed interleaved excitation. Biophysical Journal. 89 (5), 3508-3522 (2005).
- Laurence, T. A., Kong, X., Jager, M., Weiss, S. Probing structural heterogeneities and fluctuations of nucleic acids and denatured proteins. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17348-17353 (2005).
- Pollina, T., et al. PlanktonScope: Affordable modular imaging platform for citizen oceanography. bioRxiv. , 056978 (2020).
- Collins, J. T., et al. Robotic microscopy for everyone: the OpenFlexure microscope. Biomedical Optics Express. 11 (5), 2447-2460 (2020).
- Courtney, A., Alvey, L. M., Merces, G. O. T., Burke, N., Pickering, M. The Flexiscope: a low cost, flexible, convertible and modular microscope with automated scanning and micromanipulation. Royal Society Open Science. 7 (3), 191949 (2020).
- Martens, K. J. A., et al. Visualisation of dCas9 target search in vivo using an open-microscopy framework. Nature Communications. 10 (1), 3552 (2019).
- Auer, A., et al. Nanometer-scale multiplexed super-resolution imaging with an economic 3D-DNA-PAINT microscope. ChemPhysChem. 19 (22), 3024-3034 (2018).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , 424613 (2020).
- Ambrose, B., et al. The smfBox is an open-source platform for single-molecule FRET. Nature Communications. 11 (1), 5641 (2020).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An open file format for timestamp-based single-molecule fluorescence experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669-676 (2018).
- Ingargiola, A., Lerner, E., Chung, S., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing single-molecule FRET. PLOS One. 11 (8), 0160716 (2016).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule FRET experiments with burst variance analysis. Biophysical Journal. 100 (6), 1568-1577 (2011).
- Tomov, T. E., et al. Disentangling subpopulations in single-molecule FRET and ALEX experiments with photon distribution analysis. Biophysical Journal. 102 (5), 1163-1173 (2012).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Kalinin, S., Valeri, A., Antonik, M., Felekyan, S., Seidel, C. A. M. Detection of structural dynamics by FRET: A photon distribution and fluorescence lifetime analysis of systems with multiple states. The Journal of Physical Chemistry B. 114 (23), 7983-7995 (2010).
- Pirchi, M., et al. Photon-by-photon hidden Markov model analysis for microsecond single-molecule FRET kinetics. The Journal of Physical Chemistry B. 120 (51), 13065-13075 (2016).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. PAM: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophysical Journal. 114 (7), 1518-1528 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry B. 122 (49), 11554-11560 (2018).
- Bennet, I. A., et al. Regional conformational flexibility couples substrate specificity and scissile phosphate diester selectivity in human flap endonuclease 1. Nucleic Acids Research. 46 (11), 5618-5633 (2018).

