選択的リボソームプロファイリングによる共翻訳相互作用ネットワークのグローバル同定
Summary
共翻訳相互作用は、新生鎖修飾、標的化、折り畳み、およびアセンブリ経路において重要な役割を果たします。ここで、選択的リボソームプロファイリング、 in vivo、モデル真核生物 サッカロミセス・セレビシエにおけるこれらの相互作用の直接分析のための方法について説明する。
Abstract
近年、リボソームが私たちのmRNAを解読するだけでなく、混雑した細胞環境へのポリペプチド鎖の出現を導くことが明らかになりました。リボソームは、膜標的因子の空間的および速度論的に制御された結合、修飾酵素、および折り畳みシャペロンのためのプラットフォームを提供する。高次オリゴマー複合体への集合、ならびにタンパク質間ネットワーク形成ステップでさえ、合成と協調することが最近発見された。
ここでは、 インビボでの共翻訳相互作用を捕捉するために開発された方法である選択的リボソームプロファイリングについて説明する。リボソーム-新生鎖複合体を共翻訳インタラクターとともに捕捉するために必要なさまざまなアフィニティー精製ステップ、ならびにコドンに近い分解能で共翻訳相互作用を解読するために必要なmRNA抽出、サイズ排除、逆転写、ディープシーケンシング、ビッグデータ解析ステップについて詳述します。
Introduction
SeレクティブリボソームPロファイリング(SeRP)は、今日まで、共翻訳相互作用をインビボで、直接的に捕捉し、特徴付ける唯一の方法である1、2、3、4、5、6。SeRPは、任意の因子と翻訳リボソームとの相互作用のグローバルプロファイリングをコドン分解能2,7に近い状態で可能にする。
この方法は、増殖する細胞のフラッシュ凍結および活性翻訳の保存に依存している。次いで、細胞溶解物をRNase Iで処理して、「リボソームフットプリント」と呼ばれるリボソーム保護mRNA断片を除く細胞内のすべてのmRNAを消化する。その後、サンプルは 2 つの部分に分割されます。1つの部分は、すべての細胞リボソームフットプリントの単離に直接使用され、細胞内で進行中のすべての翻訳を表す。第2の部分は、例えば修飾酵素、転座因子、折り畳みシャペロン、および複合体集合相互作用など、関心のある因子に関連するリボソームの特定のサブセットの親和性精製に使用される。親和性精製されたリボソームフットプリントは、総称してインターアクトームと呼ばれる。次に、リボソームで保護されたmRNAを抽出し、cDNAライブラリーの生成に使用し、続いてディープシーケンシングを行います。
全トランスレートームサンプルとインターアクトームサンプルの比較分析により、関心のある因子に関連するすべてのorfの同定、および各orf相互作用プロファイルの特性評価が可能になります。このプロファイルは、解読されたコドンおよび新興ポリペプチド鎖のそれぞれの残基を推測できる正確な関与開始および終結配列、ならびに相互作用中のリボソーム速度変動を報告する7、8。図1は、このプロトコルを回路図として示しています。
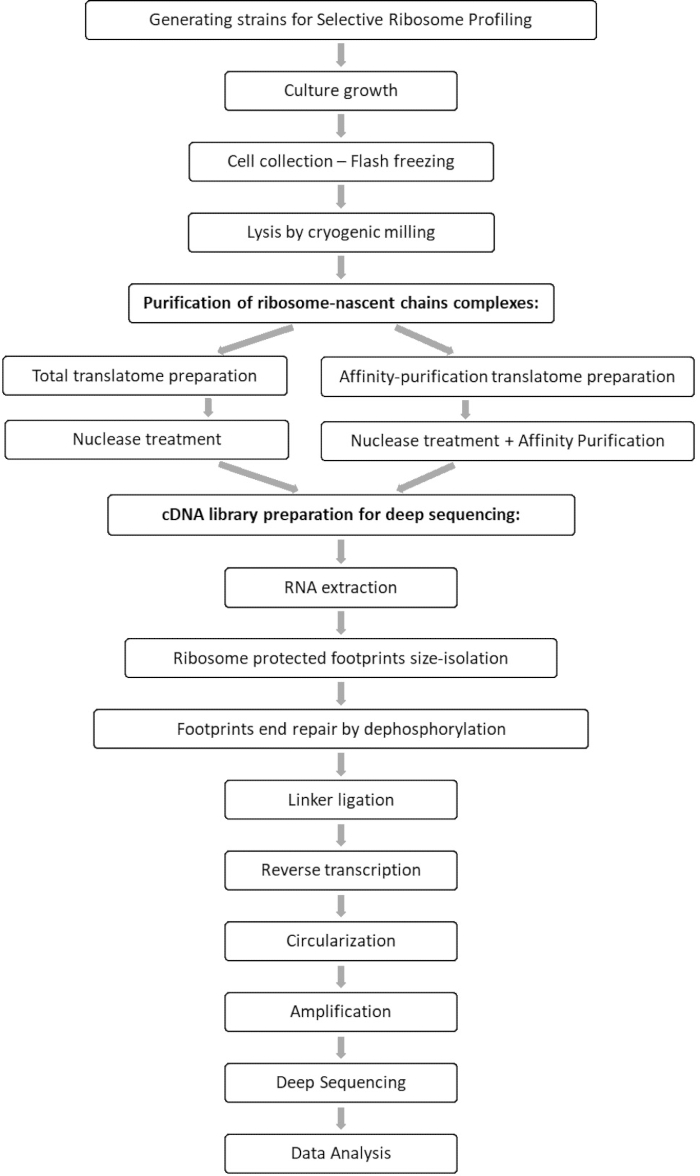
図 1: SeRP プロトコルの概要 このプロトコルは、7〜10日以内にその全体を実行することができます。 この図の拡大版を表示するには、ここをクリックしてください。
Protocol
Representative Results
Discussion
ここで、プロトコルは、コドン分解能に近い共翻訳相互作用を捕捉するための選択的リボソームプロファイリングアプローチを詳述する。リボソームが混雑した細胞質への新生鎖出現を調整するためのハブとして上昇するにつれて、これは機能的なプロテオームを確保するために必要な様々な共翻訳相互作用を同定し、特徴付けるために、そして様々な疾患を研究するための重要な方法であ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
実りある議論をしてくれた研究室のメンバー全員と、原稿を批判的に読んでくれたムハンマド・マフズミーに感謝します。この研究は、ISF(イスラエル科学財団)助成金2106/20によって資金提供されました。
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
References
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

