Wereldwijde identificatie van co-translationele interactienetwerken door selectieve ribosoomprofilering
Summary
Co-translationele interacties spelen een cruciale rol bij ontluikende ketenaanpassingen, targeting-, vouw- en assemblagepaden. Hier beschrijven we Selective Ribosome Profiling, een methode voor in vivo, directe analyse van deze interacties in het model eukaryote Saccharomyces cerevisiae.
Abstract
In de afgelopen jaren is het duidelijk geworden dat ribosomen niet alleen ons mRNA decoderen, maar ook de opkomst van de polypeptideketen in de drukke cellulaire omgeving begeleiden. Ribosomen bieden het platform voor ruimtelijk en kinetisch gecontroleerde binding van membraan-targeting factoren, het modificeren van enzymen en het vouwen van chaperonnes. Zelfs de assemblage in oligomere complexen van hoge orde, evenals eiwit-eiwitnetwerkvormingsstappen, werden onlangs ontdekt om te worden gecoördineerd met synthese.
Hier beschrijven we Selective Ribosome Profiling, een methode die is ontwikkeld om co-translationele interacties in vivo vast te leggen. We zullen de verschillende affiniteitszuiveringsstappen beschrijven die nodig zijn voor het vastleggen van ribosoom-ontluikende ketencomplexen samen met co-translationele interactoren, evenals de mRNA-extractie, grootte-uitsluiting, reverse transcriptie, deep-sequencing en big-data-analysestappen, die nodig zijn om co-translationele interacties in bijna-codonresolutie te ontcijferen.
Introduction
Selective Ribosome Profiling (SeRP) is tot op heden de enige methode die co-translationele interacties, in vivo, op een directe maniervastlegt en karakteriseert 1,2,3,4,5,6. SeRP maakt globale profilering van interacties van elke factor mogelijk met het vertalen van ribosomen in bijna codonresolutie 2,7.
De methode is gebaseerd op het flashvriezen van groeiende cellen en het behoud van actieve translatie. Cellysaten worden vervolgens behandeld met RNase I om al het mRNA in de cel te verteren, behalve ribosoom-beschermde mRNA-fragmenten die “ribosoomvoetafdrukken” worden genoemd. Het monster wordt vervolgens in twee delen gesplitst; een deel wordt direct gebruikt voor de isolatie van alle cellulaire ribosomale voetafdrukken, die alle lopende vertaling in de cel vertegenwoordigen. Het tweede deel wordt gebruikt voor de affiniteitszuivering van de specifieke subset van ribosomen geassocieerd met een factor van belang, bijvoorbeeld: het modificeren van enzymen, translocatiefactoren, vouw chaperonnes en complex-assemblage interacties. De affiniteitsgezuiverde ribosomale voetafdrukken worden gezamenlijk het interactoom genoemd. Vervolgens worden de ribosoombeveiligde mRNA’s geëxtraheerd en gebruikt voor het genereren van cDNA-bibliotheken, gevolgd door diepe sequencing.
Vergelijkende analyse van de totale translatoom- en interactoommonsters maakt het mogelijk om alle orfs te identificeren die verband houden met de factor van belang, evenals karakterisering van elk orf-interactieprofiel. Dit profiel rapporteert de precieze betrokkenheids- en beëindigingssequenties waaruit men de gedecodeerde codons en de respectieve residuen van de opkomende polypeptideketen kan afleiden, evenals op de ribosoomsnelheidsvariaties tijdens de interactie 7,8. Figuur 1 geeft het protocol weer als een schema.
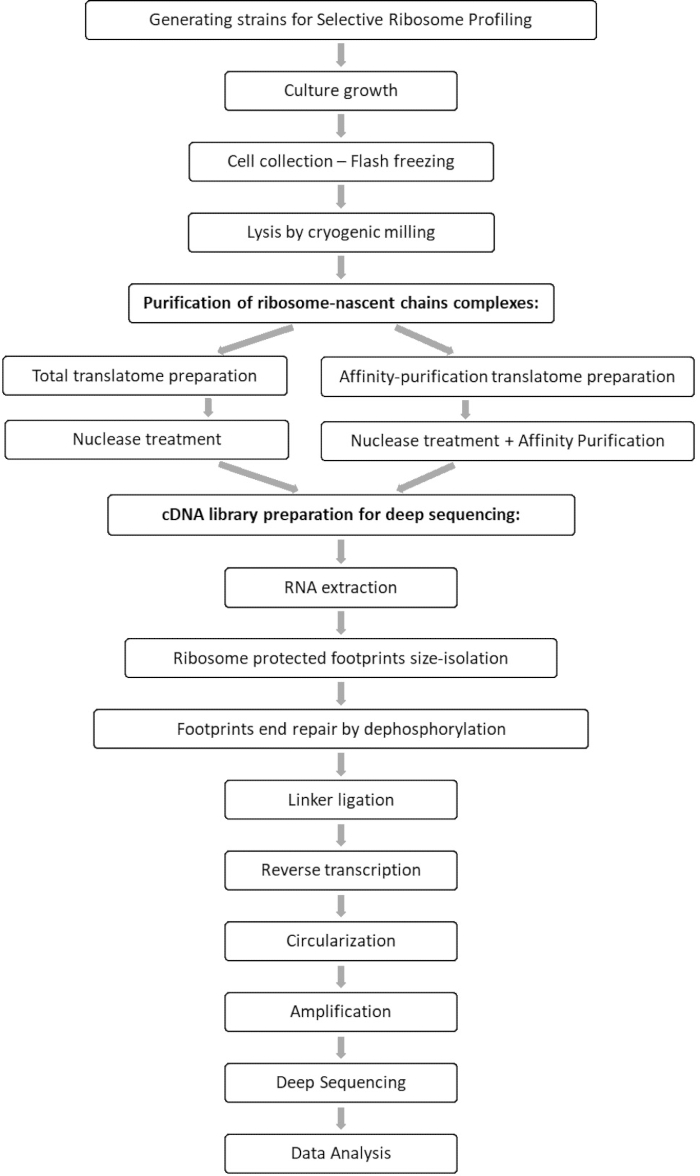
Figuur 1: Een overzicht van het SeRP-protocol. Dit protocol kan in zijn geheel binnen 7-10 dagen worden uitgevoerd. Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
Hier beschrijft het protocol de Selective Ribosome Profiling-benadering voor het vastleggen van co-translationele interacties in bijna codonresolutie. Naarmate het ribosoom stijgt als een hub voor het coördineren van de ontluikende ketenopkomst in het overvolle cytoplasma, is dit een cruciale methode om de verschillende co-translationele interacties te identificeren en te karakteriseren die nodig zijn om een functioneel proteoom te garanderen, evenals voor het bestuderen van verschillende ziekten. Tot op heden is SeRP …
Disclosures
The authors have nothing to disclose.
Acknowledgements
We willen alle lableden bedanken voor de vruchtbare discussies en Muhammad Makhzumy voor het kritisch lezen van het manuscript. Dit werk werd gefinancierd door ISF (Israeli Science Foundation) subsidie 2106/20.
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
References
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

