Análise da formação de complexos proteicos em concentrações micromolares por acoplamento de microfluídica com fotometria de massa
Summary
Este protocolo combina fotometria de massa com um novo sistema microfluídico para investigar interações proteína-proteína de baixa afinidade. Essa abordagem é baseada na rápida diluição de complexos altamente concentrados em solução, o que permite medições de baixa afinidade e amplia a aplicabilidade da fotometria de massa.
Abstract
A fotometria de massa é uma tecnologia versátil de medição de massa que permite o estudo de interações biomoleculares e formação de complexos em solução sem marcadores. A fotometria de massa é geralmente adequada para analisar amostras na faixa de concentração de 100 pM-100 nM. No entanto, em muitos sistemas biológicos, é necessário medir amostras mais concentradas para estudar interações de baixa afinidade ou transitórias. Aqui, demonstramos um método que expande efetivamente a faixa de concentrações de amostras que podem ser analisadas por fotometria de massa de nanomolar para dezenas de micromolares.
Neste protocolo, a fotometria de massa é combinada com um novo sistema microfluídico para investigar a formação de complexos proteicos em solução na faixa de concentração micromolar. Com o sistema de microfluídica, os usuários podem manter uma amostra em uma concentração mais alta desejada, seguida de diluição até a faixa nanomolar – vários milissegundos antes da medição da fotometria de massa. Devido à velocidade da diluição, os dados são obtidos antes que o equilíbrio da amostra tenha mudado (ou seja, dissociação do complexo).
A técnica é aplicada para medir as interações entre um anticorpo imunoglobulina G (IgG) e o receptor Fc neonatal, mostrando a formação de complexos de alta ordem que não foram quantificáveis com medidas de fotometria de massa estática.
Em conclusão, a combinação de fotometria de massa e microfluídica torna possível caracterizar amostras na faixa de concentração micromolar e é proficiente na medição de interações biomoleculares com afinidades mais fracas. Esses recursos podem ser aplicados em uma variedade de contextos – incluindo o desenvolvimento e o design de bioterapêuticos – permitindo a caracterização completa de diversas interações proteína-proteína.
Introduction
As interações proteína-proteína sublinham a maioria das funções celulares, desde a regulação imunológica até a replicação e tradução do DNA. Como resultado, há uma necessidade fundamental em todas as ciências da vida de investigar uma vasta gama de interações em diversos complexos heterogêneos que são comumente formados. No entanto, sua detecção, caracterização e quantificação são muitas vezes desafiadoras, principalmente para interações de baixa afinidade1.
Os ensaios de imunoprecipitação são frequentemente usados para detectar interações de alta afinidade, mas para interações transitórias e de baixa afinidade, a detecção é amplamente inviável2. Técnicas de fluorescência também podem ser usadas, mas requerem a adição potencialmente disruptiva de marcadores fluorescentes2. O Cryo-EM pode fornecer um instantâneo estrutural e uma leitura de conjunto dos complexos de proteínas formados com alta resolução espacial, mas também normalmente requer trabalhar em concentrações muito baixas para imagens de interações de baixa afinidade. O Cryo-EM também traz desafios relacionados ao custo, acessibilidade, preparação da amostra e tempo de análise3.
Além disso, a ressonância plasmônica de superfície (SPR) tornou-se uma forma popular de quantificar as interações proteína-proteína, embora exija imobilização proteica, o que pode afetar o equilíbrio de ligação e resultar em taxas de ativação variáveis, reduzindo assim a precisão da medição 4,5. Também envolve várias etapas do ensaio antes da coleta e análise dos dados6.
A fotometria de massa é uma técnica de molécula única que tem sido usada para analisar as interações proteína-proteína 5,6,7. Ele funciona medindo a massa de moléculas individuais ou complexos com base na luz que eles espalham quando pousam na superfície de uma lamínula de vidro8. Medições de fotometria de massa têm sido usadas para quantificar as afinidades de ligação da abundância relativa de parceiros de ligação e os complexos que eles formam5. No entanto, como outras técnicas de molécula única, a concentração da amostra a ser medida deve ser normalmente inferior a 100 nM. Se a concentração for maior, as moléculas que pousam na superfície do vidro se sobrepõem espacialmente, resultando em dados de baixa qualidade7. Consequentemente, interações mais fracas (KD ~ micromolares), que se dissociam nessas concentrações mais baixas, não podem ser medidas de forma confiável, uma vez que não é possível observar a mistura necessária de espécies não ligadas e ligadas5.
Aqui, descrevemos uma abordagem que supera essa limitação com base em um novo dispositivo de fotometria de massa microfluídica acoplada. Especificamente, um sistema microfluídico é usado em combinação com o fotômetro de massa para expandir efetivamente a gama de interações que podem ser quantificadas por fotometria de massa. A microfluídica demonstrou oferecer uma gama de possibilidades para investigar interações proteína-proteína, incluindo diluição rápida para detectar interações fracas 1,9. O sistema aqui descrito opera diluindo rapidamente a amostra em até 10.000 vezes em um chip microfluídico e fluindo-a imediatamente pela área de observação do chip, permitindo que a medição da fotometria de massa comece dentro de 50 ms a partir do momento em que as moléculas iniciaram o processo de diluição10. A diluição ocorre quando a amostra e o tampão são combinados em um misturador de válvula Tesla reverso no chip, com as taxas de fluxo relativas das duas soluções determinando a quantidade de diluição que ocorre (consulte a etapa 8 do protocolo). A vazão é controlável com o software de controle microfluídico. Alterar a vazão pode alterar a população relativa da espécie, pois pode afetar o número de eventos de pouso na superfície do vidro, que é o que é medido pelo fotômetro de massa.
A velocidade do processo é rápida o suficiente para que a medição seja concluída antes que a integridade da interação seja interrompida (para mais detalhes, consulte também a Discussão). Isso pode ser entendido através de uma breve olhada na teoria das reações de primeira ordem, onde 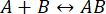 . A constante de taxa direta (associação) é kf, a constante de taxa inversa (dissociação) é kb e a constante de dissociação de equilíbrio (KD) é definida como
. A constante de taxa direta (associação) é kf, a constante de taxa inversa (dissociação) é kb e a constante de dissociação de equilíbrio (KD) é definida como
KD= kb/ kf
Para a ligação às proteínas, kfé geralmente limitado pela difusão dos reagentes11 e, portanto, é restrito à faixa de 106-10 7 M-1 · s-1. Como o intervalo de é limitado, uma reação de baixa afinidade (KD ~ micromolares) terá kb≈ 1 s-1. Ou seja, kb = kf · KD= (106 M-1·s-1) (10-6 M) = 1 s-1, com meia-vida do complexo em torno de 0,7 s11,12.
Nosso sistema de exemplo é a ligação do anticorpo monoclonal IgG trastuzumabe ao domínio solúvel do receptor Fc neonatal IgG (FcRn), que são conhecidos parceiros de interação13. Dados publicados anteriormente obtidos usando apenas fotometria de massa convencional (ou seja, com diluição manual de amostras) mostraram que as proteínas formam várias espécies. Monômeros de FcRn, dímeros de FcRn e IgG não ligados foram claramente visíveis, enquanto complexos IgG-FcRn (nas proporções de 1:1 e 1:2) também foram detectados (em pH 5,0), mas apenas com abundância muito baixa5. Essa observação levanta a questão de saber se a formação do complexo IgG-FcRn poderia ser detectada com mais clareza se medida em uma concentração mais alta. De fato, a combinação de fotometria de massa com uma abordagem de diluição rápida acoplada descrita aqui forneceu evidências mais robustas de formação complexa por um aumento em suas partículas medidas.
O protocolo de fotometria de massa e microfluídica aqui descrito permite caracterizar a formação de complexos com KD até a faixa micromolar. Uma determinação empírica do KD exigirá melhorias adicionais na precisão do sensor de fluxo, estabilidade da bomba, variações de chip a chip e localização de medição dentro da janela de observação, pois todos esses fatores influenciariam o tempo desde o momento em que a amostra é diluída até a medição.
A mesma abordagem pode ser aplicada para investigar a ligação entre quaisquer proteínas solúveis, desde que tenham pesos moleculares distintos (separados por pelo menos 25 kDa) que se enquadrem na faixa adequada para análise com um fotômetro de massa (30 kDa a 6 MDa). Os insights obtidos podem ser úteis para estudos em uma variedade de contextos – desde a obtenção de uma compreensão mecanicista das funções celulares até o design de novos medicamentos bioterapêuticos.
Protocol
Representative Results
Discussion
O protocolo descrito aqui fornece um método para detectar e quantificar interações proteína-proteína de baixa afinidade. Ele usa um fotômetro de massa acoplado a um sistema microfluídico de diluição rápida. A fotometria de massa é uma ferramenta bioanalítica sem rótulo que pode medir de forma confiável a massa molecular em solução para biomoléculas16, para aquelas dentro da faixa de 30 kDa a 6 MDa. Como a fotometria de massa é uma técnica de molécula única que analisa as amostras uma a uma, geralmente é limitada a amostras na faixa de concentração de 100 pM-100 nM. Acima dessa faixa, as moléculas que pousam na superfície do vidro se sobrepõem espacialmente, resultando em dados de baixa qualidade; abaixo desse intervalo, poucos dados são obtidos para fazer análises robustas7. Uma consequência importante é que pode limitar a investigação das interações proteicas àquelas que formam uma mistura de espécies ligadas e não ligadas dentro dessa faixa.
Aqui, detalhamos um protocolo passo a passo para usar um sistema microfluídico de diluição rápida para expandir efetivamente a faixa de concentrações de amostras que são passíveis de fotometria em massa. Ao diluir a amostra no chip microfluídico e, em seguida, fluí-la pela janela de observação do detector em 50 ms, o sistema captura os complexos presentes na amostra não diluída antes que o equilíbrio de interação mude. A amostra é continuamente entregue ao detector durante medições individuais. Nessas condições, 95% do complexo permanecerá intacto quando a amostra for medida, mesmo para interações de baixa afinidade – com um KD da ordem de micromolares e taxas de dissociação tão rápidas quanto 1 s-1.
Isso pode ser calculado da seguinte forma: Para uma reação 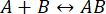 com uma taxa direta kfe uma taxa regressiva kb,
com uma taxa direta kfe uma taxa regressiva kb,
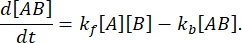
Em equilíbrio, as concentrações de todas as três espécies (A, B e o complexo AB) permanecem constantes, assim 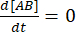 e
e 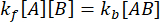 . Sob a suposição conservadora de que a perturbação (diluição, neste caso) pode fazer com que o complexo se dissocie, mas a reação direta (associação) não prossegue, o termo kf [A] [B] pode ser tratado como insignificante, e a seguinte simplificação pode ser feita:
. Sob a suposição conservadora de que a perturbação (diluição, neste caso) pode fazer com que o complexo se dissocie, mas a reação direta (associação) não prossegue, o termo kf [A] [B] pode ser tratado como insignificante, e a seguinte simplificação pode ser feita:
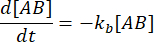
A integração dá a seguinte expressão para a concentração do complexo no tempo após a perturbação do equilíbrio:
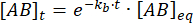
A fração do complexo que permanece ligada no tempo t após a perturbação do equilíbrio é assim:
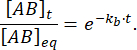
Em = 50 ms, para uma reação com kb≈ 1 s-1, o limite da fração é 0,95, ou 95%11,12.
A fotometria de massa foi usada aqui e anteriormente5 para investigar a ligação do anticorpo monoclonal IgG trastuzumabe ao domínio solúvel do FcRn. Foi relatado que os dois parceiros de ligação se ligam com afinidade nanomolar em pH ácido17. A fotometria de massa foi usada para avaliar qualitativamente a abundância dos complexos formados enquanto os parceiros de ligação estavam em pH 5,0, e as amostras foram rapidamente diluídas por meio de um sistema microfluídico adicional. O procedimento foi otimizado para a interação proteína-proteína específica com base em resultados relatados anteriormente5. O mesmo procedimento pode ser usado para estudar outras interações, desde que os usuários tenham conhecimento prévio ou otimizem as condições experimentais para o sistema em questão, como quais tampões usar, a concentração inicial de proteínas, a estequiometria esperada e a quantidade de incubação necessária para permitir que a interação atinja um equilíbrio.
Quando a mistura IgG-FcRn foi diluída manualmente, foi difícil detectar a presença de complexos IgG-FcRn, embora essas proteínas sejam conhecidas por interagir5. Este artigo mostra que a abordagem de diluição rápida resulta em um aumento notável da quantidade desses complexos. Para a mesma amostra, quando a diluição rápida foi usada, os complexos FcRn-IgG 1:1 e os complexos FcRn-IgG 2:1 foram claramente observados. Essas diferenças na formação de complexos demonstram a importância de estudar sistemas de interação biomolecular em uma ampla gama de concentrações.
Além disso, esses resultados também demonstram que é simples usar microfluídica com análise de molécula única para capturar interações fracas – preenchendo uma lacuna significativa no método. A combinação de microfluídica de diluição rápida com fotometria de massa oferece vantagens atraentes devido às vantagens da fotometria de massa como técnica analítica. Ou seja, a fotometria de massa não requer rótulos, envolve uma preparação mínima da amostra e as medições são feitas em solução. Para este protocolo, outra vantagem importante da fotometria de massa é sua capacidade de distinguir e quantificar todas as espécies formadas (desde que tenham uma massa distinta de >30 kDa). Isso contrasta com o SPR, por exemplo, que pode medir as taxas de ligação e desvinculação, mas não pode fornecer prontamente informações estequiométricas8.
Para este protocolo, bem como experimentos de fotometria de massa em geral, várias considerações são úteis. Primeiro, a concentração final da proteína deve estar dentro do limite do que a fotometria de massa pode medir (100 pM-100 nM). A concentração inicial de incubação também deve estar dentro da faixa do sistema microfluídico (até 90 μM) e teorizada como acima do KD real da interação10. O ponto de partida recomendado é uma proporção de mistura de concentração de 1:1 entre as espécies que interagem na concentração de μM. A proporção pode então ser variada para 1:2, 1:5 ou, como no caso dessa interação, 1:10. Se não houver informações prévias sobre as interações proteicas, o usuário teria que otimizar o experimento, começando com uma alta concentração (recomendado 20 μM) para cada parceiro para determinar se a afinidade dos componentes está dentro da faixa de concentração sustentada pelo método apresentado (ou seja, complexos são formados). A otimização também pode envolver a escolha de outras condições de buffer para promover as interações ou titulação de um dos componentes de interação para determinar a proporção de mistura correta. Uma vez determinados, é possível otimizar as concentrações e fluxos para permitir condições ideais para o estudo e o método, por exemplo, diminuindo as concentrações para permitir uma melhor resolução de pico.
Em segundo lugar, para replicar com sucesso este experimento, as impurezas devem ser minimizadas. Fontes comuns de impurezas que afetam adversamente as medições de fotometria de massa incluem outras proteínas ou detritos celulares que permanecem após a purificação, tampões não filtrados, detergentes formadores de micela (se presentes em uma concentração muito alta) e tampões contendo altas concentrações de sal, glicerol ou outros componentes. Conforme discutido no Protocolo acima, as bolhas no sistema microfluídico devem ser removidas. Bolhas podem se formar no sistema de tubulação ou se as amostras tiverem alta tensão superficial e forem propensas à formação de espuma. Bolhas também podem se formar no óleo de imersão, que podem ser detectadas a partir do anel de foco (Figura 3). Se as bolhas não puderem ser removidas usando as etapas descritas no protocolo, outra solução é desgaseificar a amostra usando um dessecador e uma bomba de vácuo, deixando a amostra sob pressão reduzida por alguns minutos. Não é recomendado o vórtice ou agitação de soluções proteicas altamente concentradas, pois essas ações podem promover a formação de bolhas.
Embora a medição de uma interação proteína-proteína específica seja demonstrada aqui, o mesmo protocolo pode ser aplicado a outros sistemas de interação proteína-proteína sem modificação significativa. Uma outra direção futura deste protocolo seria usar as medições para calcular os valores de KD para os complexos identificados, como foi descrito em outro lugar no contexto da fotometria de massa 5,7. Embora os estudos anteriores tenham usado dados de experimentos envolvendo diluição manual e interações mais fortes, o princípio de análise pode ser prontamente aplicado neste contexto – desde que outras melhorias no dispositivo microfluídico sejam implementadas (como maior precisão do sensor de fluxo e estabilidade da bomba).
Além das interações proteína-proteína, é provável que haja aplicações mais amplas para a abordagem combinada de fotometria de massa e microfluídica de diluição rápida. A fotometria de massa pode ser usada para avaliar a pureza, agregação e homogeneidade da amostra18,19; estudar oligomerização de proteínas20, montagem macromolecular21 ou polimerização22; e em outras áreas. A análise de fotometria de massa também se estende além das proteínas; Tem sido usado para investigar interações entre ácidos nucléicos e proteínas23, partículas virais24 e nanopartículas25. Este protocolo descreve, portanto, uma importante aplicação de um sistema microfluídico de fotometria de massa combinado – ele permite a medição direta de interações proteína-proteína fracas no nível de moléculas e complexos individuais. O valor da presente aplicação é alto, pois abre a possibilidade de caracterizar diretamente interações que geralmente têm sido difíceis de estudar – com relevância em áreas terapêuticas críticas. Essa abordagem combinada também pode servir de base para uma gama mais ampla de investigações para amostras com concentrações de até dezenas de micromolares.
Disclosures
The authors have nothing to disclose.
Acknowledgements
WS é apoiado por uma bolsa de futuros líderes do UKRI [MR / V02213X / 1]. O texto e os gráficos do manuscrito foram preparados com o apoio de membros da equipe de comunicação científica de Refeyn (Panagiota Paganopoulou, Neus Torres Tamarit e Catherine Lichten). Também agradecemos o feedback valioso de Camille Hetez, Sofia Ferreira e Matthias Langhorst.
Materials
| 2-Propanol (Isopropanol) | VWR International LLC | 20880.320 | |
| Data acquisition software | Refeyn | AcquireMP (v2022 R1) | |
| Data analysis software | Refeyn | DiscoverMP (v2022 R1) | |
| FCRN, His-Tag | Sigma | SRP0624 | |
| Herceptin (IgG) | Cambridge Bioscience | HY-P9907-1mg | |
| Mass photometer | Refeyn | TwoMP | |
| Microfluidics box | Refeyn | MassFluidix HC system | |
| Microfluidics chip | Refeyn | MassFluidix HC chip | |
| Microfluidics control software | Fluigent | OxyGEN | |
| Phosphate Buffered Saline (PBS), 1x Ultra Pure | VWR International LLC | K812 | |
| Sodium Hydroxide (NaOH) | Sigma | S2770 | |
| β-Amylase, from sweet potato | Sigma | A8781 |
References
- Arter, W. E., Levin, A., Krainer, G., Knowles, T. P. J. Microfluidic approaches for the analysis of protein-protein interactions in solution. Biophysical Reviews. 12 (2), 575-585 (2020).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry. B. 122 (49), 11554-11560 (2018).
- Li, Z. Editorial: Methods in structural biology: Cryo-electron microscopy. Frontiers in Molecular Biosciences. 9, 1041386 (2022).
- Herling, T. W., et al. A microfluidic platform for real-time detection and quantification of protein-ligand interactions. Biophysical Journal. 110 (9), 1957-1966 (2016).
- Soltermann, F., et al. Quantifying protein-protein interactions by molecular counting with mass photometry. Angewandte Chemie International Edition. 59 (27), 10774-10779 (2020).
- Wu, D., Piszczek, G. Rapid determination of antibody-antigen affinity by mass photometry. Journal of Visualized Experiments: JoVE. 168, 61784 (2021).
- Wu, D., Piszczek, G. Measuring the affinity of protein-protein interactions on a single-molecule level by mass photometry. Analytical Biochemistry. 592, 113575 (2020).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- MassFluidix® HC system for rapid dilution via microfluidics. Available from: https://www.refeyn.com/massfluidix-hc-system (2023)
- Pollard, T. D. A guide to simple and informative binding assays. Molecular Biology of the Cell. 21 (23), 4061-4067 (2010).
- Jarmoskaite, I., AlSadhan, I., Vaidyanathan, P. P., Herschlag, D. How to measure and evaluate binding affinities. eLife. 9, e57264 (2020).
- Monnet, C., et al. Selection of IgG variants with increased FcRn binding using random and directed mutagenesis: Impact on effector functions. Frontiers in Immunology. 6, 39 (2015).
- . Refeyn TwoMP: Transforming biomolecular characterisation Available from: https://www.refeyn.com/twomp-mass-photometer (2022)
- Lai, S. -. H., Tamara, S., Heck, A. J. R. Single-particle mass analysis of intact ribosomes by mass photometry and Orbitrap-based charge detection mass spectrometry. iScience. 24 (11), 103211 (2021).
- Wu, D., Piszczek, G. Standard protocol for mass photometry experiments. European Biophysics Journal. 50 (3-4), 403-409 (2021).
- Vaughn, D. E., Bjorkman, P. J. Structural basis of pH-dependent antibody binding by the neonatal Fc receptor. Structure. 6 (1), 63-73 (1998).
- Niebling, S., et al. Biophysical screening pipeline for Cryo-EM grid preparation of membrane proteins. Frontiers in Molecular Biosciences. 9, 882288 (2022).
- Paul, S. S., Lyons, A., Kirchner, R., Woodside, M. T. Quantifying oligomer populations in real time during protein aggregation using single-molecule mass photometry. ACS Nano. 16 (10), 16462-16470 (2022).
- Schulz, L., et al. Evolution of increased complexity and specificity at the dawn of form I Rubiscos. Science. 378 (6616), 155-160 (2022).
- Malay, A. D., et al. An ultra-stable gold-coordinated protein cage displaying reversible assembly. Nature. 569 (7756), 438-442 (2019).
- Hundt, N., Cole, D., Hantke, M. F., Miller, J. J., Struwe, W. B., Kukura, P. Direct observation of the molecular mechanism underlying protein polymerization. Science Advances. 8 (35), eabm7935 (2022).
- Acharya, A., et al. Distinct RPA domains promote recruitment and the helicase-nuclease activities of Dna2. Nature Communications. 12, 6521 (2021).
- Ebberink, E. H. T. M., Ruisinger, A., Nuebel, M., Thomann, M., Heck, A. J. R. Assessing production variability in empty and filled adeno-associated viruses by single molecule mass analyses. Molecular Therapy – Methods & Clinical Development. 27, 491-501 (2022).
- Melo, L., et al. Size distributions of gold nanoparticles in solution measured by single-particle mass photometry. The Journal of Physical Chemistry B. 125 (45), 12466-12475 (2021).

