Erosie-identificatie in metacarpofalangeale gewrichten bij reumatoïde artritis met behulp van perifere kwantitatieve computertomografie met hoge resolutie
Summary
Boterosies zijn een belangrijk pathologisch kenmerk van reumatoïde artritis. Het doel van dit werk is het introduceren van een trainingstool om gebruikers te voorzien van richtlijnen voor het identificeren van pathologische corticale breuken op perifere kwantitatieve computertomografiebeelden met hoge resolutie voor erosieanalyse.
Abstract
Boterosies zijn een pathologisch kenmerk van verschillende vormen van inflammatoire artritis, waaronder reumatoïde artritis (RA). De toegenomen aanwezigheid en omvang van erosies worden in verband gebracht met slechte resultaten, gewrichtsfunctie en ziekteprogressie. Perifere kwantitatieve computertomografie met hoge resolutie (HR-pQCT) biedt een ongeëvenaarde in vivo visualisatie van boteroversies. Bij deze resolutie zijn echter ook discontinuïteiten in de corticale schil (corticale breuken) zichtbaar die geassocieerd zijn met normale fysiologische processen en pathologie. De studie voor xtrEme computertomografie bij reumatoïde artritis gebruikte eerder een consensusproces om een definitie van pathologische erosie in HR-pQCT te ontwikkelen: een corticale breuk gedetecteerd in ten minste twee opeenvolgende plakjes, in ten minste twee loodrechte vlakken, niet-lineair van vorm, met onderliggend trabeculair botverlies. Ondanks de beschikbaarheid van een consensusdefinitie is erosie-identificatie echter een veeleisende taak met uitdagingen op het gebied van interbeoordelaarsvariabiliteit. Het doel van dit werk is om een trainingstool te introduceren om gebruikers te begeleiden bij het identificeren van pathologische corticale breuken op HR-pQCT-beelden voor erosieanalyse. Het hier gepresenteerde protocol maakt gebruik van een op maat gemaakte module (Bone Analysis Module (BAM) – Training), geïmplementeerd als uitbreiding op een open-source beeldverwerkingssoftware (3D Slicer). Met behulp van deze module kunnen gebruikers oefenen met het identificeren van erosies en hun resultaten vergelijken met erosies die zijn geannoteerd door deskundige reumatologen.
Introduction
Boterosies treden op wanneer een ontsteking plaatselijk botverlies veroorzaakt aan het corticale botoppervlak. Deze erosies strekken zich uit tot in het onderliggende trabeculaire botgebied. Ze zijn een pathologisch kenmerk van verschillende vormen van inflammatoire artritis, waaronder reumatoïde artritis (RA)1. De aanwezigheid en grootte van erosie worden in verband gebracht met slechte resultaten, patiëntfunctie en ziekteprogressie 2,3,4,5. Hoewel gewone radiografie de klinische standaard blijft voor erosiebeoordeling, biedt perifere kwantitatieve computertomografie met hoge resolutie (HR-pQCT) 3D-beelden en superieure gevoeligheid en specificiteit voor erosiedetectie 6,7. Voor inflammatoire artritis, zoals RA, wordt HR-pQCT vaak uitgevoerd op de2e en 3e metacarpofalangeale gewrichten – de meest aangetaste gewrichten van de hand8. Omdat HR-pQCT-beelden een hoge ruimtelijke resolutie hebben, worden fysiologische onderbrekingen in het corticale oppervlak ook waargenomen bij gezonde personen zonder RA9. Deze corticale onderbrekingen worden vaak geassocieerd met vasculaire kanalen of foramen van voedingsstoffen die door het bot gaan10. De uitdaging is dus om corticale onderbrekingen geassocieerd met een ziekteproces (d.w.z. pathologische erosies) te onderscheiden van niet-pathologische kenmerken.
De consensusdefinitie van een pathologische boterosie werd gepubliceerd door de Study grouP for xtrEme Computed Tomography in Rheumatoid Arthritis (SPECTRA) als de aanwezigheid van een duidelijke onderbreking in de corticale laag van het bot die zich uitstrekt over ten minste twee opeenvolgende plakjes en detecteerbaar is in twee of meer loodrechte vlakken11. Verder moet de onderbreking niet-lineair van vorm zijn en gepaard gaan met een verlies in het trabeculaire gebied. Visuele voorbeelden van corticale onderbrekingen die wel en niet voldoen aan de criteria van erosie worden getoond in Klose-Jensen et al.12.
Niet alle corticale onderbrekingen die aan de bovenstaande criteria voldoen, worden echter geclassificeerd als erosie. Onderbrekingen worden soms veroorzaakt door fysiologische processen zoals vasculaire kanalen (figuur 1). Deze kunnen worden geïdentificeerd en onderscheiden van erosies vanwege hun voorspelbare anatomische locaties, parallelle en rechte marges en submillimetergrootte13. Cysten zijn een andere vorm van corticale onderbreking die niet als erosie wordt beschouwd. Ze hebben vaak een afgeronde trabeculaire structuur met een duidelijke cystische wand 13. In tegenstelling tot de scherpe randen en de open trabeculaire structuur die door erosies worden getoond. Het is echter mogelijk dat erosies zich vormen op cystische plaatsen, waardoor het dubbelzinnig is om het volume van botverlies af te bakenen dat wordt veroorzaakt door de erosies en niet door de cysten. Hoewel het oplossen van deze ambiguïteit met verdere criteria niet het doel van deze studie is, is het nodig om uitgebreide voorbeelden te geven van pathologische erosie en fysiologische corticale onderbrekingen.
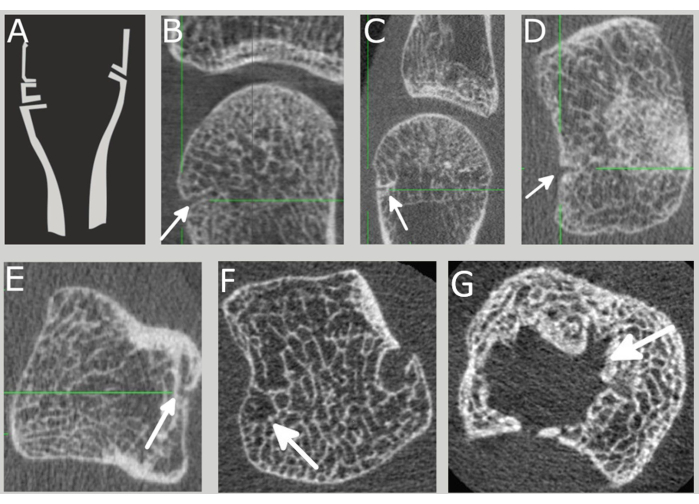
Figuur 1: Voorbeeld van corticale onderbrekingen die niet alleen door erosies werden veroorzaakt. (A) Een tekening die de gemeenschappelijke locatie van vasculaire kanalen aan de basis van de middenhandsbeentje illustreert. Voorbeelden van vasculaire kanalen in (B) coronale, (C) sagittale, (D) en (E) axiale vlakken. (F) Voorbeeld van een corticale onderbreking veroorzaakt door een cyste. (G) Voorbeeld van een leegtevolume in het trabeculaire gebied van het bot waarbij zowel cysten als erosies betrokken zijn. Klik hier om een grotere versie van deze figuur te bekijken.
Ondanks de uitdagingen bij het identificeren van erosie, zijn er momenteel geen trainingstools om minder ervaren gebruikers te begeleiden bij het interpreteren van HR-pQCT-beelden voor erosieanalyse. Onlangs is een open-source module voor erosieanalyse ontwikkeld, genaamd bone analysis module (BAM) – Erosion Volume, geïmplementeerd als uitbreiding op een open-source beeldverwerkingssoftware om erosievisualisatie en volumetrische analyses mogelijk te maken14. Het hier gepresenteerde protocol beschrijft het gebruik van een trainingsmodule die aan BAM is toegevoegd (BAM – Training), die de erosie-identificatiepogingen van een gebruiker vergelijkt door de erosie-identificatie te vergelijken met erosie-identificaties die zijn geannoteerd door deskundige reumatologen. Deze trainingstool geeft gebruikers feedback over erosie-identificatie om verbeteringen in erosieanalyse te begeleiden. Instructies voor het installeren van de software vindt u in stap 1. Voor nieuwe gegevensverzameling, zie stap 3 – 5.3. Alleen voor het gebruik van trainingsmodules, zie stap 2.
Protocol
Representative Results
Discussion
Deze trainingstool biedt de mogelijkheid om erosies te leren identificeren met behulp van de botanalysemodule. Verder gebruik van deze erosieanalysetool buiten de training vereist toegang tot beelden van goede kwaliteit, met weinig of geen bewegingsartefacten. De definitie van HR-pQCT-erosie op basis van de literatuur beschrijft anatomische kenmerken die verband houden met pathologische erosies en die met een redelijke reproduceerbaarheid kunnen worden gerapporteerd11,20. Deze definitie houdt echter geen rekening met gemeenschappelijke anatomische locaties van vasculaire kanalen, wat mogelijk kan leiden tot hun verkeerde classificatie als boterosies10.
De cruciale stappen in dit protocol zijn het genereren van het botmasker, het plaatsen van de zaadpunten en het genereren van het erosievolume. Hoewel geautomatiseerde methoden worden geïmplementeerd om de maskers en het erosievolume te genereren, vereisen de maskers vaak handmatige correctie om bevredigende resultaten te garanderen. Er wordt een uitgebreide beschrijving gegeven van de tools die beschikbaar zijn om de handmatige correcties uit te voeren. De plaatsing van de startpunten wordt geleid door de trainingsvoorbeelden van de BAM-Training-module.
Op basis van de tot nu toe gebruikte gegevens geeft dit protocol suggesties voor het oplossen van problemen wanneer de erosieanalysemodule niet de verwachte resultaten oplevert. In de toekomst zal toegang worden verleend tot aanvullende opleidingsgegevens. Een eerdere studie toonde aan dat de erosievolumes die met deze methode worden beoordeeld, vergelijkbaar zijn met bestaande methoden 14,21,22. Aan de hand van opleidingsgegevens kunnen nieuwere instrumenten voor erosieanalyse worden vergeleken met de ontwikkeling ervan23.
De hier geïntroduceerde trainingstool helpt in de eerste plaats bij het identificeren van erosie; De methode wordt momenteel echter beperkt door het ontbreken van een consensus over het definiëren van de omvang van een erosie in het trabeculaire bot. Desalniettemin zijn de BAM-modules open source, dus naarmate toekomstige definities van erosieomvang veranderen, hebben andere onderzoekers toegang om de modules aan te passen aan hun behoeften.
Naarmate het gebruik van HR-pQCT in reumatologisch onderzoek zich uitbreidt, biedt de trainingstool onervaren gebruikers richtlijnen voor het identificeren van pathologische corticale onderbrekingen op HR-pQCT-beelden voor erosieanalyse. Deze tool zal toepasbaar zijn op onderzoekers, ongeacht de methode die is geselecteerd voor erosieanalyse. Hoewel volledig geautomatiseerde erosie-identificatie wenselijk is om de reproduceerbaarheid en snelheid van analyse te verbeteren, zijn grote referentie-/benchmarkdatasets met nauwkeurige annotaties vereist om machine learning-modellen te trainen. Als open-source tool biedt deze module de mogelijkheid om gezamenlijk grote, geannoteerde datasets te ontwikkelen voor toekomstig gebruik in machine learning. Het gebruik van deze trainingstool zal meer onderzoekers in staat stellen erosieanalyse op te nemen in hun HR-pQCT-onderzoek.
Disclosures
The authors have nothing to disclose.
Acknowledgements
De auteurs willen graag de volgende financieringsinstanties erkennen die dit werk hebben ondersteund. SLM wordt gefinancierd door The Arthritis Society (STAR-18-0189) en Canadian Institutes of Health Research Planning and Dissemination Grant. JJT heeft een CIHR Fellowship-prijs.
Materials
| 3DSlicer | Open Source | N/A | Download at https://www.slicer.org/ |
| BAM Erosion Analysis Modules | Open Source | N/A | Version used in manuscript: download at https://doi.org/10.5281/zenodo.7943007 |
| XtremeCTII | Scanco Medical | N/A |
References
- Schett, G., Gravallese, E. Bone erosion in rheumatoid arthritis: mechanisms, diagnosis and treatment. Nature Reviews Rheumatology. 8 (11), 656-664 (2012).
- Ødegård, S., et al. Association of early radiographic damage with impaired physical function in rheumatoid arthritis: A ten-year, longitudinal observational study in 238 patients. Arthritis & Rheumatism. 54 (1), 68-75 (2006).
- Scott, D. L., et al. The links between joint damage and disability in rheumatoid arthritis. Rheumatology. 39 (2), 122-132 (2000).
- van Nies, J. A. B., et al. Evaluating processes underlying the predictive value of baseline erosions for future radiological damage in early rheumatoid arthritis. Annals of Rheumatic Diseases. 74 (5), 883-889 (2015).
- Versteeg, G. A., et al. Early radiological progression remains associated with long-term joint damage in real-world rheumatoid arthritis patients treated to the target of remission. Scandinavian Journal of Rheumatology. , (2021).
- Brunet, S. C., et al. Bone changes in early inflammatory arthritis assessed with High-Resolution peripheral Quantitative Computed Tomography (HR-pQCT): A 12-month cohort study. Joint Bone Spine. 88 (1), 105065 (2021).
- Finzel, S., et al. Repair of bone erosions in rheumatoid arthritis treated with tumour necrosis factor inhibitors is based on bone apposition at the base of the erosion. Annals of Rheumatic Diseases. 70 (9), 1587-1593 (2011).
- Barnabe, C., Feehan, L. High-resolution peripheral quantitative computed tomography imaging protocol for metacarpophalangeal joints in inflammatory arthritis: the SPECTRA collaboration. The Journal of Rheumatology. 39 (7), 1494-1495 (2012).
- Peters, M., et al. Assessment of Cortical Interruptions in the Finger Joints of Patients With Rheumatoid Arthritis Using HR-pQCT, Radiography, and MRI. Journal of Bone and Mineral Research. 33 (9), 1676-1685 (2018).
- Scharmga, A., et al. Vascular channels in metacarpophalangeal joints: a comparative histologic and high-resolution imaging study. Scientific reports. 7 (1), 8966-8968 (2017).
- Barnabe, C., et al. Definition for Rheumatoid Arthritis Erosions Imaged with High Resolution Peripheral Quantitative Computed Tomography and Interreader Reliability for Detection and Measurement. The Journal of Rheumatology. 43 (10), 1935-1940 (2016).
- Klose-Jensen, R., et al. High-Resolution Peripheral Quantitative Computed Tomography for Bone Evaluation in Inflammatory Rheumatic Disease. Frontiers in Medicine. 7, 337 (2020).
- Blavnsfeldt, A. G., et al. Effect of radiographic disease severity in high-resolution quantitative computed tomography assessment of metacarpophalangeal joint erosion and cysts. International Journal of Rheumatic Diseases. 24 (1), 112-119 (2021).
- Zhao, M., et al. Open-source image analysis tool for the identification and quantification of cortical interruptions and bone erosions in high-resolution peripheral quantitative computed tomography images of patients with rheumatoid arthritis. Bone. 165, 116571 (2022).
- Fedorov, A., et al. 3D Slicer as an image computing platform for the Quantitative Imaging Network. Magnetic Resonance Imaging. 30 (9), 1323-1341 (2012).
- Pauchard, Y., Liphardt, A. -. M., Macdonald, H. M., Hanley, D. A., Boyd, S. K. Quality control for bone quality parameters affected by subject motion in high-resolution peripheral quantitative computed tomography. Bone. 50 (6), 1304-1310 (2012).
- Sode, M., Burghardt, A. J., Pialat, J. -. B., Link, T. M., Majumdar, S. Quantitative characterization of subject motion in HR-pQCT images of the distal radius and tibia. Bone. 48 (6), 1291-1297 (2011).
- Stok, K. S., et al. Consensus approach for 3D joint space width of metacarpophalangeal joints of rheumatoid arthritis patients using high-resolution peripheral quantitative computed tomography. Quantitative imaging in medicine and surgery. 10 (2), 314-325 (2020).
- . Open and Reproducible Musculoskeletal Imaging Research Available from: https://github.com/SpectraCollab/ORMIR_XCT (2023)
- Finzel, S., et al. Reliability and Change in Erosion Measurements by High-resolution Peripheral Quantitative Computed Tomography in a Longitudinal Dataset of Rheumatoid Arthritis Patients. The Journal of Rheumatology. 48 (3), 348-351 (2021).
- Töpfer, D., Finzel, S., Museyko, O., Schett, G., Engelke, K. Segmentation and quantification of bone erosions in high-resolution peripheral quantitative computed tomography datasets of the metacarpophalangeal joints of patients with rheumatoid arthritis. Rheumatology (Oxford). 53 (1), 65-71 (2014).
- Peters, M., et al. The Reliability of a Semi-automated Algorithm for Detection of Cortical Interruptions in Finger Joints on High Resolution CT Compared to MicroCT. Calcified tissue international. , 1-9 (2017).
- Zhang, X., et al. Automatic 3D joint erosion detection for the diagnosis and monitoring of rheumatoid arthritis using hand HR-pQCT images. Computerized Medical Imaging and Graphics. 106, 102200 (2023).

