生成CRISPR / Cas9媒介1アレル欠失マウス胚性幹細胞でエンハンサー機能を研究するために、
Summary
Experimental validation of enhancer activity is best approached by loss-of-function analysis. Presented here is an efficient protocol that uses CRISPR/Cas9 mediated deletion to study allele-specific regulation of gene transcription in F1 ES cells which contain a hybrid genome (Mus musculus129 x Mus castaneus).
Abstract
Enhancers control cell identity by regulating tissue-specific gene expression in a position and orientation independent manner. These enhancers are often located distally from the regulated gene in intergenic regions or even within the body of another gene. The position independent nature of enhancer activity makes it difficult to match enhancers with the genes they regulate. Deletion of an enhancer region provides direct evidence for enhancer activity and is the gold standard to reveal an enhancer’s role in endogenous gene transcription. Conventional homologous recombination based deletion methods have been surpassed by recent advances in genome editing technology which enable rapid and precisely located changes to the genomes of numerous model organisms. CRISPR/Cas9 mediated genome editing can be used to manipulate the genome in many cell types and organisms rapidly and cost effectively, due to the ease with which Cas9 can be targeted to the genome by a guide RNA from a bespoke expression plasmid. Homozygous deletion of essential gene regulatory elements might lead to lethality or alter cellular phenotype whereas monoallelic deletion of transcriptional enhancers allows for the study of cis-regulation of gene expression without this confounding issue. Presented here is a protocol for CRISPR/Cas9 mediated deletion in F1 mouse embryonic stem (ES) cells (Mus musculus129 x Mus castaneus). Monoallelic deletion, screening and expression analysis is facilitated by single nucleotide polymorphisms (SNP) between the two alleles which occur on average every 125 bp in these cells.
Introduction
転写調節エレメントは、異常な遺伝子発現2に起因する疾患をもたらし得る発展1およびこれらの要素の変更時の遺伝子発現の時空間微調整のために重要です。ゲノムワイド関連解析により同定された多くの疾患関連領域は非コード領域にあり、転写エンハンサー3-4の機能を備えています。エンハンサーを特定し、彼らはしばしば、数キロベース離れて、彼らが調節する遺伝子から位置しており、組織特異的様式5-6で活性化することができるように複雑である調節する遺伝子とそれらをマッチング。エンハンサー予測は一般にヒストン修飾マーク、メディエーターコヒーシン複合体に基づいており、細胞型特異的転写因子7-10の結合されています。予測エンハンサーの検証は、ほとんどの場合、エンハンサーは、レポーター遺伝子11-12の発現を活性化するベクトルベースのアッセイを介して行われます。これらのデータは、Vを提供します推定されるエンハンサー配列の規制の可能性についてaluable情報が、それらの内因性のゲノムのコンテキストでそれらの機能を明らかにするまたはそれらが調節する遺伝子を特定するものではありません。ゲノム編集が機能喪失分析によってそれらの内因性コンテキスト内の転写調節エレメントの機能を研究するための強力なツールとして機能します。
ゲノム編集、すなわち、CRISPR / Cas9ゲノム編集システムにおける最近の進歩は、ゲノム機能の研究を促進します。 CRISPR / Cas9システムは使いやすく、多くの生物学的システムに適応可能です。 Cas9タンパク質は、ガイドRNA(gRNA)13によるゲノムの特定部位に標的化されます。 SpCas9 / gRNA複合体はprotospacer隣接モチーフ(PAM)シーケンス、NGG 14-15に5 'である必要があり、その標的ゲノム配列のためのゲノムをスキャンします。そのターゲットにgRNA、gRNAに相補的な20ヌクレオチド(nt)の配列の塩基対形成は、doublその結果SpCas9ヌクレアーゼ活性を活性化します電子鎖切断(DSB)PAM配列の上流の3bp。特異性はgRNAシード領域における完全な塩基対形成を介して達成され、6月12日は、PAMに隣接NT;逆に、シードのミスマッチが5 'は通常16-17を許容しています。導入されたDSBは、いずれかの修理非相同末端結合(NHEJ)DNA修復または相同性によって中断されることが標的部位で数塩基対の修復を(HDR)mechanisms.NHEJのDNA修復がしばしば挿入/削除を作成します(インデル)向けることができます遺伝子のオープンリーディングフレーム(ORF)。関心領域に隣接するゲノム2 gRNAs、より大きな欠失を生成するために、18〜19を使用することができます。このアプローチは、従来のエンハンサー9,18,20-22よりも大きい遺伝子座制御領域または超エンハンサーにクラスタ化転写エンハンサーの研究のために特に有用です。
1アレルの欠失は、転写のシス -regulationを研究するための貴重なモデルです。観察チャンエンハンサーの1アレル削除後の転写レベルでの電子は、両方の対立遺伝子の転写は、細胞の適応に影響を与える潜在的影響を受けているときに発生する可能性の交絡効果のない遺伝子調節におけるそのエンハンサーの役割と相関します。発現低下を評価する野生型対立遺伝子から削除を区別する能力なしには困難です。さらに、2つの対立遺伝子を識別する能力なしに各対立遺伝子に欠失を遺伝子型決定することは、特にPCRにより全体の野生型領域を増幅することは困難である1メガビット23> 10キロバイトの大きな欠失のために、困難です。 ハツカネズミのcastaneusと交差ハツカネズミ 129によって生成されたF1 ES細胞の使用は、二つの対立遺伝子は、対立遺伝子特異的PCR 18,24によって区別されることを可能にします。これらの細胞におけるハイブリッドゲノムは、対立遺伝子特異的欠失スクリーニングおよび発現分析を容易にします。平均的にSNPは、これら2つのゲノムの間のすべての125bpがあります発現および遺伝子型決定のためのプライマーの設計に柔軟性を提供する分析します。 1つのSNPの存在は、プライマーの融解温度(T m)に影響し、二つの対立遺伝子25の識別を可能にするリアルタイム定量PCR(定量PCR)増幅に特異性を標的とすることができます。さらに、プライマーの3 '末端内のミスマッチが大きく、望ましくない対立遺伝子ターゲット26の増幅を防止するプライマーから伸長するDNAポリメラーゼの能力に影響を与えます。 CRISPR / Cas9ゲノム編集システム( 図1)を使用して、1より大きいκBの対立遺伝子特異的エンハンサーの欠失、およびその後の発現解析のためにF1 ES細胞の使用は、以下の手順で説明します。
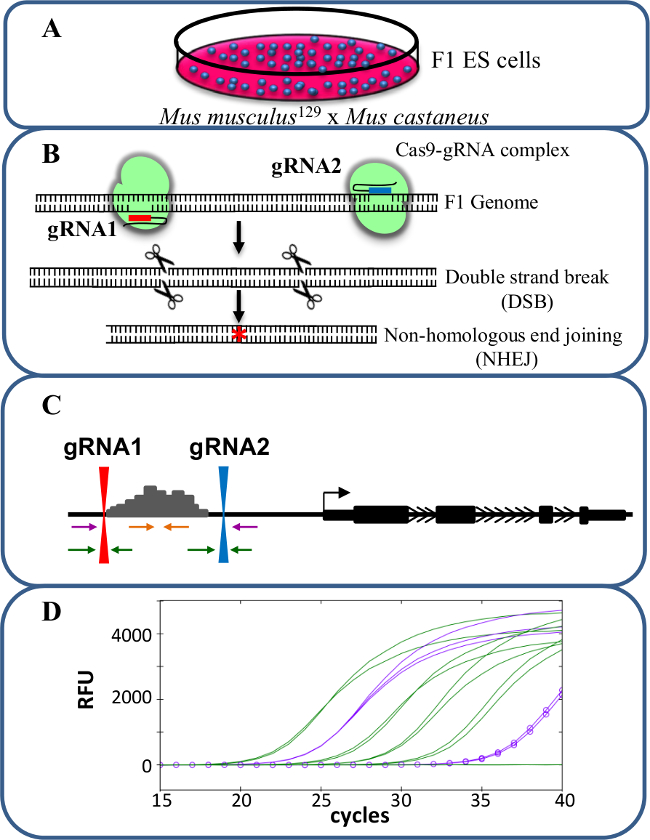
シス -regを 研究するためにCRISPR / Cas9を使用して、図1エンハンサーの削除遺伝子発現のピュレーション。 ハツカネズミ 129とハツカネズミのcastaneus間の交差によって生成された(A)F1 ES細胞は、対立遺伝子特異的欠失を可能にするために使用されます。 (B)2ガイドRNA(gRNA)は、エンハンサー領域の大Cas9媒介性欠失を誘導するために使用されます。 (C)プライマーセットは、大モノおよび二対立遺伝子の欠失を同定するために使用されます。オレンジ色のプライマーは内側プライマーである、紫色のプライマーは、外部プライマーであり、緑のプライマーはgRNAフランキングプライマーです。遺伝子発現の(D)の変更は、対立遺伝子特異的定量PCRを使用して監視しています。 RFUは相対蛍光単位を示している。 この図の拡大版をご覧になるにはこちらをクリックしてください。
Protocol
Representative Results
Discussion
CRISPR / Cas9媒介ゲノム編集技術は、ゲノム改変のための、簡単で迅速かつ安価な方法を提供します。機能的エンハンサー特徴付けのために1アレルエンハンサーの削除を生成し、分析するために、ここで具体的な方法は、F1マウス細胞におけるSNPを利用しています。このタイプのアプローチの利点は次のとおりです。1)1アレルエンハンサー欠失は重要なエンハンサーは、 すなわち 、両?…
Declarações
The authors have nothing to disclose.
Acknowledgements
We would like to thank all the members of the Mitchell lab for helpful discussions. This work was supported by the Canadian Institutes of Health Research, the Canada Foundation for Innovation and the Ontario Ministry of Research and Innovation (operating and infrastructure grants held by JAM).
Materials
| Phusion High-Fidelity DNA Polymerase | NEB | M0530S | high fidelity DNA polymerase used in gRNA assembly |
| Gibson Assembly Master Mix | NEB | E2611L | |
| gRNA_Cloning Vector | Addgene | 41824 | A target sequence is cloned into this vector to create the gRNA plasmid |
| pCas9_GFP | Addgene | 44719 | Codon-optimized SpCas9 and EGFP co-expression plasmid |
| AflII | NEB | R0520S | |
| EcoRI | NEB | R3101S | |
| Neon Transfection System 100 µL Kit | Life Technologies | MPK10096 | Microporator transfection technology |
| prepGEM | ZyGEM | PT10500 | genomic DNA extraction reagent |
| Nucleo Spin Gel & PCR Clean-up | Macherey-Nagel | 740609.5 | |
| High-Speed Plasmid Mini Kit | Geneaid | PD300 | |
| Maxi Plasmid Kit Endotoxin Free | Geneaid | PME25 | |
| SYBR select mix for CFX | Life Technologies | 4472942 | qPCR reagent |
| iScript cDNA synthesis kit | Bio-rad | 170-8891 | Reverse transcription reagent |
| 0.25% Trypsin with EDTA | Life Technologies | 25200072 | |
| PBS without Ca/Mg2+ | Sigma | D8537 | |
| 0.5M EDTA | Bioshop | EDT111.500 | |
| HBSS | Life Technologies | 14175095 | |
| 1M HEPES | Life Technologies | 13630080 | |
| BSA fraction V (7.5%) | Life Technologies | 15260037 | |
| Max Efficiency DH5α competent cells | Invitrogen | 18258012 | |
| FBS | ES cell qualified | FBS is subjected to a prior testing in mouse ES cells for pluripotency | |
| DMSO | Sigma | D2650 | |
| Glutamax | Invitrogen | 35050 | |
| DMEM | Life Technologies | 11960069 | |
| Pencillin/Streptomycin | Invitrogen | 15140 | |
| Sodium pyruvate | Invitrogen | 11360 | |
| Non-essential aminoacid | Invitrogen | 11140 | |
| β-mercaptoethanol | Sigma | M7522 | |
| 96-well plate | Sarstedt | 83.3924 | |
| Sealing tape | Sarstedt | 95.1994 | |
| CoolCell LX | Biocision | BCS-405 | alcohol-free cell freezing container |
| CHIR99021 | Biovision | 1748-5 | Inhibitor for F1 ES cell culture |
| PD0325901 | Invivogen | inh-pd32 | Inhibitor for F1 ES cell culture |
| LIF | Chemicon | ESG1107 | Inhibitor for F1 ES cell culture |
Referências
- Sagai, T., Hosoya, M., Mizushina, Y., Tamura, M., Shiroishi, T. Elimination of a long-range cis-regulatory module causes complete loss of limb-specific Shh expression and truncation of the mouse limb. Development. 132 (4), 797-803 (2005).
- Kleinjan, D. A., Lettice, L. A. Long-range gene control and genetic disease. Adv Genet. 61, 339-388 (2008).
- Visel, A., Rubin, E. M., Pennacchio, L. A. Genomic views of distant-acting enhancers. Nature. 461 (7261), 199-205 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Shen, Y., et al. A map of the cis-regulatory sequences in the mouse genome. Nature. 488 (7409), 116-120 (2012).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316 (5830), 1497-1502 (2007).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147 (6), 1408-1419 (2011).
- Whyte, W. A., et al. Master transcription factors and mediator establish super-enhancers at key cell identity genes. Cell. 153 (2), 307-319 (2013).
- Chen, C. Y., Morris, Q., Mitchell, J. A. Enhancer identification in mouse embryonic stem cells using integrative modeling of chromatin and genomic features. BMC Genomics. 13 (1), 152 (2012).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Melnikov, A., et al. Systematic dissection and optimization of inducible enhancers in human cells using a massively parallel reporter assay. Nat Biotechnol. 30 (3), 271-277 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Cho, S. W., et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 24 (1), 132-141 (2014).
- Zhou, H. Y., et al. A Sox2 distal enhancer cluster regulates embryonic stem cell differentiation potential. Genes Dev. 28 (24), 2699-2711 (2014).
- Fujii, W., Kawasaki, K., Sugiura, K., Naito, K. Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease. Nucleic Acids Res. 41 (20), e187 (2013).
- Tuan, D. Y., Solomon, W. B., London, I. M., Lee, D. P. An erythroid-specific, developmental-stage-independent enhancer far upstream of the human ‘beta-like globin’ genes. Proc Natl Acad Sci U S A. 86 (8), 2554-2558 (1989).
- Amano, T., et al. Chromosomal dynamics at the Shh locus: limb bud-specific differential regulation of competence and active transcription. Dev Cell. 16 (1), 47-57 (2009).
- Li, Y., et al. CRISPR reveals a distal super-enhancer required for Sox2 expression in mouse embryonic stem cells. PLoS One. 9 (12), e114485 (2014).
- Canver, M. C., et al. Characterization of genomic deletion efficiency mediated by clustered regularly interspaced palindromic repeats (CRISPR)/Cas9 nuclease system in mammalian cells. J Biol Chem. 289 (31), 21312-21324 (2014).
- Mlynarczyk-Evans, S., et al. X chromosomes alternate between two states prior to random X-inactivation. PLoS Biol. 4 (6), e159 (2006).
- Lefever, S., Pattyn, F., Hellemans, J., Vandesompele, J. Single-nucleotide polymorphisms and other mismatches reduce performance of quantitative PCR assays. Clin Chem. 59 (10), 1470-1480 (2013).
- Huang, M. M., Arnheim, N., Goodman, M. F. Extension of base mispairs by Taq DNA polymerase: implications for single nucleotide discrimination in PCR. Nucleic Acids Res. 20 (17), 4567-4573 (1992).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Yalcin, B., et al. Sequence-based characterization of structural variation in the mouse genome. Nature. 477 (7364), 326-329 (2011).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Gibson, D. G., Smith, H. O., Hutchison, C. A., Venter, J. C., Merryman, C. Chemical synthesis of the mouse mitochondrial genome. Nat Methods. 7 (11), 901-903 (2010).
- Ding, Q., et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell. 12 (4), 393-394 (2013).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of specific cell population by fluorescence activated cell sorting (FACS). J Vis Exp. (41), (2010).
- Forlenza, M., Kaiser, T., Savelkoul, H. F., Wiegertjes, G. F. The use of real-time quantitative PCR for the analysis of cytokine mRNA levels. Methods Mol Biol. 820, 7-23 (2012).
- Wu, J. H., Hong, P. Y., Liu, W. T. Quantitative effects of position and type of single mismatch on single base primer extension. J Microbiol Methods. 77 (3), 267-275 (2009).
- Sanyal, A., Lajoie, B. R., Jain, G., Dekker, J. The long-range interaction landscape of gene promoters. Nature. 489 (7414), 109-113 (2012).

