Sublimation von DAN Matrix für die Erkennung und Visualisierung von Gangliosiden in Rattenhirngewebe für die MALDI-Imaging-Massenspektrometrie
Summary
A protocol for the sublimation of DAN matrix onto rat brain tissue for the detection of gangliosides using MALDI Imaging Mass Spectrometry is presented.
Abstract
Probenvorbereitung ist der Schlüssel für eine optimale Detektion und Visualisierung von Analyten in Matrix-unterstützte Laser-Desorption / Ionisation (MALDI) Imaging-Massenspektrometrie (IMS) Experimenten. Bestimmen des geeigneten Protokolls während der Probenvorbereitungsprozess folgen kann schwierig sein, da jede Stufe optimiert werden, müssen mit den einzigartigen Eigenschaften der Analyten von Interesse entsprechen. Dieser Prozess beinhaltet nicht nur eine kompatible Matrix zu finden, die die Moleküle von Interesse effizient desorbieren und ionisieren kann, sondern auch die entsprechende Matrix Abscheidungstechnik ausgewählt wird. Beispielsweise eine nasse Matrix Abscheidungstechnik, die eine Matrix in einem Lösungsmittel aufgelöst ist, beinhaltet, superior zur Desorption der meisten Proteine und Peptide, während trockene Matrix Abscheidungstechniken besonders wirksam sind für die Ionisation von Lipiden. Sublimation hat sich als hocheffizientes Verfahren zur Trockenmatrixablagerung zum Nachweis von Lipiden in Gewebe durch MALDI IMS aufgrund der homogenei berichtetty der Matrix Kristallablagerung und minimale Analyten Delokalisierung im Vergleich zu vielen Nassabscheidungsverfahren 1, 2. Im weitesten Sinne handelt es mit den Proben gegen eine kalte Oberfläche gepresst in einer vakuumdichten Kammer, die eine Probe und pulverisierten Matrix platzieren. Die Vorrichtung wird dann in ein erwärmtes Bad (Sand oder Öl) gesenkt, was zu Sublimation des pulverförmigen Matrix auf der Probenoberfläche gekühlt Gewebe. Hier beschreiben wir ein Sublimations-Protokoll 1,5-Diaminonaphthalin (DAN) -Matrix für die Detektion und Darstellung von Gangliosiden in dem Rattenhirn unter Verwendung von MALDI IMS.
Introduction
Matrix-unterstützte Laser-Desorptions / Ionisations (MALDI) Imaging-Massenspektrometrie (IMS) entwickelt sich zu einem sehr begehrt Technik zur Visualisierung der räumlichen Verteilung von Lipiden, Peptiden und Proteinen in intakten Probenoberflächen. MALDI IMS wurde zuvor als analytische Technik für die vorgereinigten Analyten bekannt, aber in den letzten Jahren hat es sich in vielen anderen Disziplinen wegen der Fähigkeit, die Genauigkeit der Massenspektrometrie zu kombinieren mit einer hohen Auflösung optisch / anatomischer Referenzpunkte ohne das die Aufmerksamkeit müssen für jede externe Kennzeichnung. Da die wissenschaftliche Pool von Forschern diese Technik verwendet wird, weiter zu wachsen ist Bedarf an standardisierten, einfach zu folgen Protokolle erhöht in der Entwicklung und Optimierung von IMS Experimente zu unterstützen. Ganglioside, eine Gruppe von Membranlipiden sehr reichlich vorhanden in dem Zentralnervensystem, sind ideal für MALDI IMS Experimente wie ihre Lage in der Membran eingebettet ist, stellt sicher, speziES unmöglich mit konventionellen Immuno-Kennzeichnung zu erkennen. Darüber hinaus haben wir gezeigt, MALDI IMS verwenden, dass diese Lipide, die als Modulatoren von Zellsignalen funktionieren, unter anderem einzigartige anatomische Verteilungsmuster im gesunden Gehirn von Nagetieren, die 3 nach einer Hirnschädigung verändert, 4, 5. Ganglioside werden mit einer höheren Massenbereich im Vergleich zu den meisten Lipidspezies, und sind daher am meisten geeignet für die MALDI Bildgebungs Plattform befinden.
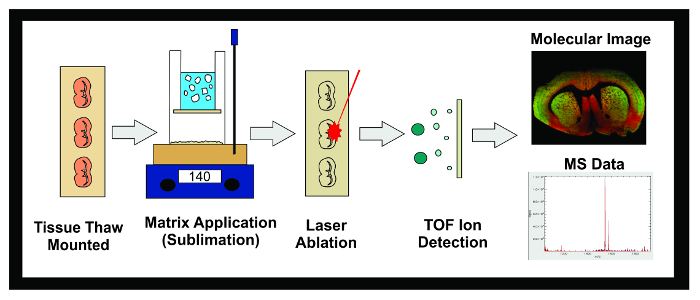
Abbildung 1: Workflow von MALDI IMS Experiment. Schematische Darstellung des allgemeinen Arbeitsablauf eines MALDI IMS Experiment unter Verwendung von Sublimation. Gewebe bei -80 ° C eingefroren ist in einem Kryostaten geschnitten und 10 & mgr; m Abschnitte sind thaw auf leitfähigen ITO Objektträger aufgebracht. Der Objektträger wird dann in einem Exsikkator bis zur Sublimation platziert. slides in die Sublimation Vorrichtung eingeführt und eine gleichmäßige Schicht der Matrix ist auf der Gewebeprobenoberfläche aufgetragen. Die Proben werden über Nacht in einem -20 ° C Gefrierschrank eingefroren dann in einem Exsikkator für 10 Minuten gestellt. Sobald Normen angewandt wurden, werden die Proben in das MALDI Instrument eingeführt, wo ein Laser über das Gewebe gerichtet ist, verursacht desorbierten Moleküle in der Matrix zu ionisieren. Die Ionen wandern ein Flugrohr nach unten und getrennt auf der Grundlage ihrer Masse (time-of-flight / TOF), bis sie den Detektor erreichen. Die Informationen über die Ionenfluss von Analyten in einem vorbestimmten Masse-zu-Ladung (m / z) -Bereich wird sowohl als Molekularbild und Massenspektrum angezeigt. Diese Daten können verwendet werden, um sowohl zu visualisieren und die Ionenfluss des Analyten von Interesse innerhalb des abgebildeten Gewebes zu quantifizieren. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Probenvorbereitungfür MALDI IMS ist sehr variabel, da jeder Schritt des Verfahrens an den Analyten von Interesse angepasst werden müssen. Das bestimmende Merkmal der MALDI-basierten Experimenten ist die Verwendung einer Matrix-Beschichtung auf die Probenoberfläche vor der Analyse abgeschieden. Zusätzlich zu der Rolle zu absorbieren und Strahlungsenergie von dem Laser während des Ablationsprozesses zu übertragen, dient die Matrix auch verschiedene Analyten aus der Probe zu isolieren, wodurch die Analyse der Verbindungen von Interesse zu erleichtern 6, 7. Homogenes Aufbringen der Matrix auf die Probenoberfläche ist der wichtigste Schritt bei der Probenvorbereitung. Eine falsche Matrix Abscheidung kann zu großen heterogenen Matrix Kristallformationen führen und die Entwicklung von Artefakten, niedrige Ionensignal und schlechte Reproduzierbarkeit 7.
Aufgrund der Affinität bestimmter Matrices spezifischen Analyten zu isolieren, kann die Art der Matrix für ein Experiment ausgewähltdeutlich das Ergebnis zu verändern. Die Matrizen verwendet für die Bildgebung von Proteinen und Peptiden unterscheiden sich oft von denen, für die Bildgebung verwendeten Lipide und das Verfahren wird weiter durch die Notwendigkeit für zusätzliche Verfahren, wie Waschen und Rehydratation Schritte, um kompliziert erfolgreich Signale von Gewebe erkennen. Obwohl Waschschritte zur Verbesserung der Lipidsignale existieren 8, sind sie keine Voraussetzung für die Detektion der meisten Lipidspezies. Wenn eine Matrix für eine Lipidbildgebungsexperiment auswählt, ist es wichtig, dass die Polarität des Lipids von Interesse, da dies zu prüfen, wird der Bereich von geeigneten Matrizen renzen. Enthalten beispielsweise Ganglioside Sialinsäurereste, die sie insgesamt eine negative Polarität geben. Es gibt eine Reihe von Matrizen, die effektiv zu desorbieren und zu ionisieren Ganglioside aus Gewebe; jedoch Faktoren wie Matrix abgeleiteten Peaks im Spektrum und Stabilität der Matrix unter Vakuum muß in Betracht gezogen werden. 1,5-Diaminonaphthalin (DAN) Matrix ist ausreichend stabil , um unter Vakuumbedingungen Instrument für die meisten Abbildungsanwendungen und hat einen hohen Grad an Empfindlichkeit für Lipid Desorption gezeigt und kann sowohl in 2 positive und negative Ionenarten für die Analyse von Lipiden verwendet werden. DAN Matrix, wenn im Vergleich zu anderen negativen Lipid Affinitätsmatrizen wie Dihydroxybenzoesäure (DHB), 9-Aminoacridin (9-AA) und 5-Chloro-2-Mercaptobenzothiazol (CMBT), konnte am effizientesten Gangliosiden aus Rattenhirn desorbieren Gewebe im negativen Ionenmodus (Manuskript in Vorbereitung).
die entsprechende Methode der Matrixablagerung Auswahl von gleicher Bedeutung ist selbst die Matrix zu wählen. Nassmatrixabscheidungsverfahren, wobei die feste Matrix in einem organischen Lösungsmittel gelöst wird, und durch pneumatische oder automatisierten Sprüher oder spotters abgeschieden sind besonders wirksam für die Desorption von Proteinen und Peptiden als die Flüssigkeit die Probe für zusätzliche zuzulassen permeiertction von Verbindungen und Co-Kristallisation mit der Matrix. Obwohl diese Techniken auch für die Lipid – Anwendungen verwendet werden kann, Analyt Delokalisierung und ungleichmäßige Matrix Kristallformationen sind häufige Vorkommen aufgrund der hohen Menge und Löslichkeit von Lipiden in Lösungsmitteln, insbesondere in Gewebe 2, 9. Weil Lipiden leicht aus Gewebe ionisiert werden, trockene Matrix Abscheidungstechniken, wie Sublimation, bieten eine einfache, kostengünstige Alternative zu Sprüher während viele der Nachteil dieser Techniken zu umgehen. Der Erfolg der Sublimierung in MALDI IMS Experimente auf Merkmale wie mikrokristalline Matrix Morphologie zugeschrieben , die für Matrix-Analyt – Bindung, erhöhte Matrix Reinheit der Oberfläche erhöht, und homogene Matrix Ablagerung zu einer erhöhten Reproduzierbarkeit im Vergleich zu nassen Matrixtechniken , 1, 10.
Sublimation invoLves eine pulverisierte Matrix unter Vakuum unmittelbar unterhalb einer gekühlten Probenoberfläche Heizgas-Phasenübergang des pulverförmigen Matrix, gefolgt von der Abscheidung auf die Gewebeprobenoberfläche in Feststoff zu ergeben. Während der Sublimation, können Matrixablagerung durch Variieren Faktoren wie Zeit, Temperatur und Druck zu liefern hoch reproduzierbare Ergebnisse gesteuert werden. Ein einzelnes Sublimation Experiment kann überall nehmen von 5 bis 20 min in Abhängigkeit von der Art der Matrix ausgewählt wird, die mehrmals vor der Entsorgung wieder eingesetzt werden können. Die Vorrichtung kann zu einem Bruchteil des Preises von automatischen Sprüher im Handel erworben werden und ist leicht auseinander zur Reinigung und Wartung entnommen. Die niedrigen Kosten und die relative Einfachheit dieser Matrix-Abscheidungstechnik machen es für die Forscher ideale Beginn oder die Erweiterung auf Lipid-Imaging-Anwendungen in MALDI IMS. Auch wenn die Informations Protokolle für die Sublimation von Geweben für IMS haben Detaillierung 11 berichtet worden, einige standardisierte Protokolle existieren which konzentrieren sich auf die Basis-Workflow beteiligt mit einer Sublimation Experiment Durchführung für die Bildgebung mit hoher Masse Lipide im Negativ-Ionen-Modus, so dass es schwierig ist, die Technik ohne aufwendige Versuch und Irrtum zu etablieren. Im Folgenden ist ein experimentelles Protokoll, diese Lücke für die Sublimation von DAN Matrix auf Rattenhirnschnitten für hochauflösende Bildgebung und Detektion von Gangliosiden zu füllen Ziel.
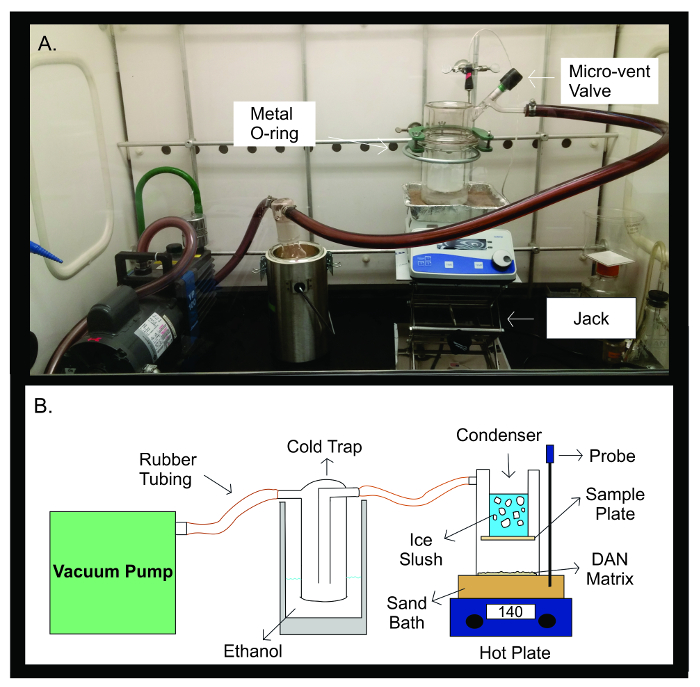
Abbildung 2: -Sublimationsapparatur. Fotografie (A) und schematische Darstellung (B) der -Sublimationsapparatur. Die Vakuumpumpe wird durch Gummischlauch in eine Kühlfalle, gefüllt mit 300 ml Ethanol verbunden. Die Kühlfalle wird dann durch Gummischlauch zur Sublimation Vorrichtung verbunden. Die Vorrichtung besteht aus zwei separaten Teilen von Glaswaren, die zusammen mit einem Metall-U-joint abgedichtet sind. Die obere Hälfte des Sublimatorschiffchenenthält den Kondensator, der mit Eisbrei gefüllt ist. Die Probenplatte wird auf den Boden des Kondensators abgeklebt, im Inneren des abgedichteten Glasapparatur. Die untere Hälfte der -Sublimationsapparatur enthält die DAN-Matrix gleichmäßig verteilt die Probenplatte gegenüber. Während der Sublimation wird die Glasapparatur auf einem Sandbad mit einer heißen Platte erhitzt auf 140 ° C gebracht direkt darunter. Der Temperaturfühler trägt, durch Rückkopplung des Sandbad Temperatur im Vergleich zu der voreingestellten Temperatur für das Experiment eine stabile Temperatur während der Sublimation Experiments aufrechtzuerhalten. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Protocol
Representative Results
Discussion
Diese Arbeit Details eines standardisierten Protokolls von Matrix Sublimation auf das Gewebe für die Detektion von negativ geladenen Lipiden, wie Ganglioside, in MALDI IMS Experimenten. Probenvorbereitung für MALDI IMS ist sehr variabel und muss angepasst werden, um die einzigartigen Eigenschaften des Analyten von Interesse zu passen. Die Anwendung der Matrix auf die Gewebeprobenoberfläche ist ein wichtiger Aspekt des Probenherstellungsverfahrens im Hinblick auf die Qualität der Ergebnisse in IMS. Besondere Vorsicht…
Declarações
The authors have nothing to disclose.
Acknowledgements
We would like to acknowledge the technical assistance of Kristina Jurcic in the University of Western Ontario MALDI MS facility, as well as the National Sciences and Engineering Council (NSERC) for funding this work. The authors would also like to acknowledge the Caprioli group (Vanderbilt University, TN) and Chaurand group (Université de Montreal, QC) for their advice in optimizing the sublimation technique presented in this manuscript.
Materials
| Sublimator | Chemglass Life Sciences | CG3038-01 | |
| 1,5- Diaminonapthalene (DAN) matrix | Sigma-Aldrich | D21200 | 100 G |
| Cryostat | Thermo-Fisher Scientific | CryoStar NX50 | |
| Hot plate with temperature feedback | Thermo-Fisher Scientific | HP88857290 | Isotemp ADVD 7×7 HP 100-120v |
| Stainless Steel Jack | Thermo-Fisher Scientific | 2216479 | 10×10 |
| Cold Trap | Custom built on site | ||
| Vacuum Pump | Franklin Electric | 1102180403 | Savant VP100 Two Stage |
| Indium-tin-oxide (ITO) Slides | Hudson Surface Technology | PSI 1111000 | type II, 1.1mm/25 each |
| MALDI TOF/TOF 5800 Instrument | AB Sciex | ||
| Desiccator | Sigma-Aldrich | D2797 | tabletop desiccator |
Referências
- Hankin, J. A., Barkley, R. M., Murphy, R. C. Sublimation as a method of matrix application for mass spectrometric imaging. J. Am. Soc. Mass Spectrom. 18, 1646-1652 (2007).
- Thomas, A., Charbonneau, J. L., Fournaise, E., Chaurand, P. Sublimation of new matrix candidates for high spatial resolution imaging mass spectrometry of lipids: enhanced information in both positive and negative polarities after 1,5-diaminonapthalene deposition. Anal. Chem. 84, 2048-2054 (2012).
- Caughlin, S., et al. Increased Expression of Simple Ganglioside Species GM2 and GM3 Detected by MALDI Imaging Mass Spectrometry in a Combined Rat Model of Aβ Toxicity and Stroke. PLoS ONE. 10, 0130364 (2015).
- Weishaupt, N., Caughlin, S., Yeung, K., Whitehead, S. Differential Anatomical Expression of Ganglioside GM1 Species Containing d18:1 or d20:1 Sphingosine Detected by MALDI Imaging Mass Spectrometry in Mature Rat Brain. Front Neuroanatomy. 9 (155), (2015).
- Whitehead, S., et al. Imaging Mass Spectrometry Detection of Gangliosides Species in the Mouse Brain following Transient Focal Cerebral Ischemia and Long-Term Recovery. PLoS ONE. 6 (6), 20808 (2011).
- Fuchs, B., Süß, R., Schiller, J. An update of MALDI-TOF mass spectrometry in lipid research. Progress in Lipid Research. 49, 450-475 (2010).
- Barceló-Coblijn, G., Fernández, J. A. Mass spectrometry coupled to imaging techniques: the better the view the greater the challenge. Front Physiol. 6 (3), 1-5 (2015).
- Angel, P. M., Spraggins, J. M., Baldwin, H. S., Caprioli, R. Enhanced sensitivity for high spatial resolution lipid analysis by negative ion mode matrix assisted laser desorption ionization imaging mass spectrometry. Anal. Chem. 84, 1557-1564 (2012).
- Murphy, R. C., Hankin, J. A., Barkley, R. M., Zemski Berry, K. A. MALDI imaging of lipids after matrix sublimation/deposition. Biochim. Biophys. Acta. 1811, 970-975 (2011).
- Jaskolla, T. W., Karas, M., Roth, U., Steinert, K. Comparison between vacuum sublimed matrices and conventional dried droplet preparation in MALDI-TOF mass spectrometry. J. Am. Soc. Mass Spectrom. 20, 1104-1115 (2009).
- O’Rourke, M. B., Raymond, B. B., Djordjevic, S. P., Padula, M. P. A versatile cost-effective method for the analysis of fresh frozen tissue sections via matrix-assisted laser desorption/ionisation imaging mass spectrometry. Rapid Commun. Mass Spectrom. 29, 637-644 (2015).
- Patterson, N. H., Thomas, A., Chaurand, P. Monitoring time-dependent degradation of phospholipids in sectioned tissues by MALDI imaging mass spectrometry. J Mass Spectrom. 49, 622-627 (2014).
- Cheng, H., Sun, G., Yang, K., Gross, R. W., Han, X. Selective desorption/ionization of sulfatides by MALDI-MS facilitated using 9-aminoacridine as matrix. J. Lipid Res. 51, 1599-1609 (2010).
- Puolitaival, S. M., Burnum, K. E., Cornett, D. S., Caprioli, R. M. Solvent-free matrix dry-coating for MALDI imaging of phospholipids. J. Am. Soc. Mass Spectrom. 19, 882-886 (2008).
- Chaurand, P., Cornett, D., Angel, P., Caprioli, R. From Whole-body Sections Down to Cellular Level, Multiscale Imaging of Phospholipids by MALDI Mass Spectrometry. Mol Cell Proteomics. 10, (2011).
- Grove, K. J., Frappier, S. L., Caprioli, R. M. Matrix pre-coated MALDI MS targets for small molecule imaging in tissues. J. Am. Soc. Mass Spectrom. 22, 192-195 (2011).
- Yang, J., Caprioli, R. M. Matrix precoated targets for direct lipid analysis and imaging of tissue. Anal. Chem. 85, 2907-2912 (2013).
- Gemperline, E., Rawson, S., Li, L. Optimization and comparison of multiple MALDI matrix application methods for small molecule mass spectrometric imaging. Anal. Chem. 86, 10030-10035 (2014).

