CRISPR/Cas9 के लिए Integrase-कमी Lentiviral वैक्टर के उत्पादन के लिए एक प्रोटोकॉल-विभाजन कोशिकाओं में मध्यस्थता जीन नॉकआउट
Summary
हम कोशिकाओं को CRISPR/Cas9 पहुंचाने के लिए वाहनों के रूप में integrase की कमी lentiviral वैक्टर (IDLVs) की उत्पादन रणनीति का वर्णन । कोशिकाओं में त्वरित और मजबूत जीन संपादन मध्यस्थता करने की क्षमता के साथ, IDLVs integrase सक्षम वैक्टर की तुलना में जीन वितरण के लिए एक सुरक्षित और समान रूप से प्रभावी वेक्टर मंच मौजूद.
Abstract
Lentiviral वैक्टर जीन-संपादन घटकों के लिए उनकी क्षमता के कारण छुरा transducing कोशिकाओं की एक व्यापक रेंज के लिए और जीन अभिव्यक्ति के उच्च स्तर मध्यस्थता कोशिकाओं को देने के लिए एक आदर्श विकल्प हैं । हालांकि, उनके मेजबान सेल जीनोम में एकीकृत करने की क्षमता mutagenicity के जोखिम को बढ़ाता है और इस तरह सुरक्षा चिंताओं को उठाती है और नैदानिक सेटिंग्स में उनके उपयोग की सीमा । इसके अलावा, इन एकीकरण-सक्षम lentiviral वैक्टर (ICLVs) द्वारा दिया जीन-संपादन घटकों की लगातार अभिव्यक्ति संकीर्ण जीन लक्ष्यीकरण की संभावना बढ़ जाती है । एक विकल्प के रूप में, integrase की कमी lentiviral वैक्टर (IDLVs) की एक नई पीढ़ी विकसित किया गया है कि इन चिंताओं के कई पते । यहां CRISPR के लिए एक नया और बेहतर IDLV मंच के उत्पादन प्रोटोकॉल-मध्यस्थता जीन संपादन और सूची में शामिल कदम शुद्धि और ऐसे वैक्टर की एकाग्रता में वर्णित है और उनके transduction और जीन संपादन क्षमता का उपयोग कर HEK-293T कक्षों का प्रदर्शन किया गया । इस प्रोटोकॉल को आसानी से स्केलेबल है और उच्च titer IDLVs है कि इन विट्रो में और vivo मेंtransducing कोशिकाओं में सक्षम है उत्पंन करने के लिए इस्तेमाल किया जा सकता है । इसके अलावा, इस प्रोटोकॉल आसानी से ICLVs के उत्पादन के लिए अनुकूलित किया जा सकता है ।
Introduction
सटीक जीन संपादन प्रमुख जैव चिकित्सा अग्रिमों कि उपंयास रणनीतियों के विकास के आनुवंशिक रोगों से निपटने के लिए शामिल की आधारशिला रूपों । जीन संपादन प्रौद्योगिकियों के सबसे आगे में सीlustered आरegularly के उपयोग पर निर्भर विधि है-interspaced sप्र॰ palindromic repeats (CRISPR)/Cas9 प्रणाली है कि शुरू में पहचान की गई थी वायरल आनुवंशिक सामग्री के आक्रमण के खिलाफ बैक्टीरियल प्रतिरक्षा के एक घटक के रूप में (संदर्भ1,2) में समीक्षा की । इस तरह के जस्ता के रूप में अंय जीन संपादन उपकरण, पर CRISPR/Cas9 प्रणाली का एक प्रमुख लाभ फिंगर nucleases (ZFNs) और प्रतिलेखन उत्प्रेरक-जैसे प्रभाव nucleases (TALENs) (संदर्भ में समीक्षित3), प्लाज्मिड डिजाइन के सापेक्ष सादगी है और CRISPR घटकों के निर्माण-एक विशेषता है कि एक बहुत व्यापक अनुसंधान समुदाय के लिए कुछ विशेष प्रयोगशालाओं से जीन संपादन के विस्तार संचालित है । इसके अतिरिक्त, CRISPR/Cas9 प्रोग्रामिंग की सादगी और मल्टीप्लेक्सीय लक्ष्य मांयता के लिए इसकी क्षमता को और अधिक प्रभावी और आसान करने के लिए उपयोग प्रौद्योगिकी के रूप में अपनी लोकप्रियता ईंधन है । विभिंन शोधकर्ताओं के लिए उपलब्ध तरीकों के अलावा ऐसी जीन कोशिकाओं को संपादन घटक देने के लिए, वायरल वैक्टर अब तक का सबसे लोकप्रिय और कुशल प्रणाली रहते हैं ।
Lentiviral वैक्टर (LVs) विविध अनुप्रयोगों4,5,6,7के लिए vivo में CRISPR/Cas9 प्रणाली के घटकों को वितरित करने के लिए पसंद के वाहन के रूप में उभरा है । कई प्रमुख विशेषताएं LVs इस प्रक्रिया के लिए एक लोकप्रिय विकल्प बनाने के लिए दोनों विभाजन और गैर विभाजित कोशिकाओं को संक्रमित करने की क्षमता सहित, कम immunogenicity, और ंयूनतम सेलुलर विषाक्तता (संदर्भ में समीक्षा की8) । नतीजतन, एल. वी. मध्यस्थता जीन चिकित्सा संक्रामक रोगों के उपचार में कार्यरत किया गया है, जैसे एचआईवी-1, एचबीवी, और एचएसवी-1, और साथ ही मानव वंशानुगत रोगों अंतर्निहित दोषों के सुधार में, जैसे सिस्टिक फाइब्रोसिस और नव संवहनी धब्बेदार अध… 4 , 5 , 7 , 9 , 10 , 11. इसके अलावा, LVs प्रभावी रूप से अलग जीनोमिक loci पर मल्टीप्लेक्स जीन संपादन प्रदर्शन के लिए एक एकल वेक्टर प्रणाली12का उपयोग कर संशोधित किया गया है ।
हालांकि, LVs के अंतर्निहित संपत्ति मेजबान जीनोम में एकीकृत करने के लिए mutagenic जा सकता है और अक्सर transgene प्रसव के वाहनों के रूप में उनकी उपयोगिता बाधाएं, नैदानिक सेटिंग्स में विशेष रूप से । इसके अलावा, के बाद से छुरा एकीकृत LVs बनाए रखने के उच्च स्तर पर अपने transgenes एक्सप्रेस, इस प्रणाली के बीमार जैसे CRISPR/Cas9 के रूप में जीन संपादन घटकों के वितरण के लिए अनुकूल है; Cas9 के एक्सप्रेस-गाइड आरएनए (gRNA), और ऐसे ZFNs के रूप में इसी तरह के प्रोटीन, बंद लक्ष्य प्रभाव है, जो अवांछनीय उत्परिवर्तनों13,14,15,16 शामिल के ऊंचा स्तर के साथ जुड़े रहे हैं , 17 और संभावित cytotoxicity18में वृद्धि कर सकते हैं । इसलिए, ंयूनतम बंद लक्ष्य प्रभाव के साथ सटीक जीन संपादन को प्राप्त करने के लिए, यह प्रणाली है कि जीन संपादन घटकों के क्षणिक अभिव्यक्ति के लिए अनुमति डिजाइन करने के लिए आवश्यक है ।
हाल के वर्षों में, कई प्रसव प्लेटफार्मों के लिए विकसित किया गया है क्षणिक एक्सप्रेस CRISPR/Cas9 कोशिकाओं में16,19,20,21 (संदर्भ में समीक्षा की22) । इन तरीकों है कि सीधे कोशिकाओं में उपयुक्त गाइड RNAs के साथ शुद्ध Cas9 शुरू करने पर भरोसा शामिल है, जो दिखाया गया था और अधिक प्रभावी जीन प्लाज्मिड की तुलना में संपादन-अभिकर्मक की मध्यस्थता के लिए16. अध्ययनों से पता चला है कि ribonucleoprotein (RNP) गाइड आरएनए से मिलकर परिसरों/Cas9 कणों तेजी से अपने लक्ष्य पर डीएनए दरार मध्यस्थता के बाद बदल रहे हैं, सुझाव है कि इन घटकों की अल्पकालिक अभिव्यक्ति को प्राप्त करने के लिए पर्याप्त है मजबूत जीन संपादन16. adeno-जुड़े वायरल वैक्टर (AAVs) के रूप में गर्भ धारण, गैर एकीकृत वायरल वेक्टर प्लेटफार्मों कोशिकाओं को जीन संपादन मशीनरी देने के लिए एक व्यवहार्य विकल्प प्रदान कर सकते हैं । दुर्भाग्यवश, AAV capsids LVs (< 5kb) की तुलना में काफी कम पैकेजिंग क्षमता रखते हैं, जो किसी एकल सदिश के भीतर बहु-घटक CRISPR टूलकिट को पैकेज करने की क्षमता को गंभीर रूप से बाधित करती है (संदर्भ8में) । यह ध्यान देने योग्य है कि यौगिकों कि हिस्टोन deacetylases (जैसे, सोडियम butyrate23) को बाधित या कोशिका चक्र में बाधा के अलावा (जैसे, कैफीन24) lentiviral titers बढ़ाने के लिए दिखाया गया है । हाल ही में प्रगति के बावजूद, क्षणिक अभिव्यक्ति प्रणालियों अब तक विकसित अब भी कम उत्पादन क्षमता है, जो कम वायरल titers के लिए सीसा, और के माध्यम से उत्पंन वायरस की कम transduction क्षमता के रूप में कई कमियों, द्वारा बाधित कर रहे है इस तरह के दृष्टिकोण25।
Integrase की कमी lentiviral वैक्टर (IDLVs) जीन प्रसव के वाहनों के विकास में एक प्रमुख उंनति का प्रतिनिधित्व करते हैं, के रूप में वे कोशिकाओं में AAV की तरह episomal रखरखाव के अतिरिक्त लाभ के साथ LVs की पैकेजिंग क्षमता गठबंधन । इन सुविधाओं IDLVs काफी हद तक संभावित genotoxic तत्वों और एकीकरण-मध्यस्थता mutagenicity की तुलना लगातार व्यक्त वैक्टर घालमेल के साथ जुड़े प्रमुख मुद्दों को दरकिनार मदद करते हैं । यह पहले से प्रदर्शित किया गया है कि IDLVs सफलतापूर्वक episomal जीन अभिव्यक्ति26,27बढ़ाने के लिए संशोधित किया जा सकता है । साथ IDLV के संबंध में मध्यस्थता CRISPR/Cas9 वितरण, कम उत्पादन titers और episome-integrase-कुशल lentiviral प्रणालियों के सापेक्ष वहन जीनोम के कम अभिव्यक्ति के जीनोम-संपादन देने के लिए बोना के रूप में अपने उपयोगिता को सीमित उपकरण ट्रांसजेनिक construction. हम हाल ही में प्रदर्शित किया है कि दोनों transgene अभिव्यक्ति और वायरल IDLV उत्पादन के साथ जुड़े titers वायरल अभिव्यक्ति की कैसेट28के भीतर प्रतिलेखन कारक Sp1 के लिए बाध्यकारी साइटों के शामिल किए जाने से काफी बढ़ा रहे हैं । संशोधित IDLVs मजबूती से समर्थित CRISPR-मध्यस्थता जीन संपादन दोनों इन विट्रो में (HEK-293T कोशिकाओं में) और vivo में (पोस्ट-mitotic मस्तिष्क न्यूरॉन्स में), जबकि संगत ICLV की तुलना में कम लक्ष्य उत्परिवर्तन उत्प्रेरण-मध्यस्थता सिस्टम28। कुल मिलाकर, हम एक उपंयास, कॉंपैक्ट, सभी में एक CRISPR toolkit एक IDLV मंच पर किया और बढ़ाया जीन संपादन के लिए इस तरह के एक प्रसव के वाहन का उपयोग करने के विभिंन लाभों को रेखांकित विकसित की है ।
यहां, IDLV-CRISPR/Cas9 प्रणाली के उत्पादन प्रोटोकॉल विभिंन विधानसभा में शामिल कदम, शुद्धि, एकाग्रता, और IDLVs के अनुमापन, साथ ही साथ रणनीतियों इन वैक्टर के जीन संपादन प्रभावकारिता को मांय करने सहित वर्णित है । इस प्रोटोकॉल को आसानी से विभिंन जांचकर्ताओं की जरूरतों को पूरा करने के लिए स्केलेबल है और सफलतापूर्वक 1 x 1010 transducing इकाइयों (TU) की श्रेणी में titers के साथ LV वैक्टर उत्पंन करने के लिए डिज़ाइन किया गया है/mL. इस प्रोटोकॉल के माध्यम से उत्पंन वैक्टर कुशलतापूर्वक कई विभिंन प्रकार के सेल को संक्रमित करने के लिए उपयोग किया जा सकता है, मुश्किल से transduce भ्रूण स्टेम सेल, टेम कोशिकाओं (टी कोशिकाओं और मैक्रोफेज), और संस्कृति और vivo मेंशामिल- इंजेक्ट न्यूरॉन्स । इसके अलावा, प्रोटोकॉल समान रूप से अच्छी तरह से integrase के उत्पादन के लिए अनुकूल है-सक्षम lentiviral वैक्टर इसी तरह की मात्रा में ।
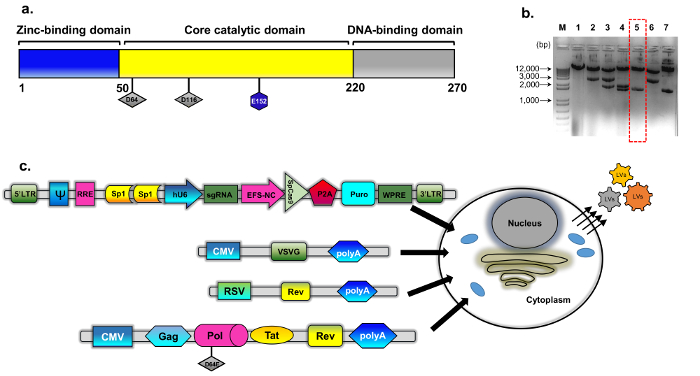
चित्र 1: IDLV पैकेजिंग (क) जंगली प्रकार integrase प्रोटीन की योजनाबद्ध (ख) संशोधित प्लाज्मिड psPAX2 से व्युत्पंन (तरीकों देखें, विवरण के लिए प्लाज्मिड निर्माण) था । प्रतिनिधि agarose जेल रूपांतरित integrase क्लोन के लिए स्क्रीन क्लोन की छवि । डीएनए नमूने एक मानक प्लाज्मिड डीएनए अलगाव मिनी किट का उपयोग कर तैयार EcoRV और SphI के साथ पाचन द्वारा विश्लेषण किया गया । सही ढंग से पचा क्लोन (संख्या 5, लाल बॉक्स धराशायी) आगे INTमें D64E प्रतिस्थापन के लिए प्रत्यक्ष (सैंज) अनुक्रमण द्वारा सत्यापित किया गया था । integrase की कमी वाली पैकेजिंग कैसेट का नाम pBK43 था. (ग) IDLV-CRISPR/Cas9 वैक्टर उत्पन्न करने के लिए कार्यरत क्षणिक अभिकर्मक प्रोटोकॉल की योजनाबद्ध, 293T के साथ transfected कोशिकाओं को दिखा-जी, पैकेजिंग, और VSV कैसेट्स (Sp1-transgene/CRISPR ऑल-इन-वन Cas9). वायरल कणों कि कोशिका झिल्ली से बाहर कली सदिश की पूरी लंबाई आरएनए होते है (transgene कैसेट से व्यक्त) । IDLV-पैकेजिंग प्रणाली की दूसरी पीढ़ी है, जो विनियामक प्रोटीन जैसे और rev. rev अभिव्यक्ति आगे एक अलग कैसेट (RSV-rev-प्लाज्मिड) से पूरक है शामिल किया गया था । Abbrev: लीटर लंबी टर्मिनल दोहराने, VSV-जी, vesicular stomatitis वायरस जी प्रोटीन, pCMV-cytomegalovirus प्रवर्तक; रोस सार्कोमा वायरस (RSV) प्रवर्तक; RRE-(Rev प्रतिक्रिया तत्व) । अभिव्यक्ति कैसेट पर अंय विनियामक तत्वों Sp1-बाध्यकारी साइटों, Rev प्रतिक्रिया तत्व (RRE), Woodchuck हेपेटाइटिस वायरस Posttranscriptional विनियामक तत्व (WPRE), एक कोर-बढ़ाव कारक 1α प्रमोटर (EFS-नेकां), वेक्टर पैकेजिंग शामिल तत् ψ (psi), ह्यूमन Cytomegalovirus (hCMV) प्रवर्तक, और मानव U6 प्रवर्तक । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।
Protocol
Representative Results
Discussion
IDLVs के लिए पसंद के वाहन के रूप में उभरने शुरू कर दिया है vivo जीन-संपादन, विशेष रूप से आनुवंशिक रोगों के संदर्भ में, इन वैक्टर के साथ जुड़े mutagenesis के कम जोखिम के लिए मोटे तौर पर वितरण प्लेटफार्मों के घालमेल क?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
हम तंत्रिका जीव विज्ञान, ड्यूक यूनिवर्सिटी स्कूल ऑफ मेडिसिन के डिपार्टमेंट और डीन के पद बेसिक साइंस, ड्यूक यूनिवर्सिटी के लिए शुक्रिया अदा करना चाहेंगे । हम भी पांडुलिपि पर टिप्पणी के लिए ड्यूक वायरल वेक्टर कोर के सदस्यों को धंयवाद । प्लाज्मिड pLenti CRISPRv2 को फेंग जांग (ब्रॉड इंस्टिट्यूट) से गिफ्ट किया गया । plasmids psPAX2, VSV-जी, pMD2. जी और pRSV-Rev सहित LV-पैकेजिंग प्रणाली डिडिएर Trono (EPFL, स्विट्जरलैंड) से एक तरह का उपहार था । इस काम के लिए वित्तीय सहायता विश्वविद्यालय के दक्षिण कैरोलिना स्कूल ऑफ मेडिसिन, ग्रांट RDF18080-E202 (बी. के.) द्वारा प्रदान की गई थी ।
Materials
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 – BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
References
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products – Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).

